Recherche et analyse de polymorphismes SNP Alexis Dereeper






- Slides: 22

Recherche et analyse de polymorphismes SNP Alexis Dereeper Formation Bio-informatique IRD

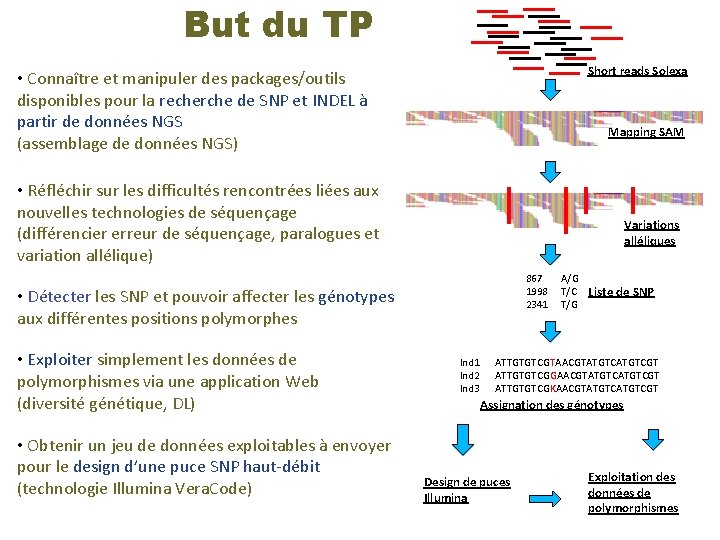
But du TP Short reads Solexa • Connaître et manipuler des packages/outils disponibles pour la recherche de SNP et INDEL à partir de données NGS (assemblage de données NGS) Mapping SAM • Réfléchir sur les difficultés rencontrées liées aux nouvelles technologies de séquençage (différencier erreur de séquençage, paralogues et variation allélique) Variations alléliques 867 1998 2341 • Détecter les SNP et pouvoir affecter les génotypes aux différentes positions polymorphes • Exploiter simplement les données de polymorphismes via une application Web (diversité génétique, DL) • Obtenir un jeu de données exploitables à envoyer pour le design d’une puce SNP haut-débit (technologie Illumina Vera. Code) Ind 1 Ind 2 Ind 3 A/G T/C Liste de SNP T/G ATTGTGTCGTAACGTATGTCGT ATTGTGTCGGAACGTATGTCGT ATTGTGTCGKAACGTATGTCGT Assignation des génotypes Design de puces Illumina Exploitation des données de polymorphismes

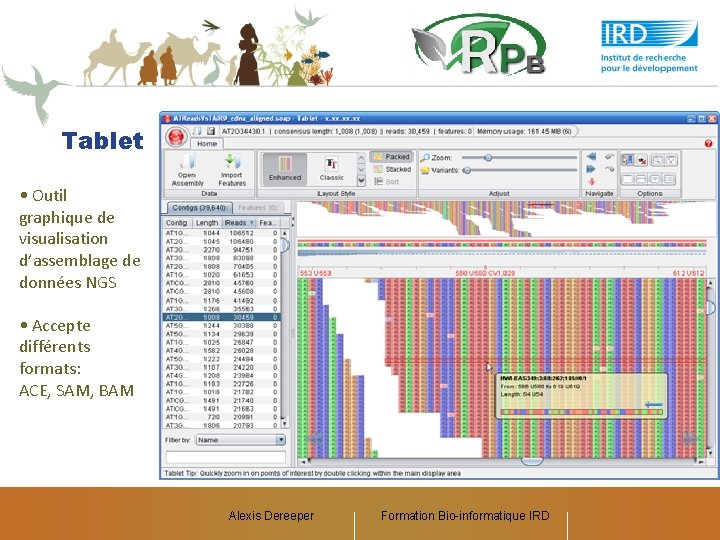
Tablet • Outil graphique de visualisation d’assemblage de données NGS • Accepte différents formats: ACE, SAM, BAM Alexis Dereeper Formation Bio-informatique IRD

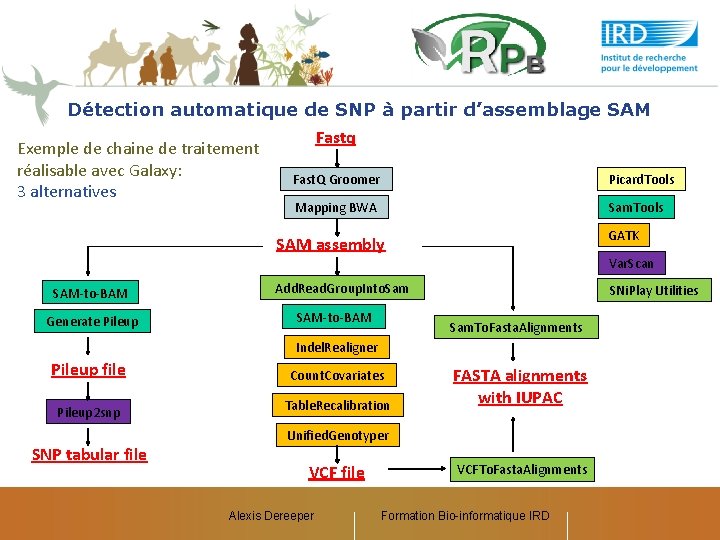
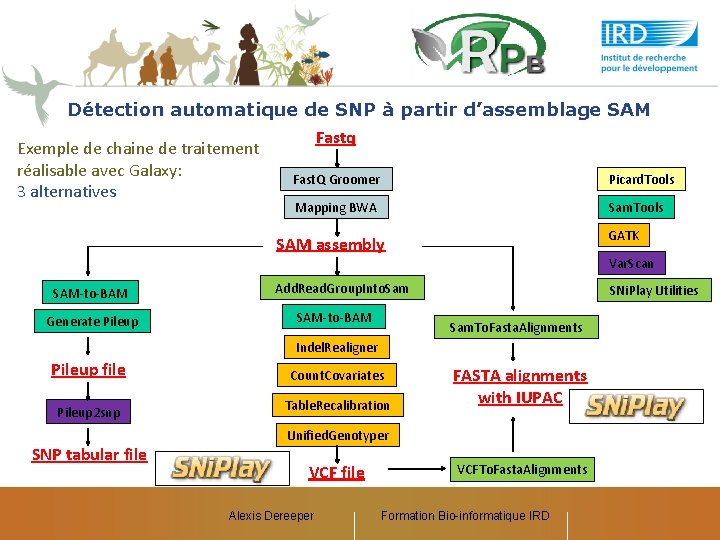
Détection automatique de SNP à partir d’assemblage SAM Exemple de chaine de traitement réalisable avec Galaxy: 3 alternatives Fastq Fast. Q Groomer Picard. Tools Mapping BWA Sam. Tools GATK SAM assembly Var. Scan SAM-to-BAM Generate Pileup Add. Read. Group. Into. Sam SAM-to-BAM SNi. Play Utilities Sam. To. Fasta. Alignments Indel. Realigner Pileup file Count. Covariates Pileup 2 snp Table. Recalibration SNP tabular file FASTA alignments with IUPAC Unified. Genotyper VCF file Alexis Dereeper VCFTo. Fasta. Alignments Formation Bio-informatique IRD

Varscan Logiciel de détection de SNP à partir de fichier Pileup : Pileup 2 snp Il existe aussi Pileup 2 indel mais non implémenté encore dans Galaxy South. Green Format Pileup Fichier texte décrivant pour chaque position: la base de la référence, la profondeur, les variations, la qualité seq 1 seq 1 272 273 274 275 276 277 278 279 T T T A G T G C 24 23 23 23 22 22 23 23 , . $. . . , , . , . . . , , , . . ^+. <<<+; <<<<<<=<; <; 7<& , . . . , , , . . A <<<; <<<<<3<=<<<; <<+ , . $. . , , . , . . . , , , . . . 7<7; <; <<<<<=<; <; <<6 , $. . , , . , . . . , , , . . . ^l. <+; 9*<<<<<=<<: ; <<<<. . . T, , . , . . . , , , . . . . 33; +<<7=7<<7<&<<1; <<6<. . , , . , . C. , , , . . G. +7<; <<<<<<<&<=<<: ; <<&<. . , , . , . . . , , , . . . . ^k. %38*<<; <7<<7<=<<<; <<<<< A. . T, , . , . . . , , , . . . ; 75&<<<<<=<<<9<<: << Alexis Dereeper Formation Bio-informatique IRD

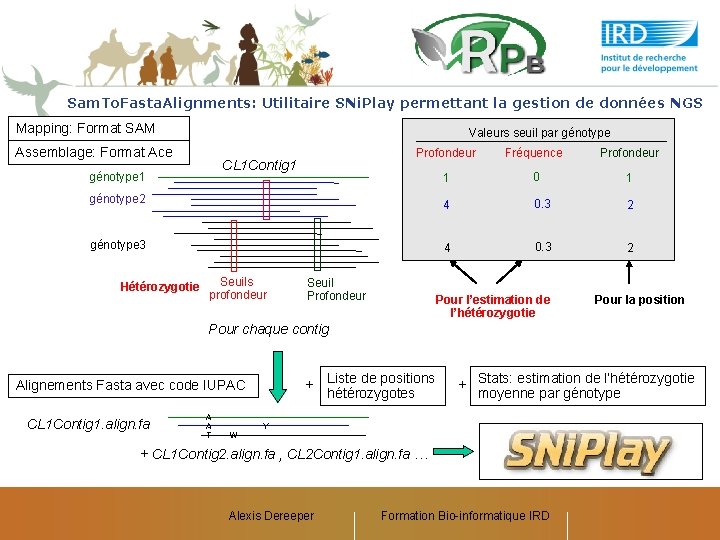
Sam. To. Fasta. Alignments: Utilitaire SNi. Play permettant la gestion de données NGS Mapping: Format SAM Valeurs seuil par génotype Assemblage: Format Ace Profondeur Fréquence Profondeur 1 0 1 génotype 2 4 0. 3 2 génotype 3 4 0. 3 2 CL 1 Contig 1 génotype 1 Hétérozygotie Seuils profondeur Seuil Profondeur Pour l’estimation de l’hétérozygotie Pour la position Pour chaque contig + Liste de positions Alignements Fasta avec code IUPAC CL 1 Contig 1. align. fa A A T hétérozygotes + Stats: estimation de l’hétérozygotie moyenne par génotype Y W + CL 1 Contig 2. align. fa , CL 2 Contig 1. align. fa … Alexis Dereeper Formation Bio-informatique IRD

GATK (Genome Analysis Tool. Kit) • Librairie logicielle pour l'analyse de données NGS. • Développé pour l'analyse des projets de reséquençage médical chez l'Humain (1000 Génomes, The Cancer Genome Atlas) PREPROCESS: * Index human genome (Picard), we used HG 18 from UCSC. * Convert Illumina reads to Fastq format * Convert Illumina 1. 6 read quality scores to standard Sanger scores FOR EACH SAMPLE: • Inclut des outils d'analyse de profondeur, recalibrateur de score de qualité, détection de SNP/In. Del • Complémentaire des 2 autres packages: Sam. Tools, Picard. Tools Alexis Dereeper 1. Align samples to genome (BWA), generates SAI files. 2. Convert SAI to SAM (BWA) 3. Convert SAM to BAM binary format (SAM Tools) 4. Sort BAM (SAM Tools) 5. Index BAM (SAM Tools) 6. Identify target regions for realignment (Genome Analysis Toolkit) 7. Realign BAM to get better Indel calling (Genome Analysis Toolkit) 8. Reindex the realigned BAM (SAM Tools) 9. Call Indels (Genome Analysis Toolkit) 10. Call SNPs (Genome Analysis Toolkit) 11. View aligned reads in BAM/BAI (Integrated Genome Viewer) Formation Bio-informatique IRD

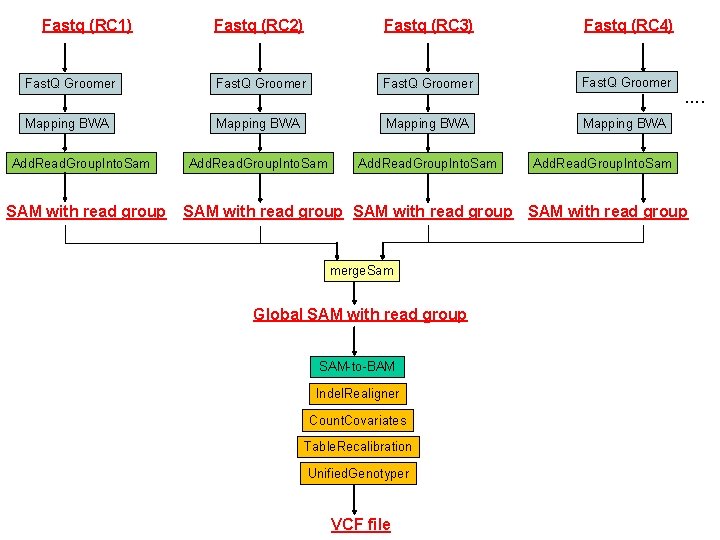
Fastq (RC 1) Fastq (RC 2) Fastq (RC 3) Fastq (RC 4) Fast. Q Groomer Mapping BWA Add. Read. Group. Into. Sam SAM with read group …. Add. Read. Group. Into. Sam SAM with read group merge. Sam Global SAM with read group SAM-to-BAM Indel. Realigner Count. Covariates Table. Recalibration Unified. Genotyper VCF file

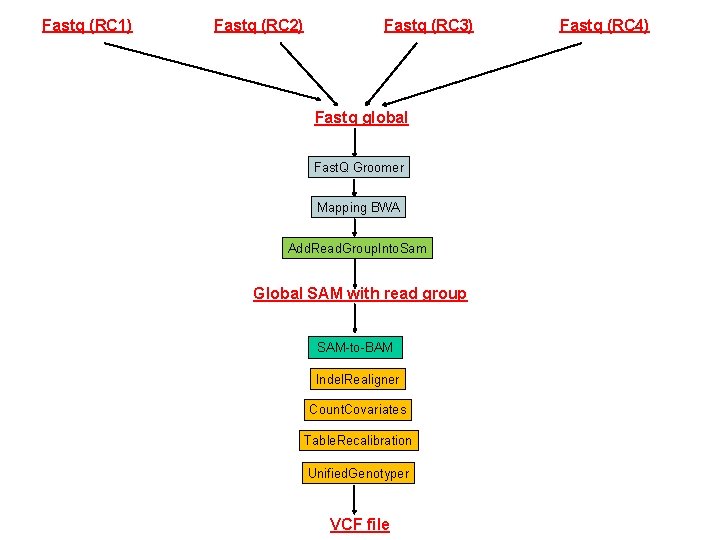
Fastq (RC 1) Fastq (RC 2) Fastq (RC 3) Fastq global Fast. Q Groomer Mapping BWA Add. Read. Group. Into. Sam Global SAM with read group SAM-to-BAM Indel. Realigner Count. Covariates Table. Recalibration Unified. Genotyper VCF file Fastq (RC 4)

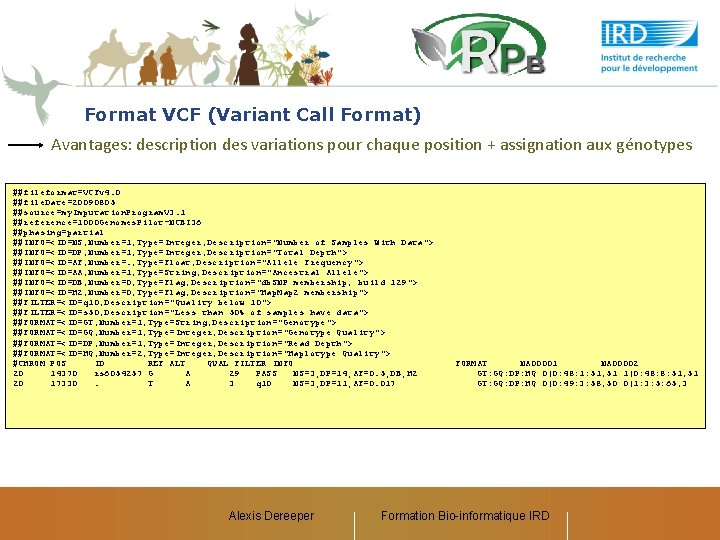
Format VCF (Variant Call Format) Avantages: description des variations pour chaque position + assignation aux génotypes ##fileformat=VCFv 4. 0 ##file. Date=20090805 ##source=my. Imputation. Program. V 3. 1 ##reference=1000 Genomes. Pilot-NCBI 36 ##phasing=partial ##INFO=<ID=NS, Number=1, Type=Integer, Description="Number of Samples With Data"> ##INFO=<ID=DP, Number=1, Type=Integer, Description="Total Depth"> ##INFO=<ID=AF, Number=. , Type=Float, Description="Allele Frequency"> ##INFO=<ID=AA, Number=1, Type=String, Description="Ancestral Allele"> ##INFO=<ID=DB, Number=0, Type=Flag, Description="db. SNP membership, build 129"> ##INFO=<ID=H 2, Number=0, Type=Flag, Description="Hap. Map 2 membership"> ##FILTER=<ID=q 10, Description="Quality below 10"> ##FILTER=<ID=s 50, Description="Less than 50% of samples have data"> ##FORMAT=<ID=GT, Number=1, Type=String, Description="Genotype"> ##FORMAT=<ID=GQ, Number=1, Type=Integer, Description="Genotype Quality"> ##FORMAT=<ID=DP, Number=1, Type=Integer, Description="Read Depth"> ##FORMAT=<ID=HQ, Number=2, Type=Integer, Description="Haplotype Quality"> #CHROM POS ID REF ALT QUAL FILTER INFO 20 14370 rs 6054257 G A 29 PASS NS=3; DP=14; AF=0. 5; DB; H 2 20 17330. T A 3 q 10 NS=3; DP=11; AF=0. 017 Alexis Dereeper FORMAT NA 00001 NA 00002 GT: GQ: DP: HQ 0|0: 48: 1: 51, 51 1|0: 48: 8: 51, 51 GT: GQ: DP: HQ 0|0: 49: 3: 58, 50 0|1: 3: 5: 65, 3 Formation Bio-informatique IRD

Autres fonctionalités GATK • Module Depth. Of. Coverage: Permet de renseigner de la profondeur de séquençage pour chaque gène, chaque position et chaque individu • Module Read. Backed. Phasing: Permet de définir dans la mesure du possible les associations d’allèles (phase ou haplotype) quand il y a hétérozygotie… Et non AGG GGA Alexis Dereeper Formation Bio-informatique IRD

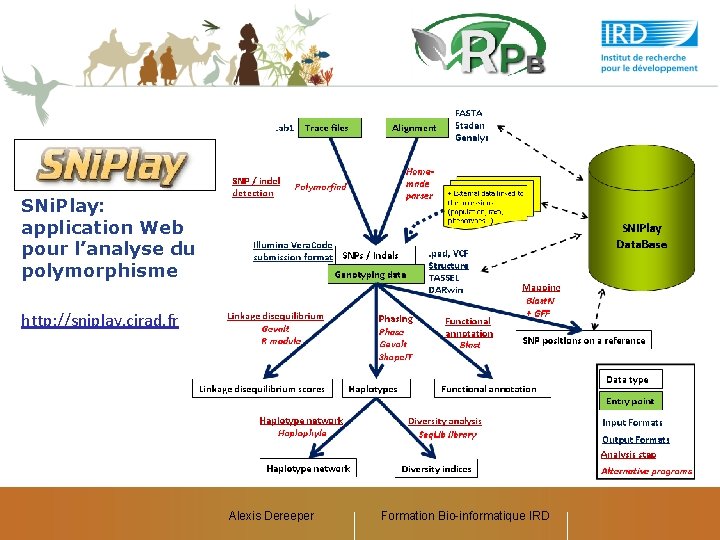
SNi. Play: application Web pour l’analyse du polymorphisme http: //sniplay. cirad. fr Alexis Dereeper Formation Bio-informatique IRD

Détection automatique de SNP à partir d’assemblage SAM Exemple de chaine de traitement réalisable avec Galaxy: 3 alternatives Fastq Fast. Q Groomer Picard. Tools Mapping BWA Sam. Tools GATK SAM assembly Var. Scan SAM-to-BAM Generate Pileup Add. Read. Group. Into. Sam SAM-to-BAM SNi. Play Utilities Sam. To. Fasta. Alignments Indel. Realigner Pileup file Count. Covariates Pileup 2 snp Table. Recalibration SNP tabular file FASTA alignments with IUPAC Unified. Genotyper VCF file Alexis Dereeper VCFTo. Fasta. Alignments Formation Bio-informatique IRD

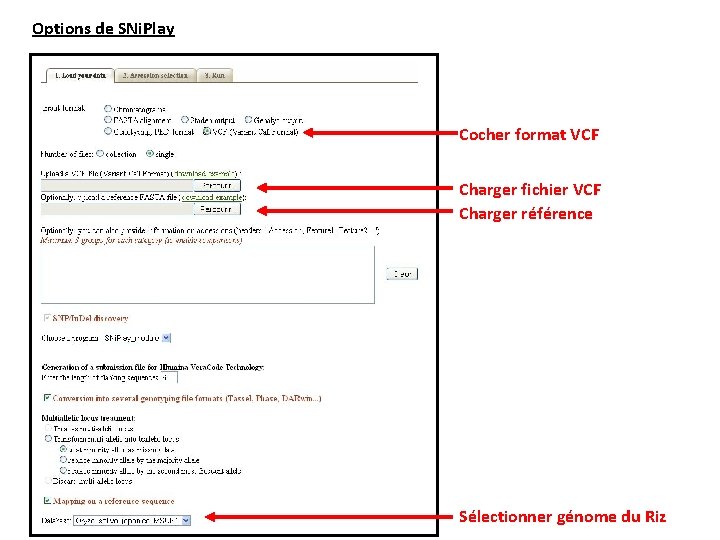
Options de SNi. Play Cocher format VCF Charger fichier VCF Charger référence Sélectionner génome du Riz

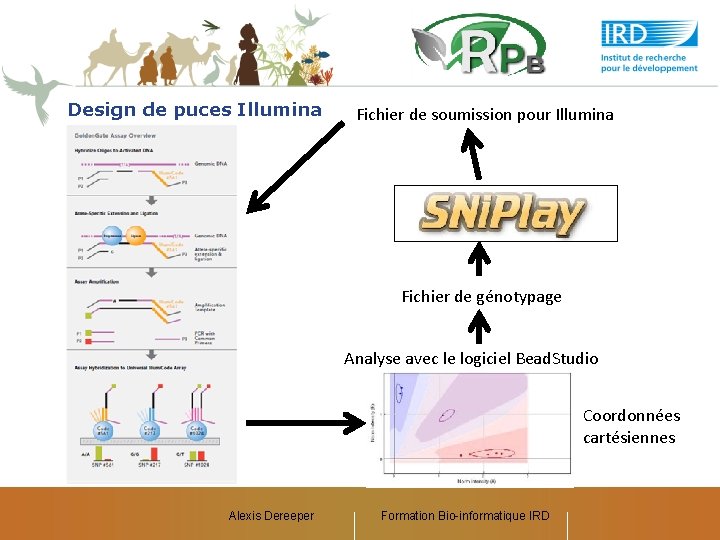
Design de puces Illumina Fichier de soumission pour Illumina Fichier de génotypage Analyse avec le logiciel Bead. Studio Coordonnées cartésiennes Alexis Dereeper Formation Bio-informatique IRD

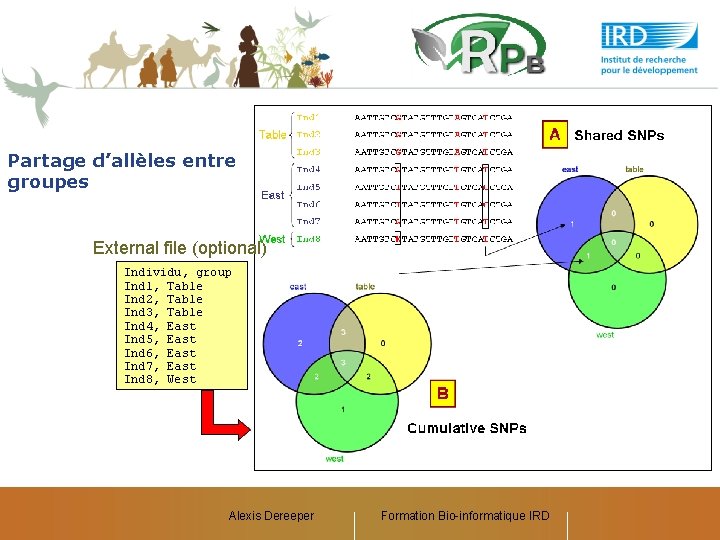
Partage d’allèles entre groupes External file (optional) Individu, group Ind 1, Table Ind 2, Table Ind 3, Table Ind 4, East Ind 5, East Ind 6, East Ind 7, East Ind 8, West Alexis Dereeper Formation Bio-informatique IRD

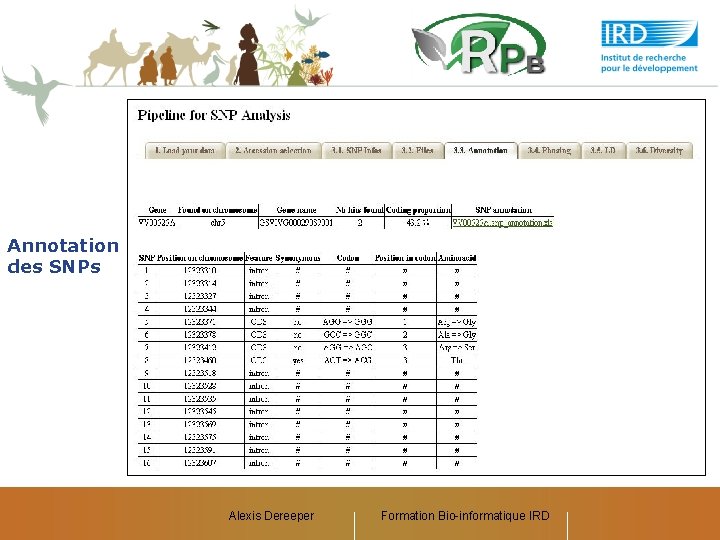
Annotation des SNPs Alexis Dereeper Formation Bio-informatique IRD

Annotation des SNPs Alexis Dereeper Formation Bio-informatique IRD

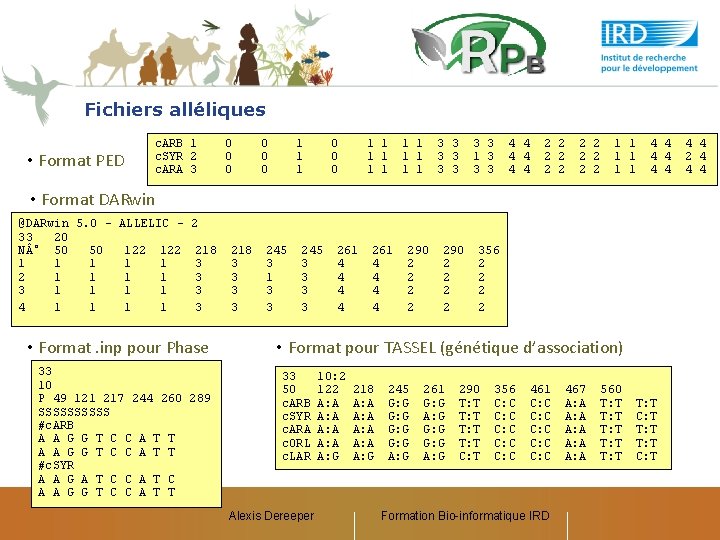
Fichiers alléliques • Format PED c. ARB 1 c. SYR 2 c. ARA 3 0 0 0 1 1 1 1 1 1 3 3 3 3 1 3 3 3 4 4 4 2 2 2 1 1 1 4 4 4 • Format DARwin @DARwin 5. 0 - ALLELIC 33 20 N ° 50 50 122 1 1 1 2 1 1 3 1 1 4 1 1 2 218 3 3 • Format. inp pour Phase 33 10 P 49 121 217 244 260 289 SSSSS #c. ARB A A G G T C C A T T #c. SYR A A G A T C C A T C A A G G T C C A T T 218 3 3 245 3 1 3 3 245 3 3 261 4 4 4 4 290 2 2 2 2 356 2 2 • Format pour TASSEL (génétique d’association) 33 50 c. ARB c. SYR c. ARA c. ORL c. LAR Alexis Dereeper 10: 2 122 A: A A: G 218 A: A A: G 245 G: G A: G 261 G: G A: G 290 T: T C: T 356 C: C C: C 461 C: C C: C Formation Bio-informatique IRD 467 A: A A: A 560 T: T T: T C: T 4 4 2 4 4 4

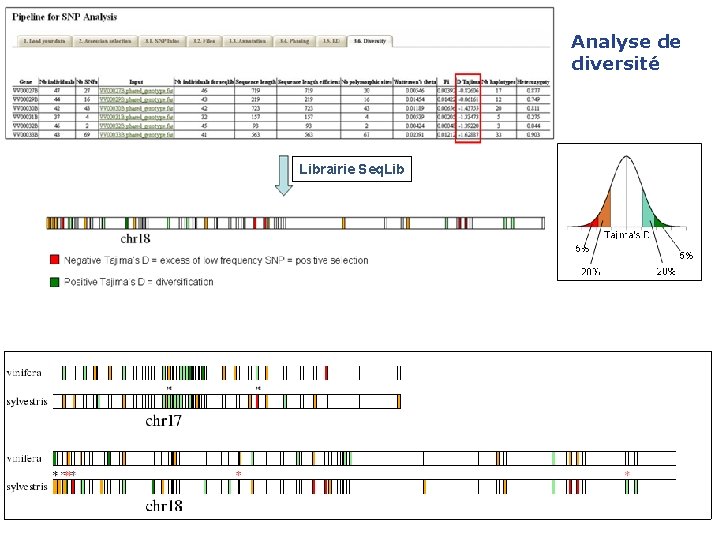
Analyse de diversité Librairie Seq. Lib

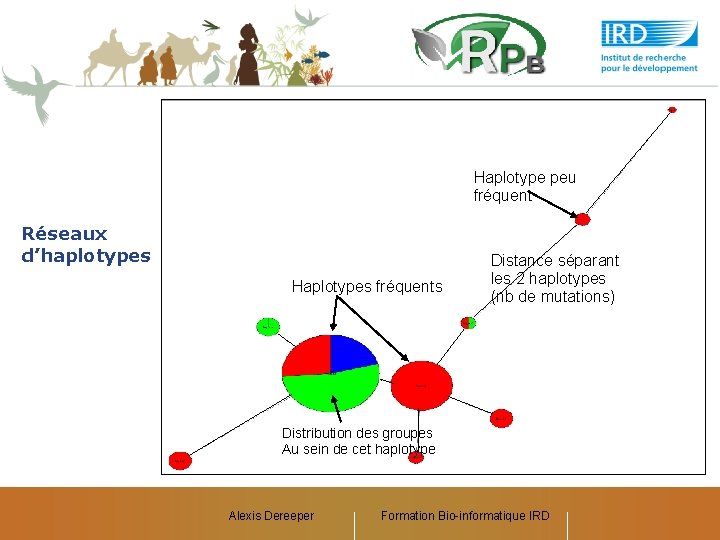
Haplotype peu fréquent Réseaux d’haplotypes Haplotypes fréquents Distance séparant les 2 haplotypes (nb de mutations) Distribution des groupes Au sein de cet haplotype Alexis Dereeper Formation Bio-informatique IRD

Alexis Dereeper Formation Bio-informatique IRD