Recherche dhomologie et Phylognie molculaire Alexis Dereeper Formation













- Slides: 15

Recherche d'homologie et Phylogénie moléculaire Alexis Dereeper Formation Bio-informatique IRD

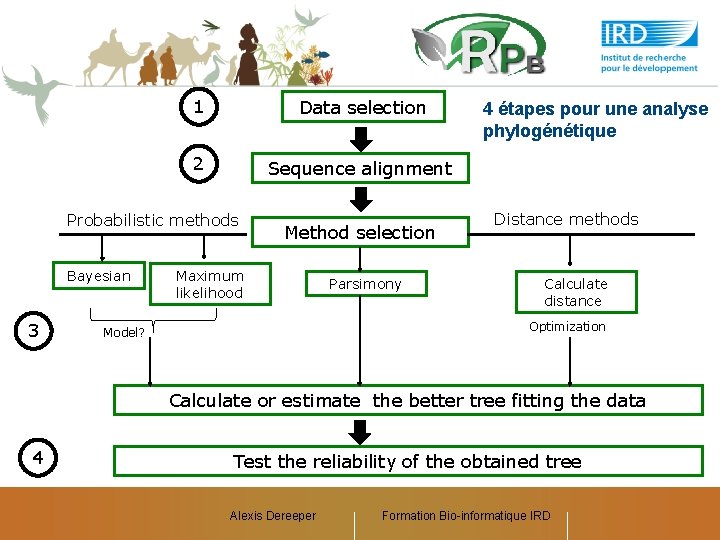
1 Data selection 2 Sequence alignment Probabilistic methods Bayesian 3 Method selection Maximum likelihood Parsimony 4 étapes pour une analyse phylogénétique Distance methods Calculate distance Optimization Model? Calculate or estimate the better tree fitting the data 4 Test the reliability of the obtained tree Alexis Dereeper Formation Bio-informatique IRD

Phylogeny. fr “The Phylogeny. fr platform transparently chains programs to automatically perform phylogenetic analysis tasks” Alexis Dereeper Formation Bio-informatique IRD

Recherche d’homologie Qu’est-ce que l’homologie de séquence? • Pas une notion quantitative (à différencier de similarité ou d’identité : 28%identité): on est homologue ou on ne l’est pas • Homologues: gènes provenant d’un ancêtre commun • Paralogues: gènes homologues issus d’un phénomène de duplication • Orthologues: gènes homologues issues d’une spéciation • Homologie et fonction: homologie ne signifie pas forcément même fonction. Orthologues rapprochés peuvent avoir la même fonction mais orthologues distants ont rarement le même rôle phénotypique (par contre même rôle dans une voie donnée) Paralogues acquièrent rapidement des fonctions différentes Alexis Dereeper Formation Bio-informatique IRD

Recherche d’homologie Comment les séquences homologues se ressemblent-elles? • De 100% à quelques aa/nt en commun • Pas vraiment de règle, de limite. On se base sur la probabilité que 2 séquences soit similaires par le fruit du hasard (e-value): ü ADN: e-value < 10 -6 et identité > 70% ü Protéine: e-value < 10 -3 et identité > 25% • Des séquences sans ressemblance apparente peuvent être homologues (ressemblance retrouvée au niveau 3 D). • Par contre, étant donné la dimension des séquences possibles, une ressemblance importante est généralement interprétée comme une homologie, et non comme une évolution convergente Alexis Dereeper Formation Bio-informatique IRD

Recherche d’homologie Comment détecter une homologie? Par comparaison de séquences = alignement de séquences 1 - Alignement local (ex: Blast) Conçu pour recher des régions similaires Alignement d’une séquence contre une banque de séquence (Swith &Waterman) 2 - Alignement global (ex: Clustal. W) Conçu pour comparer des séquences homolgues sur toute leur longueur (Needleman & Wunsh) Alexis Dereeper Formation Bio-informatique IRD

Recherche d’homologie Sortie de Blast classique Evalue= indicatif de la fiabilité du score Différents programmes Blast: ● ● ● Blast. N (Query: DNA / Subject : DNA) Blast. P (Query: protein/ Subject : protein) Blast. X (Query: DNA / Subject : protein) TBlast. N (Query: protein/ Subject : DNA) TBlast. X (Query: DNA traduit / Subject : DNA traduit) Alexis Dereeper Formation Bio-informatique IRD

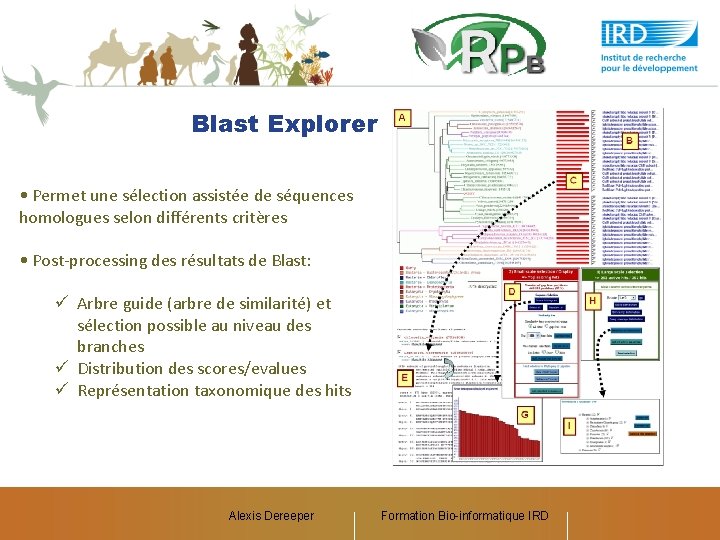
Blast Explorer • Permet une sélection assistée de séquences homologues selon différents critères • Post-processing des résultats de Blast: ü Arbre guide (arbre de similarité) et sélection possible au niveau des branches ü Distribution des scores/evalues ü Représentation taxonomique des hits Alexis Dereeper Formation Bio-informatique IRD

Recherche d’homologie Méthode BBMH (Best Blast Mutual Hits) ou RBH (Reciprocal Best Hit) Protéome Espèce 1 Protéome Espèce 2 Banques de séquences orthologues: ● ● ● Inparanoid (eucaryotes) Homolo. Gene (eucaryotes) Ortho. MCL DB COG (Clusters of Ortholog Groups of proteins) (procaryotes et eucaryotes) Green. Phyl (plantes) Alexis Dereeper Formation Bio-informatique IRD

Analyse phylogénétique Etape 1 : Alignement multiple (alignement global) • Logiciels d’alignement: ü Clustal. W rapides ü Muscle ü Tcoffee lents ü 3 DCoffee (optimise l’alignement avec structure 3 D) ü Mafft • Formats d’alignement : Fasta, Clustal, Phylip, Nexus • Logiciels de visualisation/édition d’alignement ü Sea. View ü Jalview ü Bio. Edit Alexis Dereeper Formation Bio-informatique IRD

Analyse phylogénétique Etape 2 : Nettoyage de l’alignement • Elimination des régions divergentes et présentant peu de signal phylogénétiques (peu informatives) Ces régions peuvent ne pas être homologues ou avoir été saturées par des substitutions (ex: sites synonymes en régions codantes) => Alignement nettoyé plus adéquat pour une analyse phylogénétique • Logiciels de curation d’alignement ü GBlocks Alexis Dereeper Formation Bio-informatique IRD

Analyse phylogénétique Etape 3 : Reconstruction phylogénétique Etape 3 a: Choix d’une méthode de reconstruction • 4 grandes familles de méthodes/algorithmes: ü Méthode des distances 2 à 2 (UPGMA, Neighbor Joining) o Fast. Dist, BIONJ, Neighbor ü Méthode du maximum de parcimonie o DNAPars, TNT ü Méthode du maximum de vraisemblance (max likelihood) o Phy. ML, PAML ü Inférence Bayesienne o Mr. Bayes, Beast Choisir le bon compromis vitesse/fiabilité • Format de sortie : matrice de distance, format Newick Alexis Dereeper Formation Bio-informatique IRD

Analyse phylogénétique Etape 3 : Reconstruction phylogénétique Etape 3 b: Choix des paramètres et modèles d’évolution • Différents modèles d’évolution indiquant le taux de substitution d’aa ou nt: ü DNA o Juke Cantor, Kimura, F 81, HKY 85, GTR ü protein o JTT, WAG, Dayhoff • Logiciels de test d’évolution: Test et sélection du modèle de substitution (et paramètres) le mieux adapté au jeu de donnée ü Prot. Test, Model. Test (basé sur Phy. ML) Alexis Dereeper Formation Bio-informatique IRD

Analyse phylogénétique Etape 3 : Reconstruction phylogénétique Etape 3 c: Estimation de la robustesse des branches • Procédure Bootstrap 1 - On rééchantillonne les séquences sur les colonnes: création d’un pseudo-alignement en tirant au hasard un certain nombre de sites puis recalcul de l’arbre. 2 - On réitère le processus N fois. 3 - Pour chaque branche de l’arbre initial, on regarde combien de fois on l’observe dans les arbres de bootstrap. Plus le nombre est elevé, plus la branche est fiable • Test a. LRT (approximate Likelihood Ratio Test) (Anisimova & Gascuel, Syst Biol, 2006) ü Intégré dans Phy. ML ü Beaucoup plus rapide (Phy. ML lancé qu’une seule fois) Alexis Dereeper Formation Bio-informatique IRD

Analyse phylogénétique Etape 4 : Visualisation et édition de l’arbre phylogénétique • Outils graphiques permettant l’affichage d’arbres à partir de format Newick: ü Tree. Dyn ü Draw. Gram, Draw. Tree ü ATV ü NJPlot • Formats de sortie: PNG, SVG, PDF… Etape 5 : Interprétation de l’arbre Alexis Dereeper Formation Bio-informatique IRD
 Formation initiale vs formation continue
Formation initiale vs formation continue Cross-docking définition
Cross-docking définition Algorithme de recherche tabou en python
Algorithme de recherche tabou en python Recherche documentaire
Recherche documentaire Frais de recherche et de développement syscohada
Frais de recherche et de développement syscohada Recherche d image
Recherche d image Recherche opérationnelle
Recherche opérationnelle Initiation à la recherche en soins infirmiers
Initiation à la recherche en soins infirmiers Recherche endometriose irm
Recherche endometriose irm Moteur de recherche exemple
Moteur de recherche exemple Cidreq recherche simple
Cidreq recherche simple V
V Google accs
Google accs Recherche séquentielle
Recherche séquentielle Recherche dichotomique
Recherche dichotomique Zb uzh recherche
Zb uzh recherche