Quest ce que la vie grand pa grand





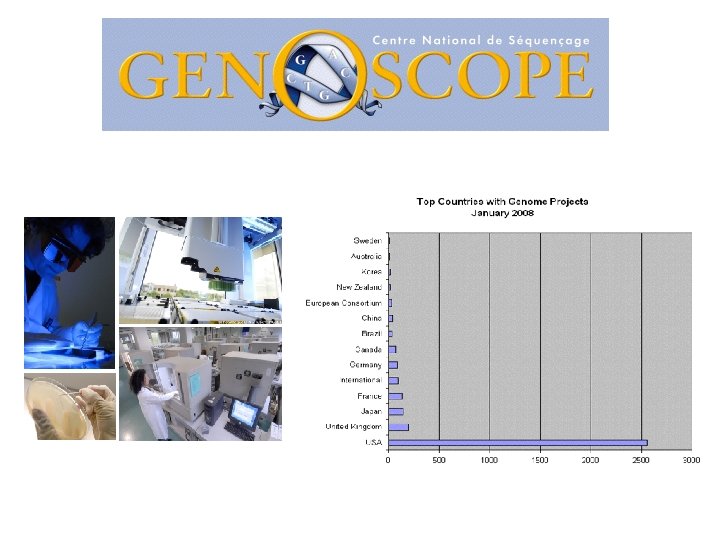







- Slides: 40

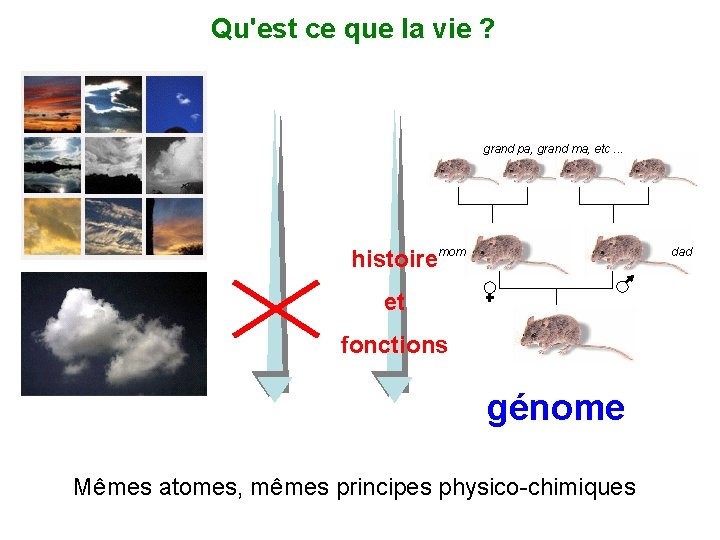
Qu'est ce que la vie ? grand pa, grand ma, etc. . . histoiremom dad et fonctions génome Mêmes atomes, mêmes principes physico-chimiques

Qu'est ce qu'un génome ? Génome: (1920 Hans Winkler, Hamburg) ensemble des déterminants héréditaires (gènes) propres à une espèce donnée. Génome: ensemble de l'information héréditaire d'un organisme. Cette information est présente en totalité dans chacune des cellules de l'organisme. Lorsqu'une cellule se divise l'information est copiée et transmise aux deux cellules filles. Génome: contient les instructions nécessaires au développement, au fonctionnement, au maintien de l'intégrité et à la reproduction des cellules et de l'organisme. 1944: l'ADN est le support de l'hérédité 1953: les propriétés de la molécule d'ADN expliquent la dualité de son rôle: - instructions fonctionnelles pour l'organisme: séquence des nucléotides - reproduction (formation du semblable): appariement des nucléotides

La dualité fonctionnelle de l'ADN est intrinsèque à sa structure OO CH 3 Base 5’ CH 2 4’ C 5 4 H C 3’ OH T 3 O H 6 N H C 1’ 5 1 NH 4 N sucre thymine C 2’ H C 3 2 O O NH 2 O N 6 N 5 9 N 2 sucre cytosine N NH 2 7 8 1 O O P 4 3 N 6 A 2 adenine 8 9 1 N N O 7 5 4 G 3 sucre N 6 1 NH 2 guanine H Double hélice désoxyribonucléotides Séquence des nucléotides L'information génétique (le message) est fournie par la succession des nucléotides le long des brins d'ADN Génomique Combinaisons possibles = Fourche de réplication 4 n n = 50 > nbre particules de l'univers La reproduction des organismes est assurée par l'appariement des nucléotides. Complémentarité des nucléotides Les deux molécules filles sont identiques à la molécule mère et donc identiques entre elles Deux double hélices filles

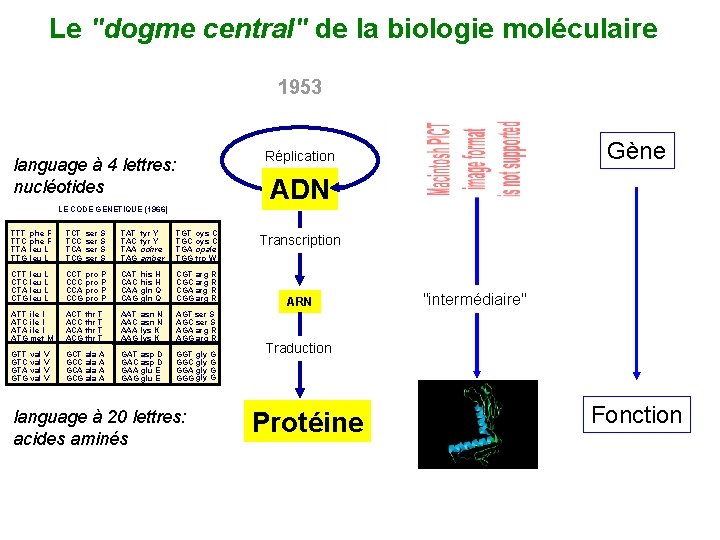
Le "dogme central" de la biologie moléculaire 1953 ADN LE CODE GENETIQUE (1966) TTT TTC TTA TTG phe F leu L TCT TCC TCA TCG CTT CTC CTA CTG leu L CCT pro P CCC pro P CCA pro P CCG pro P CAT his H CAC his H CAA gln Q CAG gln Q CGT arg R CGC arg R CGA arg R CGG arg R ATT ATC ATA ATG ile I met M ACT thr T ACC thr T ACA thr T ACG thr T AAT asn N AAC asn N AAA lys K AAG lys K AGT ser S AGC ser S AGA arg R AGG arg R GCT ala A GCC ala A GCA ala A GCG ala A GAT asp D GAC asp D GAA glu E GAG glu E GGT gly G GGC gly G GGA gly G GGG gly G GTT val V GTC val V GTA val V GTG val V ser S TAT TAC TAA TAG tyr Y ochre amber TGT TGC TGA TGG language à 20 lettres: acides aminés Gène Réplication language à 4 lettres: nucléotides cys C opale trp W Transcription ARN "intermédiaire" Traduction Protéine Fonction

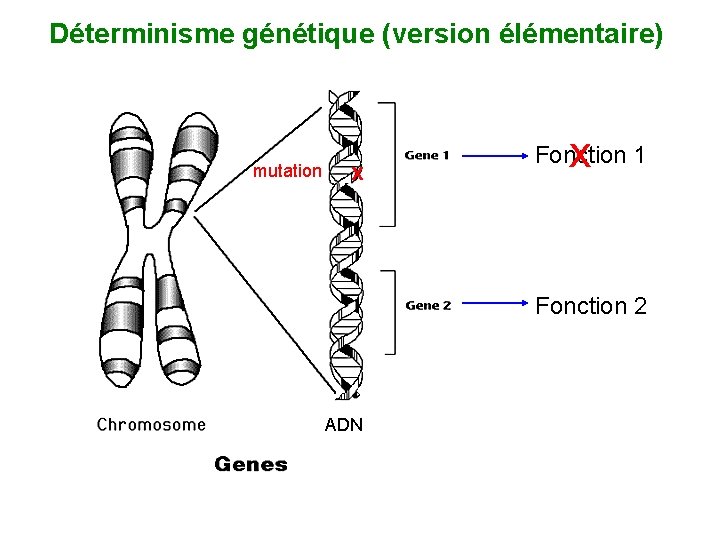
Déterminisme génétique (version élémentaire) mutation x x Fonction 1 Fonction 2 ADN

Taille des génomes et séquençage: bases de la génomique

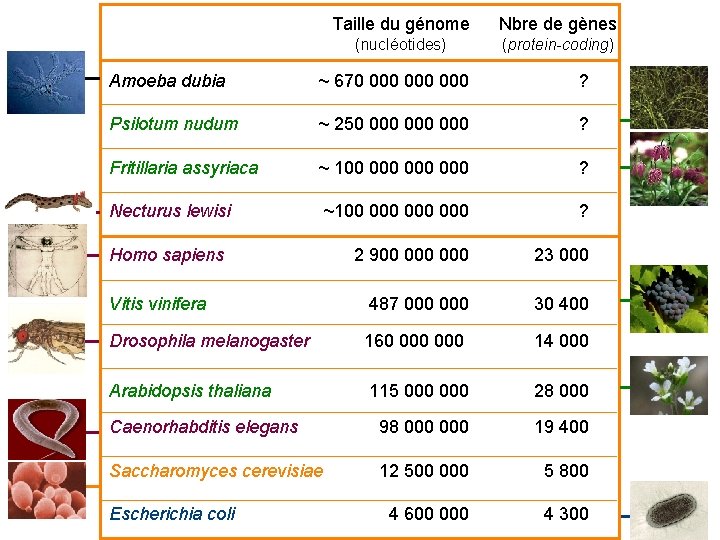
Taille du génome Nbre de gènes (nucléotides) (protein-coding) Amoeba dubia ~ 670 000 000 ? Psilotum nudum ~ 250 000 000 ? Fritillaria assyriaca ~ 100 000 000 ? Necturus lewisi ~100 000 000 ? 2 900 000 23 000 487 000 30 400 Homo sapiens Vitis vinifera Drosophila melanogaster Arabidopsis thaliana 160 000 14 000 115 000 28 000 Caenorhabditis elegans 98 000 19 400 Saccharomyces cerevisiae 12 500 000 5 800 4 600 000 4 300 Escherichia coli

Les génomes sont (trop) grands Echelle de taille des molécules d’ADN et des génomes distance = 1 paire de bases mammifères animaux champignons levures 10 102 103 104 kilobases = kb Le paradoxe de la valeur C plantes bactéries archaea virus 1 homme 105 106 amibes 107 mégabases = Mb 108 109 1010 1011 1012 paires de bases gigabases = Gb C = complexité du génome = nombre total de nucléotides du génome haploïde (taille du génome) å en général, les génomes sont trop grands pour le nombre de protéines qu'ils codent å la complexité des génomes n'est pas en relation directe avec la complexité des organismes et le nombre de gènes å les génomes d’espèces proches peuvent différer considérablement en taille

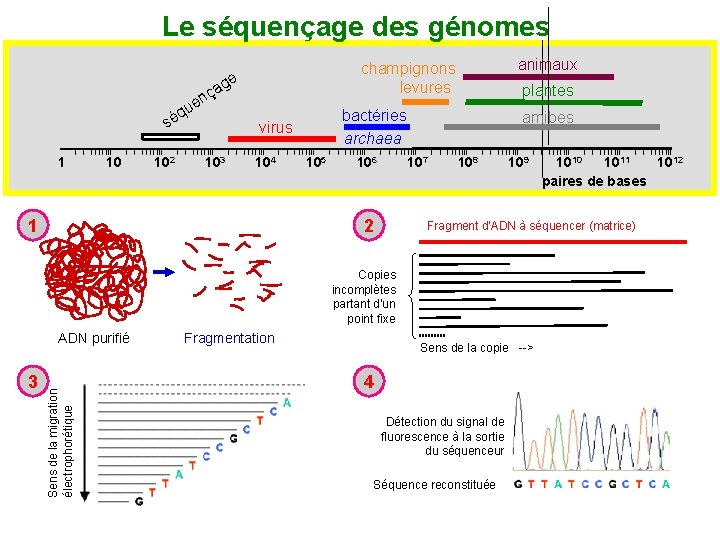
Le séquençage des génomes ge q sé 1 10 102 a nç ue 104 1 plantes bactéries archaea virus 103 animaux champignons levures 105 106 amibes 107 2 108 109 Fragment d'ADN à séquencer (matrice) Copies incomplètes partant d'un point fixe 3 Sens de la migration électrophorétique ADN purifié Fragmentation 1010 1011 1012 paires de bases Sens de la copie --> 4 Détection du signal de fluorescence à la sortie du séquenceur Séquence reconstituée

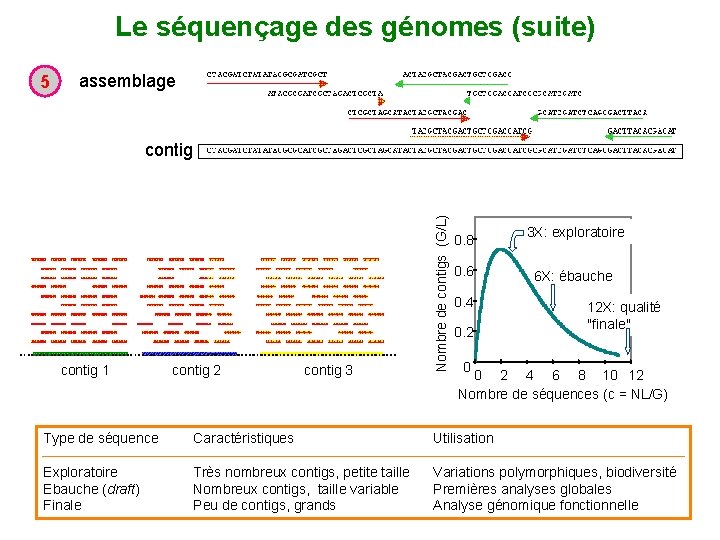
Le séquençage des génomes (suite) 5 assemblage contig 1 contig 2 contig 3 Nombre de contigs (G/L) contig 0. 8 0. 6 0. 4 0. 2 3 X: exploratoire 6 X: ébauche 12 X: qualité "finale" 0 0 2 4 6 8 10 12 Nombre de séquences (c = NL/G) Type de séquence Caractéristiques Utilisation Exploratoire Ebauche (draft) Finale Très nombreux contigs, petite taille Nombreux contigs, taille variable Peu de contigs, grands Variations polymorphiques, biodiversité Premières analyses globales Analyse génomique fonctionnelle

Le séquençage des génomes (fin) 6 Finition (supercontigs) Ossature de supercontigs (scaffolds) 7 Finition (remplissage des trous et zones de basse qualité vérification des assemblages, examen des séquences répétées, … ) Séquence finie, complète et de haute qualité 8 Annotation: ensemble de procédures informatiques qui: 1 - prédisent (± efficacement) les limites des gènes, des éléments de contrôle et de tout autre élément du génome 2 - suggèrent les fonctions des gènes à partir des comparaisons avec ce qui est déjà connu


Les premiers génomes séquencés 1995 Haemophilus influenzae Mycoplasma genitalium 1. 8 Mb 0. 6 Mb Bactéries 1996 Mycoplasma pneumoniae Synechocystis sp. 0. 8 Mb 3. 6 Mb Methanococcus jannaschii 1. 7 Mb (Première Archae) Saccharomyces cerevisiae 12. 3 Mb (Premier Eucaryote) 1998 Caenorhabditis elegans 98 Mb (Premier organisme multicellulaire) 2000 Arabidopsis thaliana 115 Mb (Première plante) Drosophila melanogaster 160 Mb (ébauche) Homo sapiens 2 900 MB Annonce internationale 1ère ébauche 90 % (150 000 trous) 2004 2008 Homo sapiens 2ème ébauche (99, 9 % de l' euchromatine) Deux individus Projet "1000 genomes" Consortium international 1000 individus à travers le monde

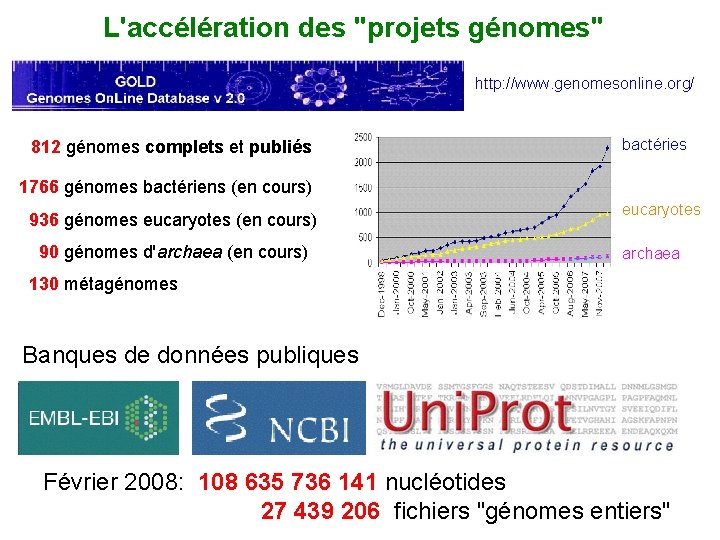
L'accélération des "projets génomes" http: //www. genomesonline. org/ 812 génomes complets et publiés bactéries 1766 génomes bactériens (en cours) 936 génomes eucaryotes (en cours) 90 génomes d'archaea (en cours) eucaryotes archaea 130 métagénomes Banques de données publiques Février 2008: 108 635 736 141 nucléotides 27 439 206 fichiers "génomes entiers"

Pourquoi séquencer les génomes ? Biotechnologies fermentations et bioconversions (acetate, acetone, butanol, éthanol, hydrogène …. ) additifs alimentaires (alginate, succinate, glutamate … ) production d'enzymes (cellulase, biocatalyse …. ) et protéines Environnement: cycles naturels (carbone, azote , conversion de la biomasse …) traitements (pesticides, fongicides, algicides, …) énergie, pétrole, détergents traitements des eaux, détoxification des sols Alimentation: produits laitiers, fromages, suppléments diététiques, fermentations alimentaires … Agronomie: animaux, plantes, et leurs pathogènes, résistance … Biologie et écologie marine: pêche, aquaculture, algues, plancton …. Pharmacie: vitamines, antibiotiques, acides aminés, acide lactique …. Santé humaine: pathogènes, cancer, vaccins, infections nosocomiales, insectes vecteurs … Connaissance: éducation, évolution, origine de la vie, arbre de la vie, compréhension des mécanismes fondamentaux de la vie, biodiversité …

La génomique comparative (Eucaryotes) Chlorophyta Ciliophora Ostreococcus tauri (2006) Paramecium tetraurelia (2006) Tetrahymena thermophila (2006) Mammalia Viridiplantae Conosa Homo sapiens Pan troglodytes Mus musculus Rattus norvegicus Gallus gallus Tetraodon negrovirids Fugu rubripes Apicomplexa Plasmodium falciparum (2002) Plasmodium yoeli (2002) Cryptosporidium hominis (2004) Cryptosporidium parvum (2004) Theileria annulata (2005) Theileria parva (2005) Toxoplasma gondi Arabidospis thaliana Oryza sativa Populus nigra Vitis vinifera Ascomycota Saccharomyces cerevisiae (1996) Schizosaccharomyces pombe (2002) Entamoeba histolytica (2005) Dictyostelium discoideum (2005) Nematoda Stramenopiles Thalassiosira pseudonana (2004) Euglenozoa Rhodophyta Leishmania major (2005) Trypanosoma brucei (2005) Trypanosoma cruzi (2005) Cyanidioschyzon merolae (2004) Galdieria sulphuraria (2005) Arthropoda Echinodermata Caenorhabditis elegans (1998) Caenorhabditis briggsae (2003) Strongylocentrus purpuratus Oscheius tipulae (2006) Drosophila melanogaster (2000) Meloidogyne incognita

Ernst Haeckel, 1866

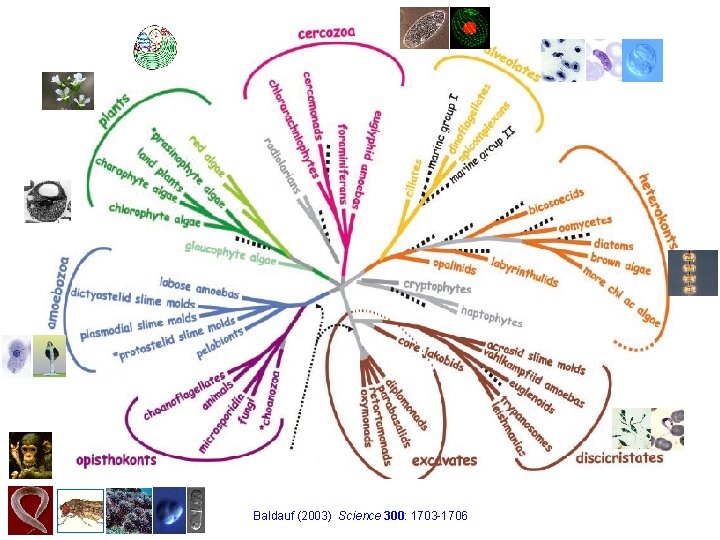
Baldauf (2003) Science 300: 1703 -1706

Qu'apprenons nous dans les génomes ? Catalogue complet des gènes et autres éléments Présence de nombreux gènes et autres éléments de fonctions inconnues Processus dynamiques de modification (altération et évolution) du génome

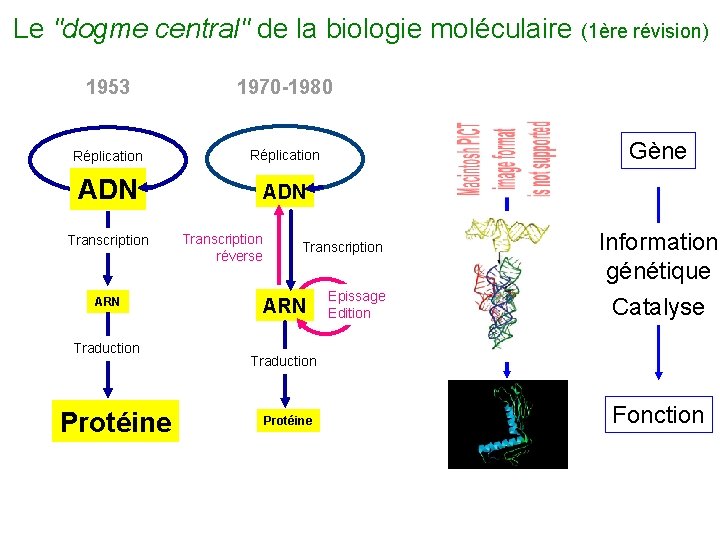
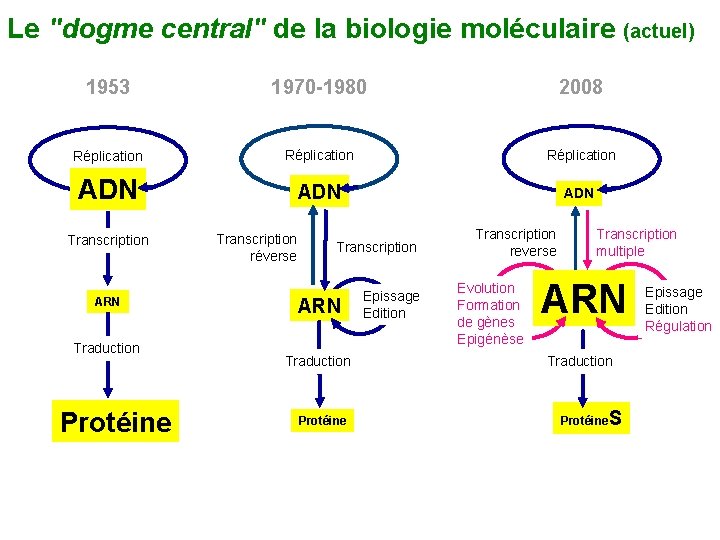
Le "dogme central" de la biologie moléculaire (1ère révision) 1953 1970 -1980 Réplication ADN Transcription ARN Traduction Protéine Transcription réverse Gène Transcription ARN Epissage Edition Information génétique Catalyse Traduction Protéine Fonction

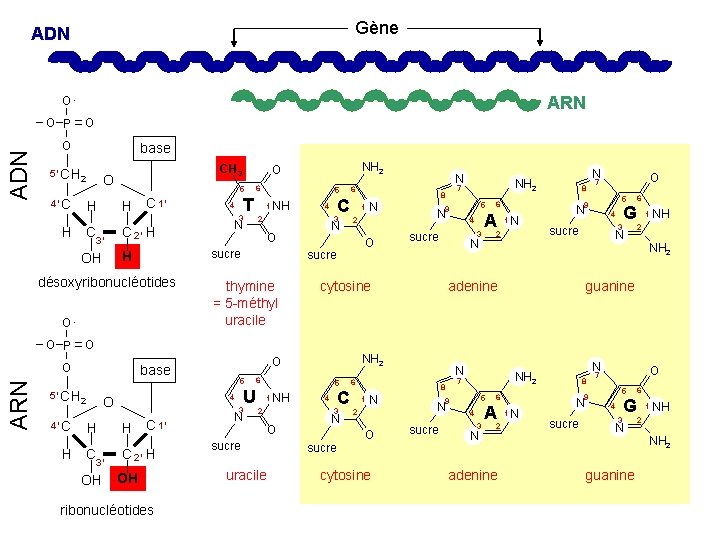
Gène ADN ARN O- O base 5’ CH 2 4’ C CH 3 O H H C 5 H 3’ C 1’ 3 C 2’ H désoxyribonucléotides O- C 4 2 N 3 N 6 thymine = 5 -méthyl uracile N 2 5 9 N 4 3 sucre 6 A 8 1 N O 7 5 9 2 N cytosine N NH 2 7 8 1 sucre N 4 3 sucre G N adenine 5’ CH 2 H H C 3’ OH 5 4 O 3 H C 1’ C 2’ H OH ribonucléotides NH 2 O base N 6 U 5 1 NH 2 C 4 3 O ARN 1 NH N 6 1 NH 2 guanine O O 4’ C T 5 sucre uracile N sucre N 6 N 2 cytosine 5 9 N sucre N NH 2 7 8 1 O O P 6 sucre H OH 4 NH 2 O O ADN O O O P 4 3 N 6 A 2 adenine 8 9 1 N N sucre O 7 5 4 3 G N guanine 6 1 NH 2

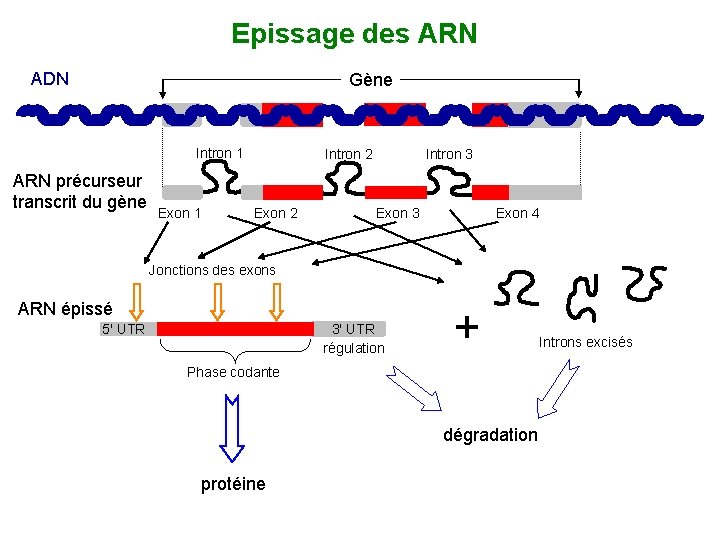
Epissage des ARN ADN Gène Intron 1 ARN précurseur transcrit du gène Exon 1 Intron 2 Exon 2 Intron 3 Exon 4 Jonctions des exons ARN épissé 5' UTR 3' UTR régulation + Phase codante dégradation protéine Introns excisés

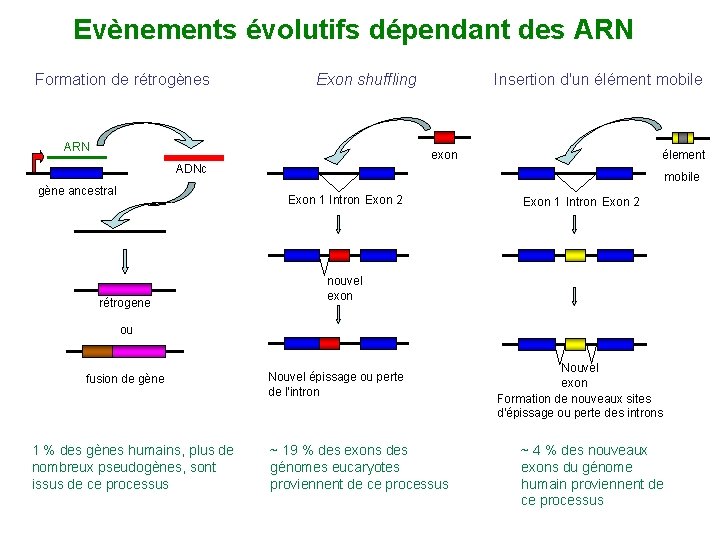
Evènements évolutifs dépendant des ARN Formation de rétrogènes Exon shuffling ARN Insertion d'un élément mobile exon élement ADNc gène ancestral mobile Exon 1 Intron Exon 2 rétrogene Exon 1 Intron Exon 2 nouvel exon ou fusion de gène 1 % des gènes humains, plus de nombreux pseudogènes, sont issus de ce processus Nouvel épissage ou perte de l'intron ~ 19 % des exons des génomes eucaryotes proviennent de ce processus Nouvel exon Formation de nouveaux sites d'épissage ou perte des introns ~ 4 % des nouveaux exons du génome humain proviennent de ce processus

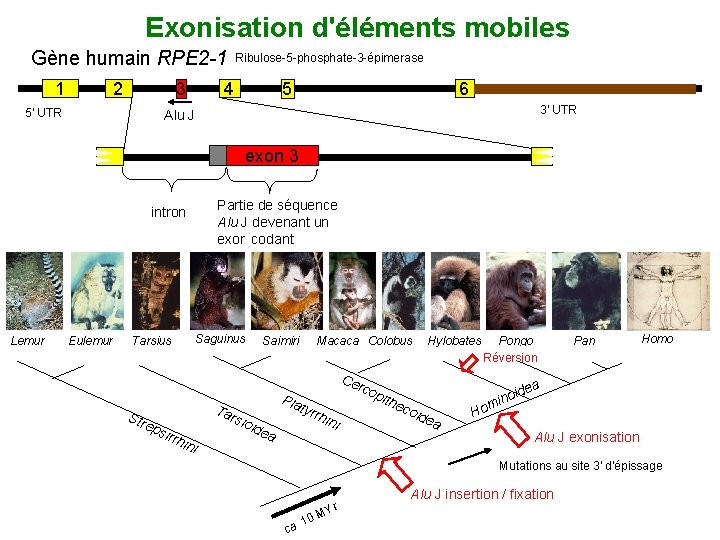
Exonisation d'éléments mobiles Gène humain RPE 2 -1 1 2 3 5' UTR Ribulose-5 -phosphate-3 -épimerase 4 5 6 3' UTR Alu J exon 3 intron Lemur Eulemur Partie de séquence Alu J devenant un exon codant Saguinus Tarsius Saimiri irrh ini Ce rc Pla ty Ta Str eps opi th rrh rsio ide Hylobates Macaca Colobus a ini Pongo Réversion Pan Homo eco ea ide a id ino m Ho Alu J exonisation Mutations au site 3' d'épissage 10 ca. M Yr Alu J insertion / fixation

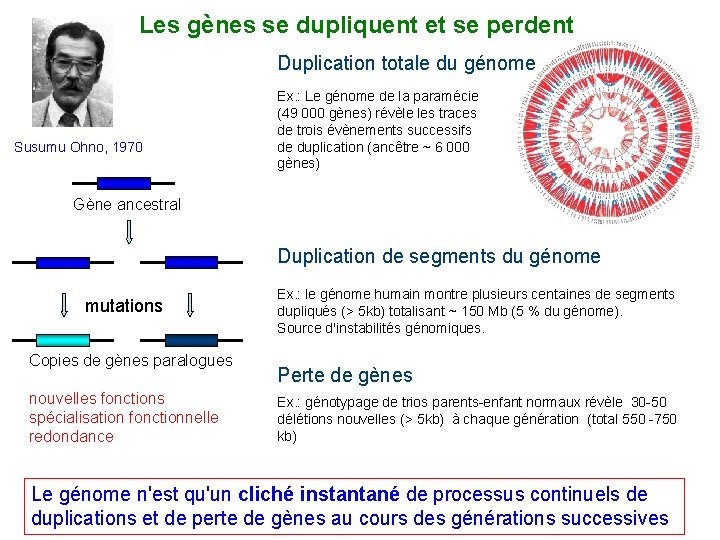
Les gènes se dupliquent et se perdent Duplication totale du génome Susumu Ohno, 1970 Ex. : Le génome de la paramécie (49 000 gènes) révèle les traces de trois évènements successifs de duplication (ancêtre ~ 6 000 gènes) Gène ancestral Duplication de segments du génome mutations Copies de gènes paralogues nouvelles fonctions spécialisation fonctionnelle redondance Ex. : le génome humain montre plusieurs centaines de segments dupliqués (> 5 kb) totalisant ~ 150 Mb (5 % du génome). Source d'instabilités génomiques. Perte de gènes Ex. : génotypage de trios parents-enfant normaux révèle 30 -50 délétions nouvelles (> 5 kb) à chaque génération (total 550 -750 kb) Le génome n'est qu'un cliché instantané de processus continuels de duplications et de perte de gènes au cours des générations successives

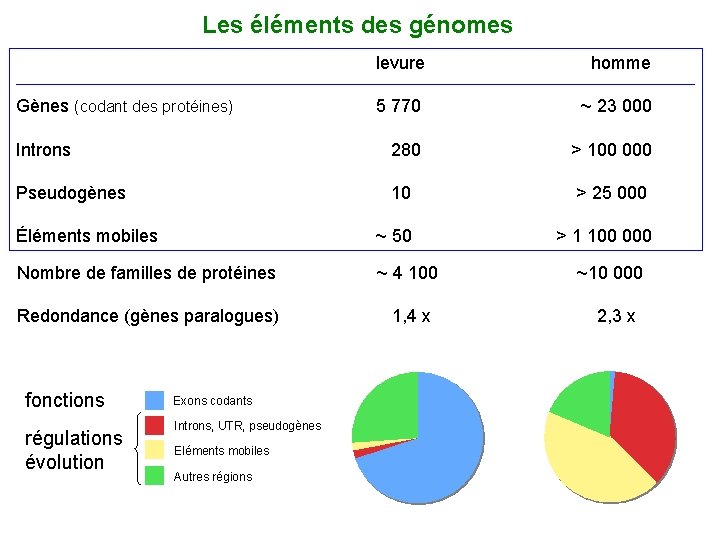
Les éléments des génomes levure homme 5 770 ~ 23 000 Introns 280 > 100 000 Pseudogènes 10 > 25 000 Éléments mobiles ~ 50 > 1 100 000 Nombre de familles de protéines ~ 4 100 Redondance (gènes paralogues) 1, 4 x Gènes (codant des protéines) fonctions régulations évolution Exons codants Introns, UTR, pseudogènes Eléments mobiles Autres régions ~10 000 2, 3 x

Buts et outils de la génomique fonctionnelle Buts: connaître les fonctions de tous les gènes comprendre leurs interactions prédire les phénotypes à partir du génotype

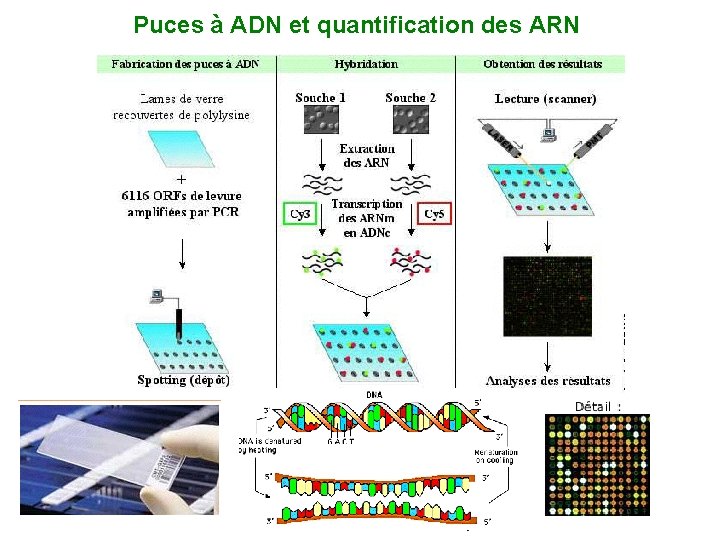
Puces à ADN et quantification des ARN

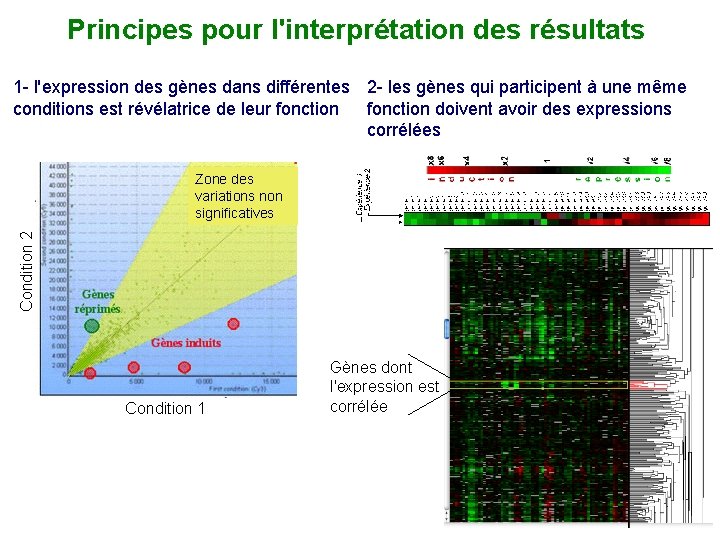
Principes pour l'interprétation des résultats 1 - l'expression des gènes dans différentes 2 - les gènes qui participent à une même conditions est révélatrice de leur fonction doivent avoir des expressions corrélées Condition 2 Zone des variations non significatives Condition 1 Gènes dont l'expression est corrélée

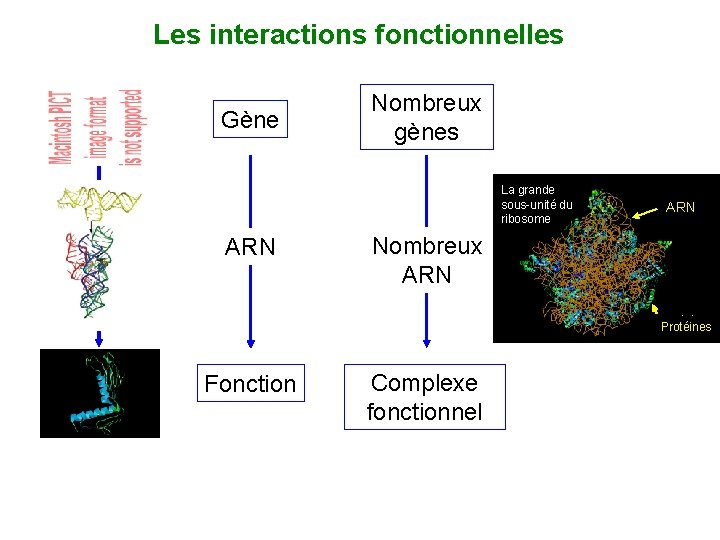
Les interactions fonctionnelles Gène Nombreux gènes La grande sous-unité du ribosome ARN RNA Nombreux ARN proteins Protéines Fonction Complexe fonctionnel

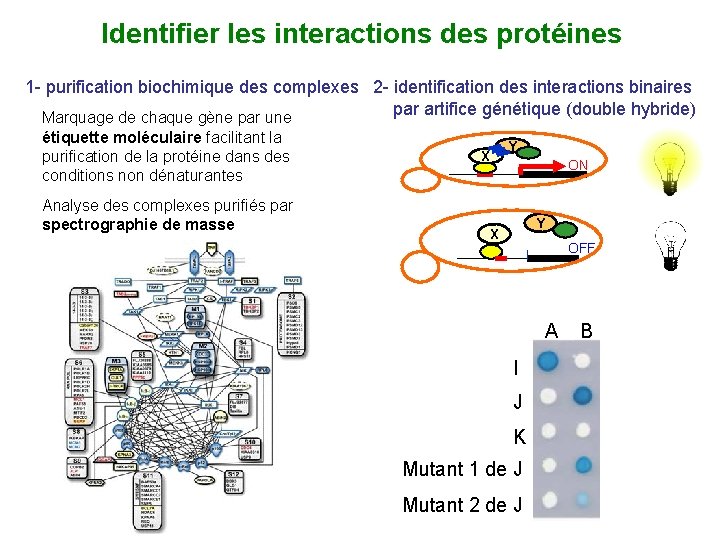
Identifier les interactions des protéines 1 - purification biochimique des complexes 2 - identification des interactions binaires par artifice génétique (double hybride) Marquage de chaque gène par une étiquette moléculaire facilitant la purification de la protéine dans des conditions non dénaturantes Analyse des complexes purifiés par spectrographie de masse X Y ON Y X OFF A I J K Mutant 1 de J Mutant 2 de J B

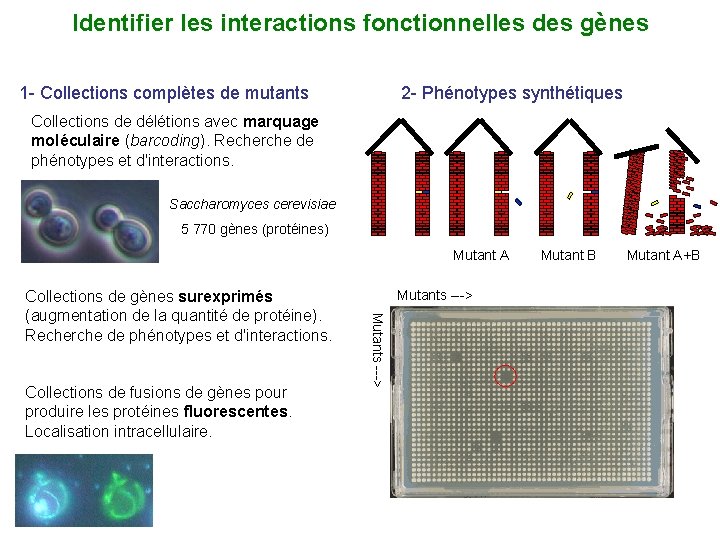
Identifier les interactions fonctionnelles des gènes 1 - Collections complètes de mutants 2 - Phénotypes synthétiques Collections de délétions avec marquage moléculaire (barcoding). Recherche de phénotypes et d'interactions. Saccharomyces cerevisiae 5 770 gènes (protéines) Mutant A Collections de fusions de gènes pour produire les protéines fluorescentes. Localisation intracellulaire. Mutants ---> Collections de gènes surexprimés (augmentation de la quantité de protéine). Recherche de phénotypes et d'interactions. Mutant B Mutant A+B

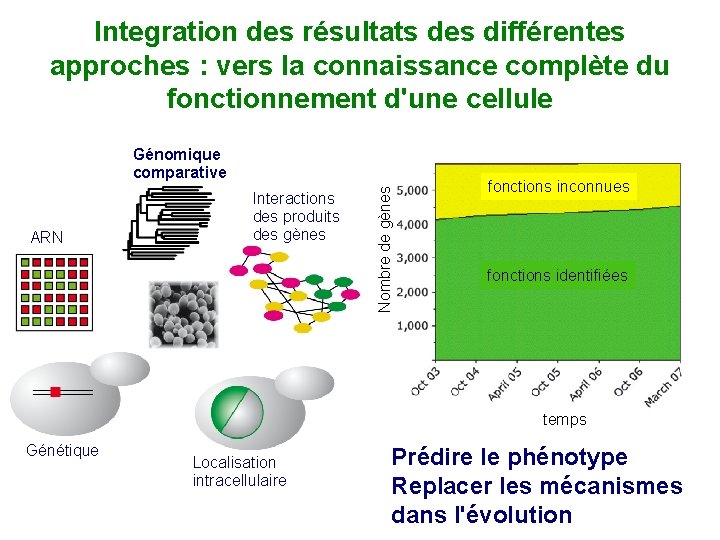
Integration des résultats des différentes approches : vers la connaissance complète du fonctionnement d'une cellule ARN Interactions des produits des gènes Nombre de gènes Génomique comparative fonctions inconnues fonctions identifiées temps Génétique Localisation intracellulaire Prédire le phénotype Replacer les mécanismes dans l'évolution

Et maintenant ?

Le "dogme central" de la biologie moléculaire (actuel) 1953 1970 -1980 2008 Réplication ADN ADN Transcription ARN Traduction Protéine Transcription réverse Transcription ARN Traduction Protéine Epissage Edition Transcription reverse Evolution Formation de gènes Epigénèse Transcription multiple ARN Traduction Protéine S Epissage Edition Régulation

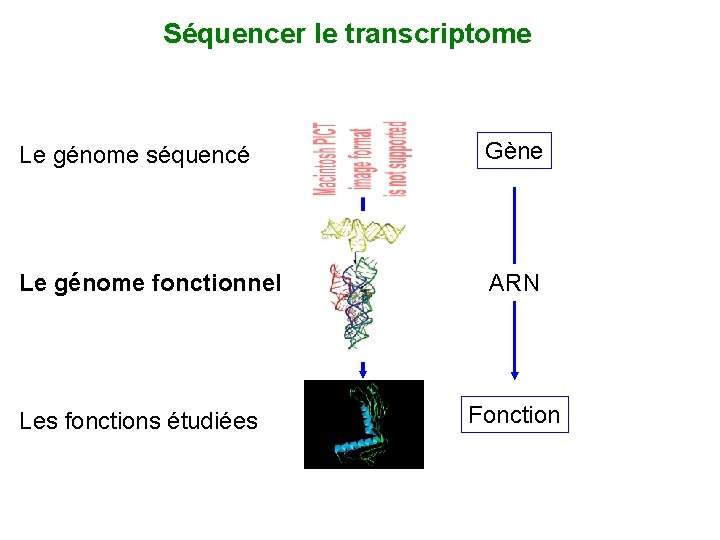
Séquencer le transcriptome Le génome séquencé Gène Le génome fonctionnel ARN Les fonctions étudiées Fonction

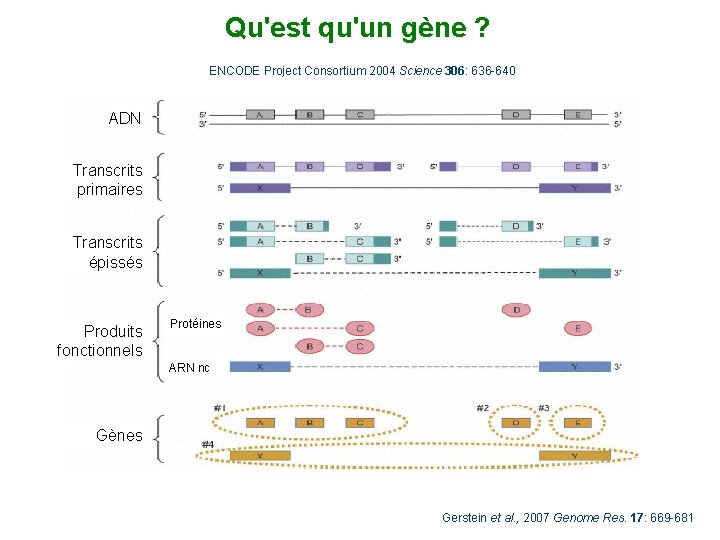
Qu'est qu'un gène ? ENCODE Project Consortium 2004 Science 306: 636 -640 ADN Transcrits primaires Transcrits épissés Produits fonctionnels Protéines ARN nc Gènes Gerstein et al. , 2007 Genome Res. 17: 669 -681

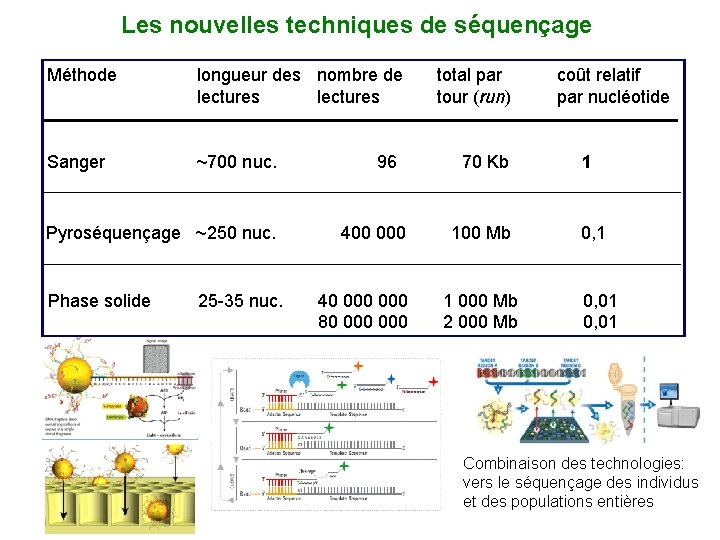
Les nouvelles techniques de séquençage Méthode longueur des nombre de lectures Sanger ~700 nuc. Pyroséquençage ~250 nuc. Phase solide 25 -35 nuc. 96 400 000 40 000 80 000 total par tour (run) 70 Kb 100 Mb 1 000 Mb 2 000 Mb coût relatif par nucléotide 1 0, 01 Combinaison des technologies: vers le séquençage des individus et des populations entières

Quelques grands projets en cours ou annoncés ENCyclopedia Of DNA Elements (ENCODE) Universités américaines, NHGRI But: identifier tous les éléments fonctionnels du génome humain 1000 genomes Consortium international But: cartographier le polymorphisme génétique de la population humaine (1000 individus) Origine de la multicellularité NHGRI But: identifier les gènes et complexes impliqués dans l'apparition de la vie multicellulaire (animaux, champignons, protistes) Microbiome humain Plusieurs centres de séquençage américain But: identification exhaustive de la flore microbienne commensale de l'homme Encyclopédie génomique des bactéries et archaea JGI But: étudier toutes les branches phylogénétiques de l'arbre de la vie (procaryote) Bar coding of life 130 organisations, 43 pays But: attribuer une signature moléculaire standard à chaque espèce identifiée

Qu'est ce que la vie ? principes physico-chimiques + hérédité génome
 Grand corps malade train
Grand corps malade train Composition dragibus
Composition dragibus Tu fais quoi le week end
Tu fais quoi le week end Quest ce que le tourisme
Quest ce que le tourisme Quest-ce que le management
Quest-ce que le management Comment ne pourrais je te louer
Comment ne pourrais je te louer Quest 2 uf
Quest 2 uf Jason and the golden fleece moral lesson
Jason and the golden fleece moral lesson Leading motives quest
Leading motives quest Ivory
Ivory The quest archetype
The quest archetype Statquest slides
Statquest slides Sempre caro mi fu quest'ermo colle significato
Sempre caro mi fu quest'ermo colle significato Slidetodoc.com
Slidetodoc.com Matthew brumley
Matthew brumley Touching spirit bear webquest
Touching spirit bear webquest Webquest co to jest
Webquest co to jest Lions quest program
Lions quest program Quest tec valves
Quest tec valves Eastman kodak's quest for a digital future
Eastman kodak's quest for a digital future Brownie quest journey
Brownie quest journey Admt microsoft
Admt microsoft Web quest
Web quest Metoda quest
Metoda quest Vision quest delaware
Vision quest delaware Quest
Quest Global mindset definition
Global mindset definition Statsquest
Statsquest Eft quests
Eft quests Newman projection practice
Newman projection practice Idaho food stamp balance
Idaho food stamp balance Pirate math equation quest
Pirate math equation quest Quest webtech
Quest webtech Lockheed code quest
Lockheed code quest Lenape vision quest
Lenape vision quest South america webquest
South america webquest Ahe quest
Ahe quest Kansas health quest portal
Kansas health quest portal Quest vdi
Quest vdi Inmersive portals
Inmersive portals Christmas around the world qr quest
Christmas around the world qr quest