Ogni proteina posso clonarla in infiniti modi Come





















- Slides: 35

Ogni proteina posso clonarla in infiniti modi: Come scelgo le sequenze a. a. da clonare? Su quali parametri posso basarmi per scegliere bene?

Scelta delle sequenze da clonare ü Rational design: • Omologie di sequenze con proteine note • Identificazione di domini funzionali mediante predizioni di struttura 2 D e 3 D

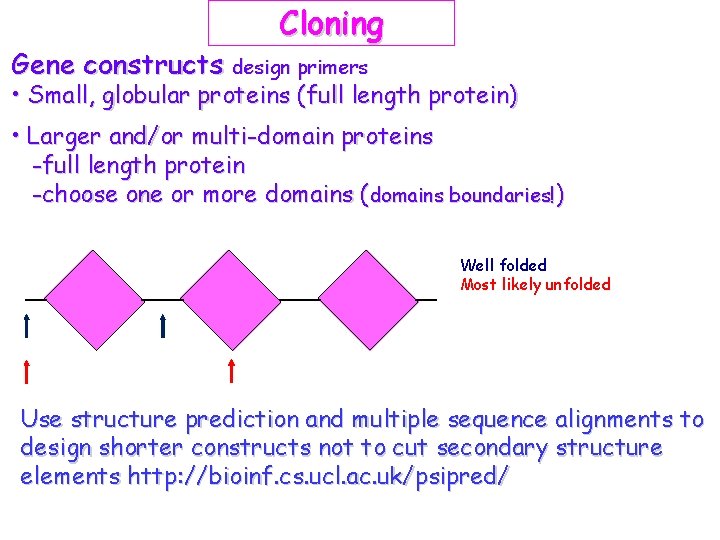
Cloning Gene constructs design primers • Small, globular proteins (full length protein) • Larger and/or multi-domain proteins -full length protein -choose one or more domains (domains boundaries!) Well folded Most likely unfolded Use structure prediction and multiple sequence alignments to design shorter constructs not to cut secondary structure elements http: //bioinf. cs. ucl. ac. uk/psipred/

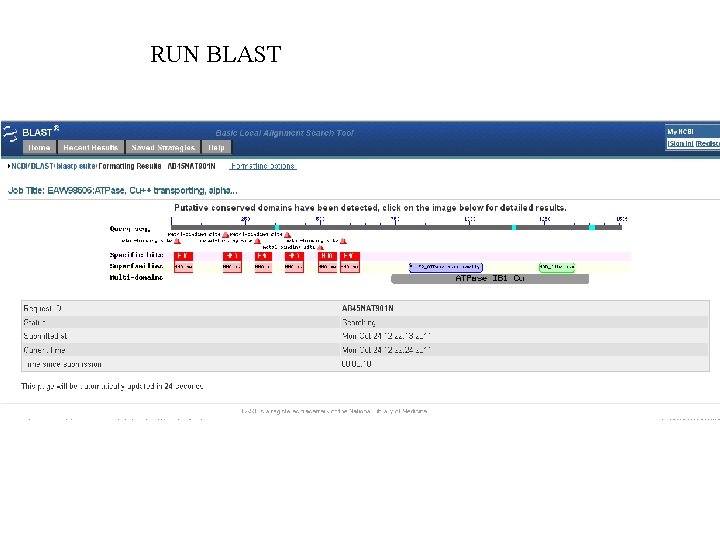
RICERCHE BIOINFORMATICHE ü SEQUENZA NUCLEOTIDICA Gene. Bank http: //www. ncbi. nlm. nih. gov/sites/entrez Ensembl http: //www. ensembl. org/index. html ü SEQUENZA PROTEICA E INFORMAZIONI SULLA PROTEINA Swissprot website http: //www. ebi. ac. uk/swissprot/ Ex. PASy Proteomics Server http: //www. expasy. org/proteomics Uniprot http: //www. uniprot. org/ ü INFORMAZIONI SU SNPs (is a free public archive for genetic variation within and across different species) db. SNPs http: //www. ncbi. nlm. nih. gov/projects/SNP/ ü RICERCHE DI OMOLOGIE E DOMINI CONSERVATI BLAST http: //www. ncbi. nlm. nih. gov/blast/Blast. cgi? CMD=Web&PAGE_TYPE=Blast. Home Pfam http: //pfam. sanger. ac. uk/ SMART http: //smart. embl-heidelberg. de/


RUN BLAST


Pfam

Pfam

Pfam

http: //smart. emblheidelberg. de/smart/job_status. pl? jobid=150217145174205841319 494839 v. VWWz. Ood. NA

ü ALLINEAMENTI DI SEQUENZE Clustal. W http: //npsa-pbil. ibcp. fr/cgibin/npsa_automat. pl? page=npsa_clustalw. html ü INFORMAZIONI STRUTTURALI PDB http: //www. rcsb. org/pdb/home. do ü PREDIZIONE DI REGIONI TRANSMEMBRANA http: //www. sbc. su. se/~miklos/DAS/ http: //www. cbs. dtu. dk/services/TMHMM-2. 0/, http: //www. ch. embnet. org/software/TMPRED/form. html ü PREDIZIONE DI SEQUENZE SEGNALE http: //www. cbs. dtu. dk/services/Signal. P/ ü PREDIZIONI TOPOLOGICHE IUPRED http: //iupred. enzim. hu/ Fold. Index http: //bip. weizmann. ac. il/fldbin/findex

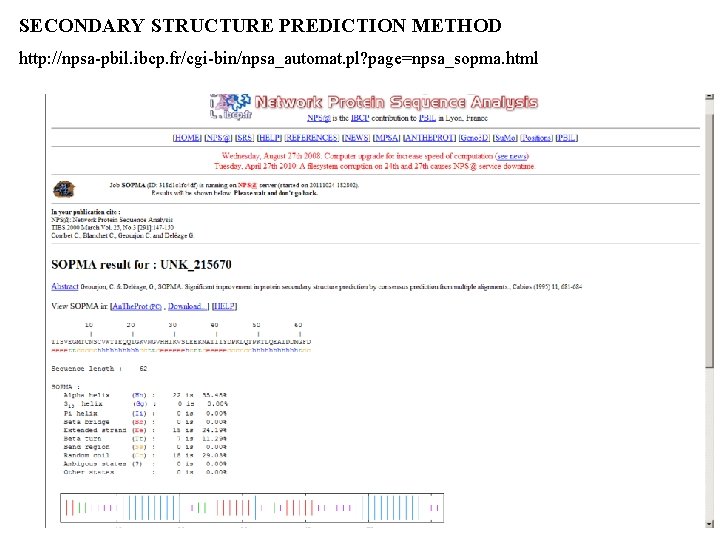
SECONDARY STRUCTURE PREDICTION METHOD http: //npsa-pbil. ibcp. fr/cgi-bin/npsa_automat. pl? page=npsa_sopma. html

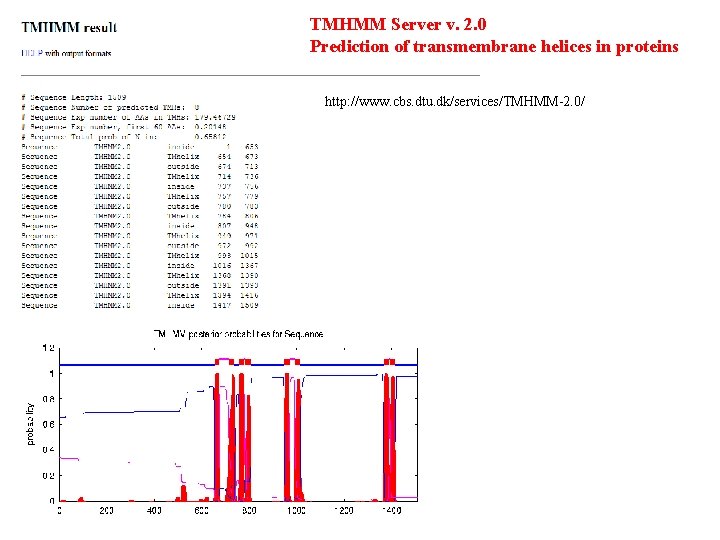
TMHMM Server v. 2. 0 Prediction of transmembrane helices in proteins http: //www. cbs. dtu. dk/services/TMHMM-2. 0/

PREDIZIONE DI SEQUENZE SEGNALE


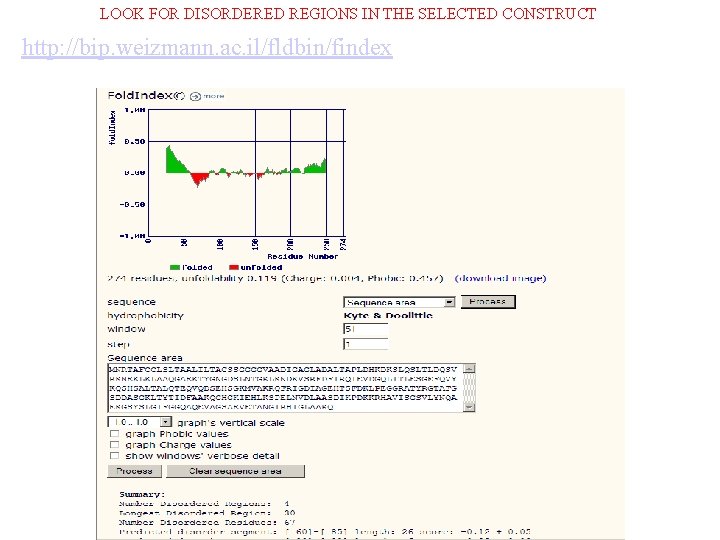
LOOK FOR DISORDERED REGIONS IN THE SELECTED CONSTRUCT http: //bip. weizmann. ac. il/fldbin/findex

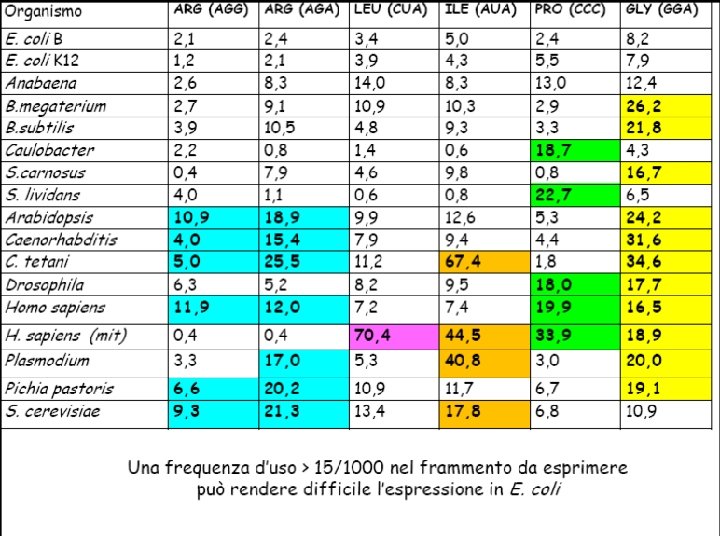
ü ALTRE ANALISI DI SEQUENZA Presenza di codoni rari http: //nihserver. mbi. ucla. edu/RACC N-end rule (Varshavsky, A. (1996) Proc. Natl. Acad. Sci. USA 93, 12142 -1214)


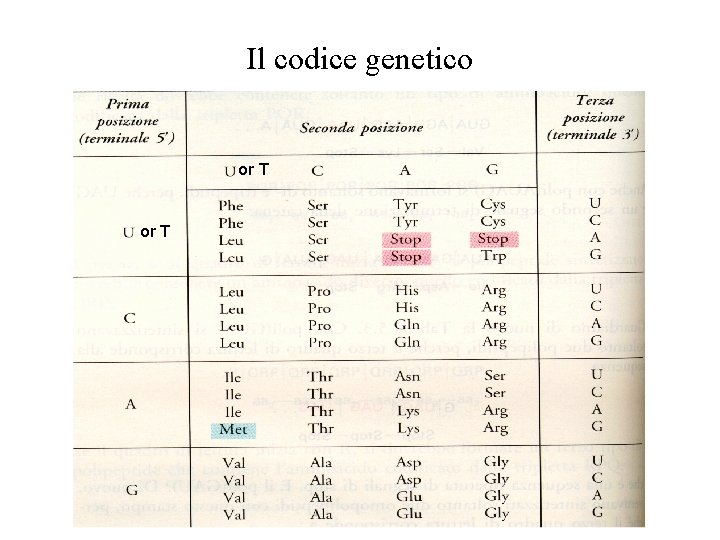
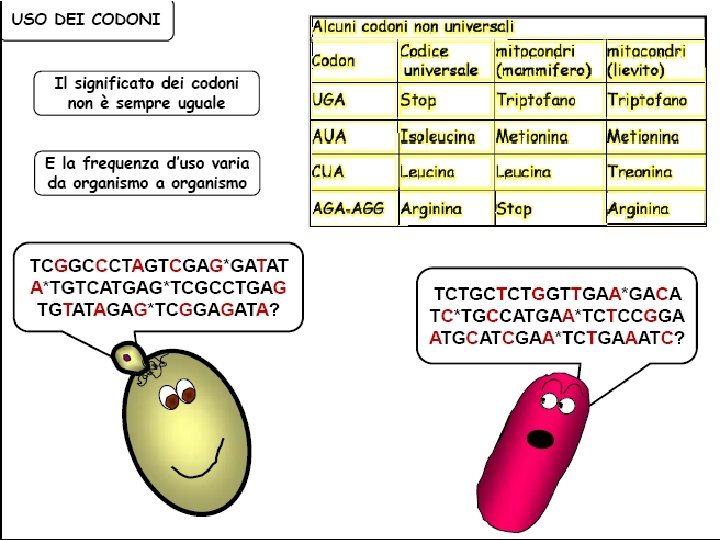
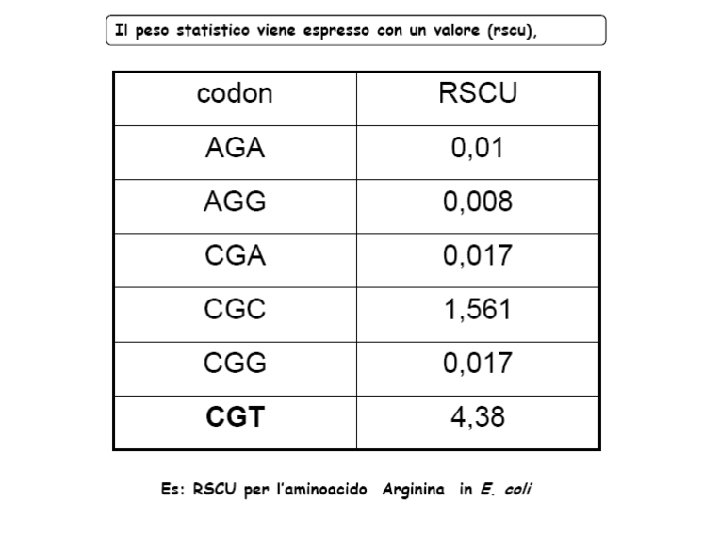
Il codice genetico or T


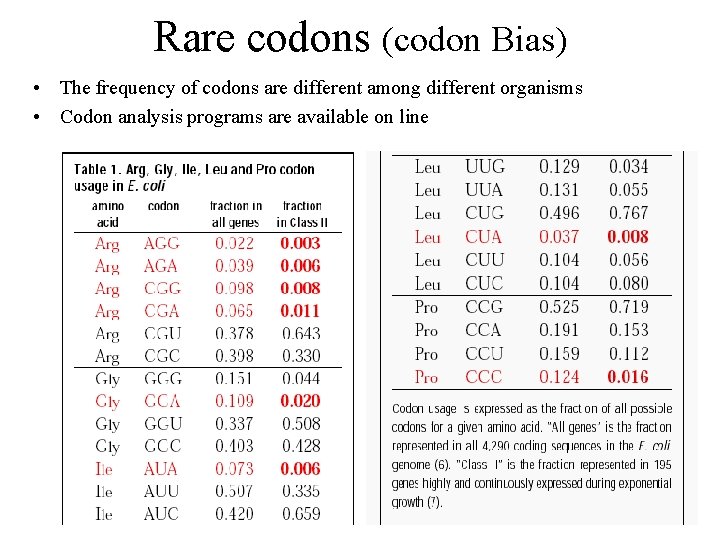
Rare codons (codon Bias) • The frequency of codons are different among different organisms • Codon analysis programs are available on line







Espressione di SOD 1 umana (HSOD) Amplificazione del gene SOD 1 da c. DNA umano via PCR Preparazione del vettore (plasmide) di espressione Inserimento del gene in vettore di espressione Trasformazione del vettore in ceppo di E. coli Screening delle colonie Coltura batterica Isolamento della proteina Purificazione Caratterizzazioni biofisiche preliminari



Amplificazione del gene SOD 1 Direct Primer Inizio seq. SOD trascritta Reverse Primer

Condizioni di reazione H 2 O sterile Buffer per Taq (10 x) d. NTPmix (10 m. M ognuno, 200 M ognuno fin) Templato (c. DNA genomico umano) Primer SODHInd. III (100 pmol/ l) Primer SODSal. I (100 pmol/ l) Taq polimerasi* 5 U/ l *Aggiungere la Taq alla fine Cicli 94 °C 55 °C 72 °C 15 °C 5’ 30’’ 60’’ 2’ 10’ O/N 30 cicli 36. 5 l 5. 0 l 1. 0 l 0. 5 l 50. 0 l

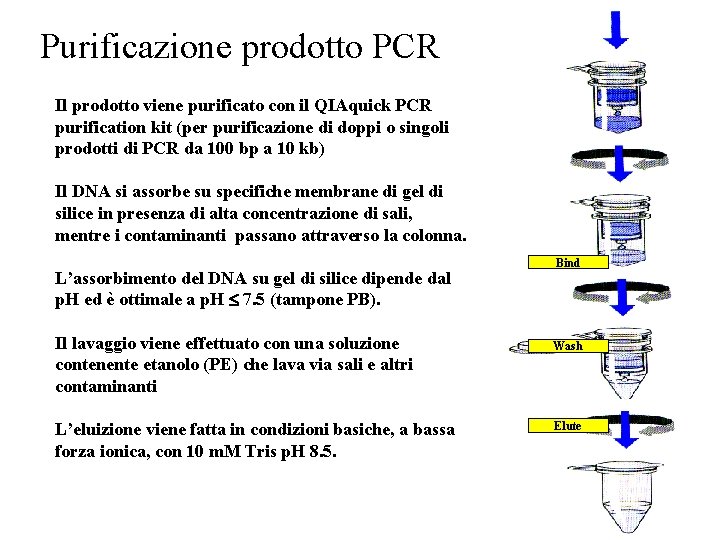
Purificazione prodotto PCR Il prodotto viene purificato con il QIAquick PCR purification kit (per purificazione di doppi o singoli prodotti di PCR da 100 bp a 10 kb) Il DNA si assorbe su specifiche membrane di gel di silice in presenza di alta concentrazione di sali, mentre i contaminanti passano attraverso la colonna. L’assorbimento del DNA su gel di silice dipende dal p. H ed è ottimale a p. H 7. 5 (tampone PB). Il lavaggio viene effettuato con una soluzione contenente etanolo (PE) che lava via sali e altri contaminanti L’eluizione viene fatta in condizioni basiche, a bassa forza ionica, con 10 m. M Tris p. H 8. 5. Bind Wash Elute

Protocollo di purificazione del frammento PCR
 Zymoliase
Zymoliase Interacciones proteina proteina
Interacciones proteina proteina Come rico
Come rico Perché i numeri naturali sono infiniti
Perché i numeri naturali sono infiniti Differenza tra infiniti e infinitesimi
Differenza tra infiniti e infinitesimi Testogenix reviews
Testogenix reviews Para poder falar
Para poder falar Felipenses 4:5
Felipenses 4:5 Aprendi que nao posso exigir
Aprendi que nao posso exigir Tudo posso naquele que me fortalece
Tudo posso naquele que me fortalece Texto seja feliz voce pode ter defeitos
Texto seja feliz voce pode ter defeitos Lauro trevisan eu posso
Lauro trevisan eu posso Não posso mais olhar nosso jardim
Não posso mais olhar nosso jardim Deus de aliança deus de promessas letra
Deus de aliança deus de promessas letra Antico sono ubriacato dalla voce
Antico sono ubriacato dalla voce 6 modi di rappresentare lo spazio
6 modi di rappresentare lo spazio Metode transportasi
Metode transportasi Frasi interrogative in francese
Frasi interrogative in francese Modi verbali
Modi verbali Modus deutsch grammatik
Modus deutsch grammatik Verbo cantare in tutti i modi
Verbo cantare in tutti i modi Verbi transitivi e intransitivi
Verbi transitivi e intransitivi Elettricità statica scuola primaria
Elettricità statica scuola primaria Modo congiuntivo
Modo congiuntivo Are ere ire nastavci
Are ere ire nastavci Transportation problem by modi method
Transportation problem by modi method Modi lipi
Modi lipi Diritti reali
Diritti reali Metode modi riset operasi
Metode modi riset operasi Come si traduce la proposizione finale in latino
Come si traduce la proposizione finale in latino Infinito presente
Infinito presente Ad ogni azione corrisponde una reazione
Ad ogni azione corrisponde una reazione Ogni uomo semplice porta in cuore un sogno
Ogni uomo semplice porta in cuore un sogno Ogni favola ne ha una
Ogni favola ne ha una Ogni azione ha una reazione
Ogni azione ha una reazione A ciascun giorno basta la sua pena in latino
A ciascun giorno basta la sua pena in latino


























































