Nama Kelompok 1 Lailatul Hikmah 121810301008 2 Fajrin





















- Slides: 24


Nama Kelompok: 1. Lailatul Hikmah 121810301008 2. Fajrin Nurul Hikmah 121810301022 3. Anita Rahman 4. Lailatul Badriyah 121810301036 5. R. Yuni Ristanti S 121810301039 6. Indah Purwanti 121810301064 7. Mar’atus Sholihah 121810301078 121810301027 Kelompok: H

Pendekatan komputasi untuk Desain Struktur Obat Berdasarkan Senyawa Antiviral dari Alam untuk Herpesviridae

PENDAHULUAN Struktur tiga dimensi dari suatu target bimolekular dapat mengalami perubahan secara dramatis karena adanya perbedaan sifat struktural dan elektronik tentang ligan. Hal ini telah menginspirasi perkembangan pesat dari desain obat berbasis struktur. Desain obat berbasis struktur adalah dasar statistik dari struktur 3 -D dari suatu target biologis

Desain obat berbasis struktur dibagi menjadi dua yaitu berbasis ligan dan berbasis reseptor Desain obat berdasarkan struktur memiliki lima tahapan yaitu Ø Langkah pertama, mengidentifikasi target obat atau reseptor yang merupakan makromolekul penting untuk penyakit yang menyebabkan melalui materi genetik atau proteomik. Ø Langkah kedua melibatkan informasi struktural akurat biomolekul untuk memahami fungsi dan aktivitas target obat.

Ø Langkah ketiga adalah identifikasi situs mengikat di mana ligan dapat memasang dan menghambat protein penyebab penyakit. Ø Langkah keempat, studi komputasi digunakan untuk mengevaluasi kompleks yang mengikat inhibitor. Ø Langkah terakhir berlaku untuk evaluasi potensi senyawa timbal sebelum melanjutkan ke uji praklinik.

Ø Pemodelan homologi protein digunakan untuk desain ligan Ø Identifikasi dan visualisasi protein adalah titik awal untuk desain struktur obat berbasis aplikasi (SBDD) Ø Ukuran dan bentuk protein menentukan geometri tiga dimensi ligan Ø Auto Dock adalah alat docking otomatis yang dirancang untuk memprediksi bagaimana molekul kecil mengikat reseptor dengan struktur 3 D Ø Selain itu juga digunakan sebagai parameter dalam pengikatan ligan dengan sejumlah konformasi yang berbeda, serta untuk menentukan energi minimum yang digunakan untuk mengikat ligan

SENYAWA ANTIVIRUS Bahan alam yang digunakan untuk mendapatkan 21 senyawa antivirus, yaitu: v Bawang putih (Allium sativum) v Daun Mimba (Azadirachta indica) v Echinacea v Jahe (Zingiber officinale) v Goldenseal v Kelabat (Methika /Fenugreek) v Peganum harmala v Kembang Telang (Clitorea ternatea) v Ruku- ruku (Ocimum sanctum) v Scrophularia frutescens

TINJAUAN PUSTAKA Ø Metode yang paling populer untuk mengidentifikasi obat adalah dengan studi penyakit berdasarkan pengamatan dan identifikasi gen yang terkait dengan penyakit tersebut Ø Sebagian besar data biologis digunakan untuk identifikasi dan karakterisasi faktor virulen dalam patogen

Metodologi Metode desain obat ini bioinformatikamelibatkan, proteomik, biokimia, dan pemodelan komputer dari struktur protein 3 -dimensi. pemodelan komparatif menggunakan MODELLER 9. 9. Model terbaik dipilih atas dasar DOPE Score dan analisis Plot RC. Sifat senyawa antiviral yang diusulkan dilihat menggunakan Molinspiration dan Lipinski aturan lima. Docking ligan dengan protein target menggunakan Auto. Dock 4. 4.

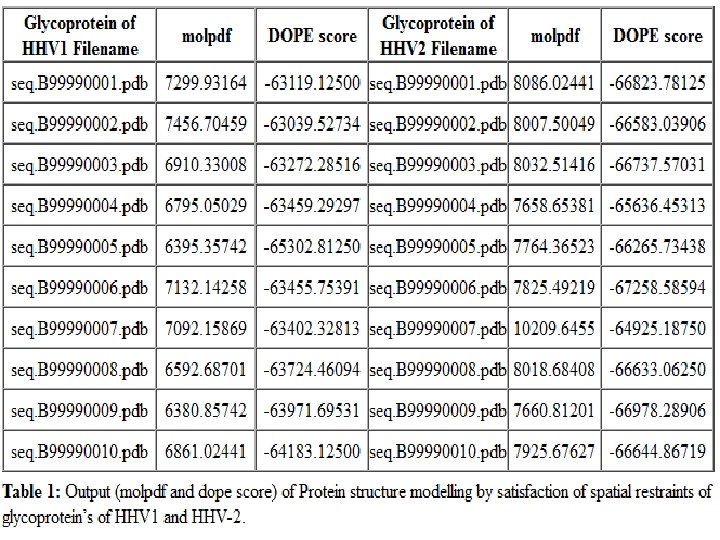
Pemodelan Homolog Struktur tiga dimensi (pdb) polimerase DNA dan pengembangan model glikoprotein dengan mempertimbangkan struktur protein yang sesuai template diteliti dengan baik dan diidentifikasi dengan pencarian kemiripan Kemudian memilih sepuluh model yang mungkin untuk glikoprotein menggunakan modeller 9. 9 seperti yang ditunjukkan dalam tabel 1. analisis plot Ramachandran untuk 20 glikoprotein yang sama dari HHV-1 dan HHV-2 diwakili dalam tabel 2.



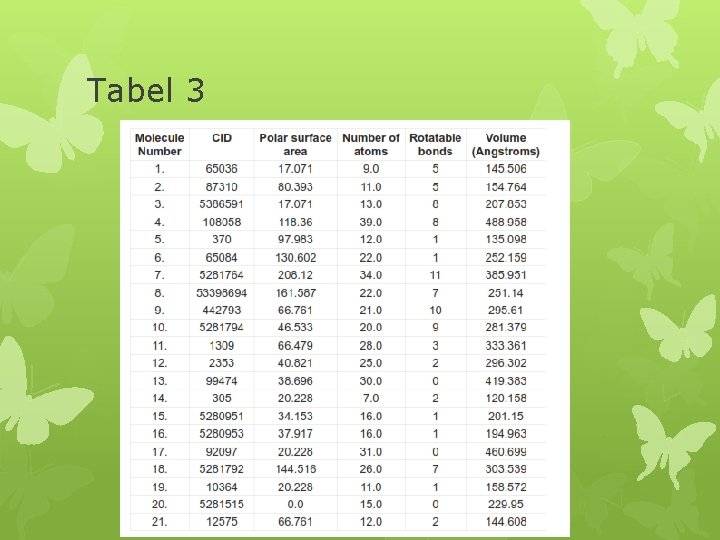
Alat yang digunakan dalam penelitian ini adalah Molinspiration Fungsi dari Molinspiration adalah mengevaluasi sifat fisik dan kimia dari senyawa yang akan yang digunakan sebagai senyawa antivirus yang telah tercantum dalam tabel 3.

Tabel 3

Besarnya volume dari molekul yang berkisar 300 unit ditemukan dalam senyawa nomer 4, 7, 11, 13 dan 17. Berdasarkan data tersebut volume tertinggi di antara lima senyawa tersebut adalah senyawa nomor empat. Rotatable bonds meliputi fleksibilitas molekul dan bioavailabilitas dari obat. Hanya dua senyawa (7 dan 9) yang memiliki rotatable bonds ≥ 10.

Docking molekul

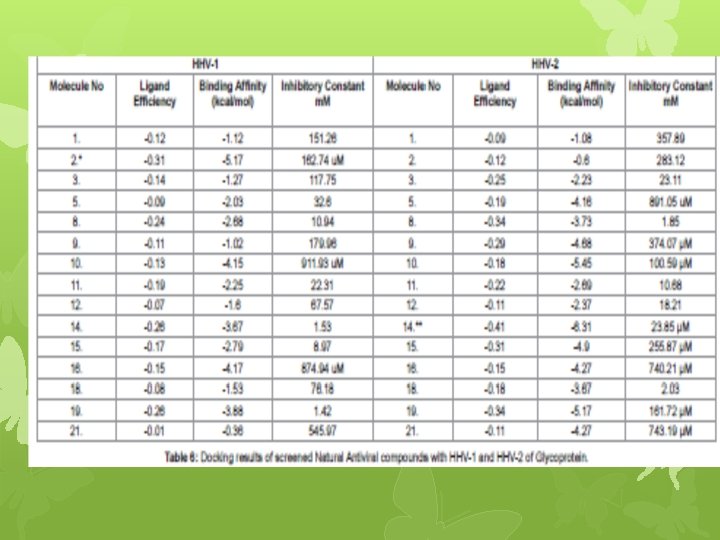
Docking molekular menggunakan simulasi komputasi yang memprediksi orientasi ligan untuk reseptor ketika berinteraksi satu sama lain untuk membentuk kompleks yang stabilitasnya tinggi. Dalam tulisan ini, docking menunjukkan penggunaan alat Auto. Dock 4. 2, untuk memprediksi afinitas, efisiensi ligan dan penghambat konstan.

Hasil Auto. Dock untuk polimerase DNA (HHV-I dan HHV-II) mewakili dalam tabel 5 dan 6. Ligan Efisiensi (LE) mengukur rasio energi bebas Gibbs untuk jumlah atom non hidrogen dari senyawa.



DISCUSSION Model homologi digunakan untuk memprediksi struktur protein menggunakan DOPE potensial Keberhasilan desain struktur obat dipengaruhi oleh: Kepolaran Volum molekul Rotasi atom Aturan lipinski (log ≤ 5, MW ≤ 500, jumlah akseptor ikatan hidrogen ≤ 10, dan jumlah donor ikatan hidrogen ≤ 5) Docking umumnya tergantung pada: Efesiensi ligan Afinitas Konstanta penghambatan

KESIMPULAN Mendesain obat dengan bantuan komputer (modeller, Auto. Dock dan Molinspiration) telah menjadi komponen penting dari bioinformatika untuk mengidentifikasi senyawa anti-virus. Senyawa alami dipilih sebagai ligan yang mampu mengendalikan infeksi pada manusia melalui glikoprotein dan DNA polimerase Aturan Lipinski juga mendukung empat ligan (nomor 10364, 5281794, 87310 dan 305 )untuk bioavailabilitas

 Apa itu ktb
Apa itu ktb Nama kelompok yang bagus
Nama kelompok yang bagus Swakelola adalah
Swakelola adalah Sekunder dan kelompok
Sekunder dan kelompok Model kelompok kerja yang efektif
Model kelompok kerja yang efektif Kelompok deskriptif dan kelompok preskriptif
Kelompok deskriptif dan kelompok preskriptif Setiap kelompok terdiri dari
Setiap kelompok terdiri dari Klasifikasi kelompok sosial berdasarkan pencapaian tujuan
Klasifikasi kelompok sosial berdasarkan pencapaian tujuan Nama kelompok akuntansi
Nama kelompok akuntansi Volume tabung
Volume tabung Nama kelompok 2
Nama kelompok 2 Nama kelompok tugas
Nama kelompok tugas Nama kelompok berbau teknologi
Nama kelompok berbau teknologi Background ppt nama kelompok
Background ppt nama kelompok Nadiva addina
Nadiva addina Nama anggota kelompok
Nama anggota kelompok Nama kelompok tentang lingkungan
Nama kelompok tentang lingkungan Nama kelompok matematika yang keren
Nama kelompok matematika yang keren Gambar sitoskeleton
Gambar sitoskeleton Judul dan nama anggota kelompok informasi gambar informasi
Judul dan nama anggota kelompok informasi gambar informasi Bahasa inggris kelompok 2
Bahasa inggris kelompok 2 Judul dan nama anggota kelompok
Judul dan nama anggota kelompok Nama kelompok berbau teknologi
Nama kelompok berbau teknologi Nama kelompok matematika
Nama kelompok matematika Desain nama kelompok
Desain nama kelompok