mutazioni puntiformi Vincenzo Nigro Dipartimento di Patologia Generale






































- Slides: 52

mutazioni puntiformi Vincenzo Nigro Dipartimento di Patologia Generale Seconda Università degli Studi di Napoli Telethon Institute of Genetics and Medicine (TIGEM)

classificazione funzionale delle mutazioni 1. allele equivalente 2. allele ipomorfo 3. allele amorfo 4. allele ipermorfo 5. allele neomorfo 6. allele antimorfo

1. allele equivalente • variazione che non modificano né la quantità, né la qualità biochimica e funzionale del prodotto genico • il prodotto genico risulta invariato e normalmente localizzato • esempi sono le circa 12, 000 differenze della sequenza del DNA codificante che si osservano nella popolazione umana che non hanno alcuna conseguenza patologica

2. allele ipomorfo • variazione della sequenza del DNA che riduce la quantità di prodotto genico e/o la sua qualità funzionale • tali alleli sono silenti e recessivi e possono agire più come modificatori del fenotipo che come causa diretta di patologia • alleli ipomorfi possono però essere causa di malattia se in emizigosi • esempio: gli alleli ipomorfi del gene della distrofina localizzato sul cromosoma X che determinano quadri di distrofia muscolare di Becker nei maschi in quanto hanno una singola copia del gene

3. allele amorfo • variazione di sequenza del DNA più drastica: corrisponde classicamente alla delezione (cancellazione) della sequenza codificante del gene • un allele amorfo può essere prodotto da altri tipi di mutazione che abbiano la medesima conseguenza di una delezione totale del gene • causa in emizigosi una malattia genetica quando colpisce una funzione genica essenziale (es emofilia, distrofia muscolare di Duchenne, ecc) • l’allele amorfo in eterozigosi in genere è presente in un portatore sano di una malattia autosomica recessiva • può da solo essere causa di malattia se riguarda un gene in cui il 50% del dosaggio (prodotto dall’altro allele non mutato) è insufficiente a mantenere lo stato di salute (aploinsufficienza)

4. allele ipermorfo • ipermorfo (iper= aumentato) è l’allele che determina l’aumentata quantità o funzione di un prodotto genico • l’allele ipermorfo può essere semplice o avere una combinazione di altri effetti come ad esempio, quello di essere presente in una localizzazione impropria o in un tempo sbagliato • è associato di regola ad un tratto genetico dominante, in quanto l’aumentata espressione/funzione non può essere limitata dall’allele non mutato • un esempio è l’aumentata funzione del recettore per l’FGF 3 che causa l’acondroplasia (nanismo dismorfico) che è appunto a trasmissione autosomica dominante

5. allele neomorfo • neomorfo (neo=nuovo) definito come causato da una mutazione che porta ad un prodotto genico nuovo o una funzione nuova • si distingue solo didatticamente dall’allele ipermorfo, in quanto è in pratica una variante che è difficile da distinguere nelle singole condizioni • valgono le stesse considerazioni fatte per l’allele ipermorfo sulla natura dominante della mutazione • in alcune forme di cancro l’allele neomorfo è una chimera di due geni, dovuta ad una traslocazione cromosomica, come il cromosoma di fusione Philadelphia con la comparsa di nuove proteine Bcr-abl in casi di leucemia mieloide cronica

6. allele antimorfo o dominante negativo • antimorfo (anti=contro) definito come causato da una mutazione che porta ad un prodotto genico antagonistico • è il risultato di una mutazione che colpisce un gene il cui prodotto proteico funziona in cooperazione con altre proteine • particolari mutazioni rendono la proteina di disturbo a tutte le altre pur essendo queste ultime perfettamente normali • un esempio è dato dal collageno in cui più geni (e due alleli per ogni gene) contribuiscono alla formazione delle proteine ciascuno producendo una parte delle catene di base: una mutazione in un solo allele produce un effetto negativo complessivo

classificazione strutturale delle mutazioni 1. sostituzioni 2. piccole inserzioni, delezioni o inserzioni + delezioni contemporaneamente (indels) 3. riarrangiamenti genomici a due (delezioni, duplicazioni) o più punti di rottura (traslocazioni, inversioni ecc. ) 4. copy number variations (CNV) a queste quattro classi appartengono in modo indistinguibile tanto le variazioni innocue quanto le mutazioni causative di malattia

mutazioni I dati di sequenziamento totale del genoma provano che almeno 10 -8 sostituzioni geniche per base si verificano nella prole de novo, cioè senza essere ereditate dai genitori tutte le nuove sostituzioni sono in eterozigosi o in emizigosi se si verificano nei cromosomi sessuali maschili le nuove sostituzioni possono produrre varianti private meno dell’ 1% cadono negli esoni codificanti dei geni • mutazioni silenti, quando l’aminoacido non cambia • mutazioni missenso quando un aminoacido è sostituito da un altro aminoacido • mutazioni nonsenso quando un aminoacido è sostituito da un codone prematuro di terminazione • mutazioni nonstop quando al contrario un codone di terminazione è sostituito da un codone di un aminoacido



Numerazione dei nucleotidi Nucleotidi del c. DNA • Il nucleotide +1 è la A dell’ ATG-codone di inizio della traduzione • Il nucleotide che precede al 5' l’ATG-codone di inizio della traduzione è denominato -1; non esiste una base 0 • Il nucleotide che segue al 3' il codone di terminazione è denominato *1

sostituzioni • le sostituzioni sono indicate dal carattere “>”. Ad esempio, 76 A>C indica che in posizione 76 un’adenina è sostituita da una citosina 88+1 G>T (oppure IVS 2+1 G>T) indica che una guanina è sostituita da una timina in posizione +1 dell’introne 2, posizionato tra i nucleotidi 88 e 89 del c. DNA 89 -2 A>C (oppure IVS 2 -2 A>C) indica che un’adenina è sostituita da una citosina in posizione -2 dell’introne 2, posizionato tra i nucleotidi 88 e 89 del c. DNA


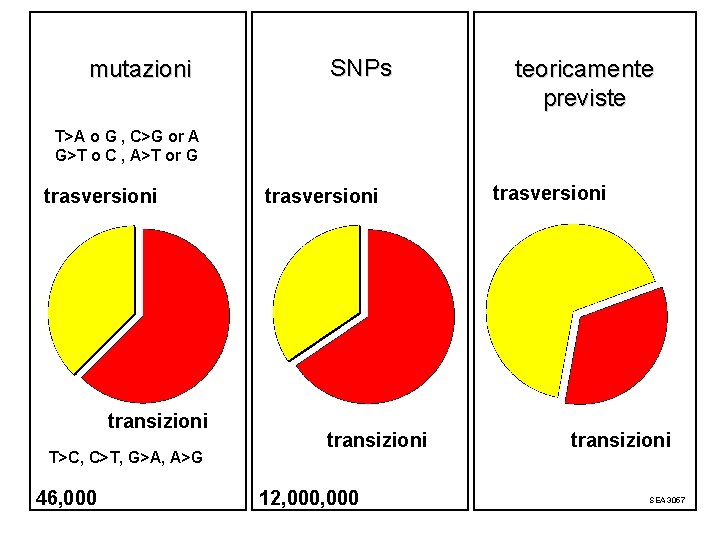
mutazioni SNPs teoricamente previste T>A o G , C>G or A G>T o C , A>T or G trasversioni transizioni T>C, C>T, G>A, A>G 46, 000 trasversioni transizioni 12, 000 trasversioni transizioni SEA 3057

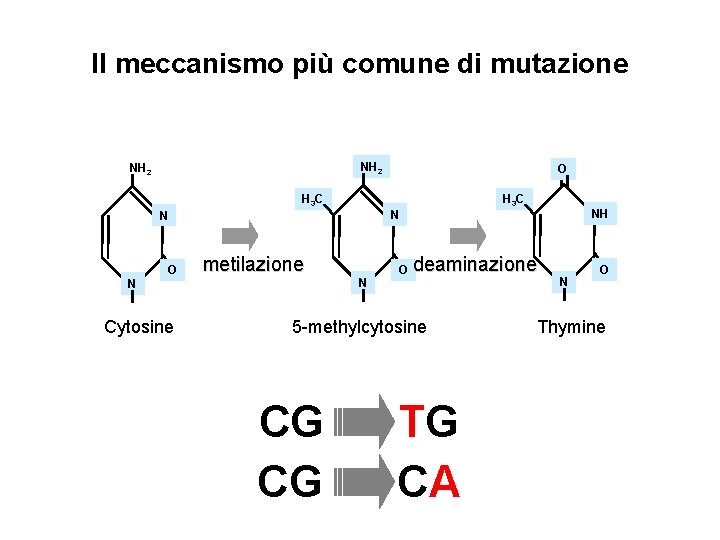
Il meccanismo più comune di mutazione NH 2 H 3 C N Cytosine H 3 C N N O O metilazione N O deaminazione 5 -methylcytosine CG CG NH TG CA N O Thymine

mutazioni puntiformi missenso • Le mutazioni missenso sono quelle in cui il cambiamento determina nel prodotto proteico la sostituzione di un aminoacido con un aminoacido differente • Sebbene queste alterazioni generalmente non provochino conseguenze nella funzionalità della proteina (polimorfismi o varianti) , ci sono casi in cui anche una minima alterazione può avere conseguenze gravi

acrocefalosindattilia sindrome di Apert • 1: 65. 000 alla nascita • craniosinostosi, volta cranica a forma conica • ipertensione endocranica • ritardo mentale • ipoplasia della parte centrale della faccia • sindattilia delle dita delle mani e dei piedi • sordità e atrofia ottica

acrocefalosindattilia sindrome di Apert • tutti i pazienti hanno la stessa mutazione Apert (Cys 755 Gly) del gene human fibroblast growth factor receptor 2 (FGFR 2) • la mutazione è in eterozigosi • de novo • cromosoma 10 q 26 • la sindrome è allelica con Crouzon e Pfeiffer

sindrome di Pfeiffer • alcuni pazienti hanno la mutazione Pfeiffer (Cys 342 Arg) del gene human fibroblast growth factor receptor 2 (FGFR 2) • altri la mutazione Pro 252 Arg in FGFR 1 • la mutazione è in eterozigosi • de novo • cromosoma 10 q 26 • la sindrome è allelica con Crouzon e Apert

disostosi cranio facciale sindrome di Crouzon • alcuni pazienti hanno la mutazione (Cys 342 Tyr) del gene human fibroblast growth factor receptor 2 (FGFR 2) • la mutazione è in eterozigosi • de novo • cromosoma 10 q 26 • la sindrome è allelica con Pfeiffer e Apert con alcune mutazioni in comune

acondroplasia • • nanismo dismorfico (1: 35. 000) arti corti e testa sproporzionatamente più grossa fronte prominente e naso appiattito altezza media 130 cm nei maschi 125 cm nelle femmine • La mutazione è in eterozigosi • Gly 380 Arg nel recettore 3 del "fibroblast growth factor" (FGFR 3) a 4 p 16. 3 • autosomico dominante a penetranza completa

acondroplasia • La mutazione conferisce una funzione aumentata al recettore dell'FGF (allele ipermorfo) che è una tirosinchinasi di membrana • In risposta all'FGF il recettore dimerizza e si fosforila trasducendo un segnale con la funzione di rallentare la proliferazione dei condrociti e quindi la crescita ossea • Topi senza il gene FGF 3 R hanno ossa lunghe e vertebre allungate


frequenza relativa di mutazioni de novo che causano acondroplasia i rapporto all’età paterna

numero di divisioni nelle linea germinale maschile


ipocondroplasia • L'ipocondroplasia ha caratteristiche simili all'acondroplasia, ma di gravità minore con un coinvolgimento craniofacciale inferiore. L'altezza può risultare ai limiti della norma e la malattia viene spesso non diagnosticata. • L'ipocondroplasia è meno omogenea: circa il 70% dei casi è dovuto alla sostituzione N 540 K del gene FGFR 3, mentre non si conosce la mutazione nel restante 30%.

mutazioni puntiformi nonsenso • La mutazione nonsenso è quella in cui la modificazione nucleotidica provoca la creazione di un tripletta di stop, che blocca la sintesi della proteina prematuramente. • In questo caso, la funzionalità della proteina dipenderà dalla posizione dello stop.

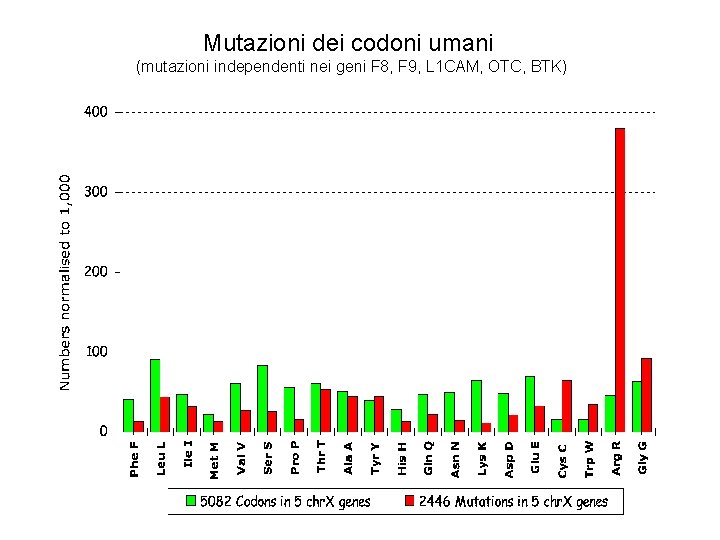
Mutazioni dei codoni umani (mutazioni independenti nei geni F 8, F 9, L 1 CAM, OTC, BTK)

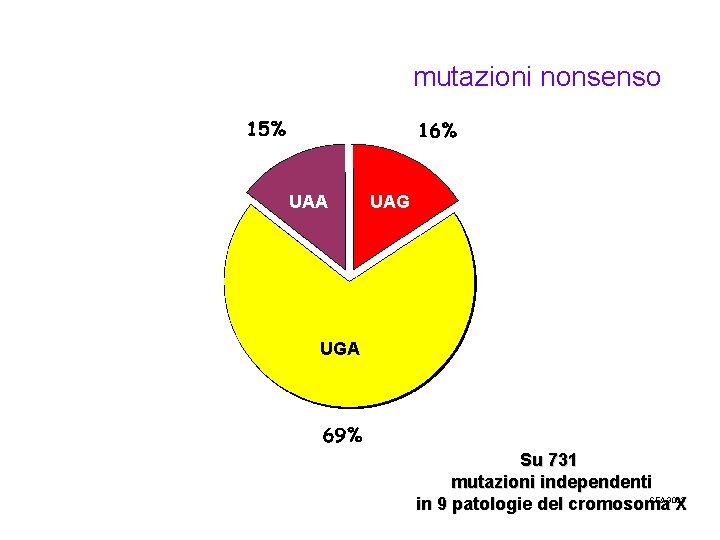
mutazioni nonsenso UAA UAG UGA Su 731 mutazioni independenti SEA 3063 in 9 patologie del cromosoma X

mutazioni frame-shift • Le mutazioni frame-shift o di slittamento del modulo di lettura consistono nell’inserzione o delezione di un numero di nucleotidi non divisibile per 3 (1, 2, 4, 5, 7, 8, 10, ecc. ) conseguente sfasamento della cornice di lettura delle triplette dell'RNA messaggero. • Questa mutazione determina la traduzione non corretta della proteina a valle della mutazione.

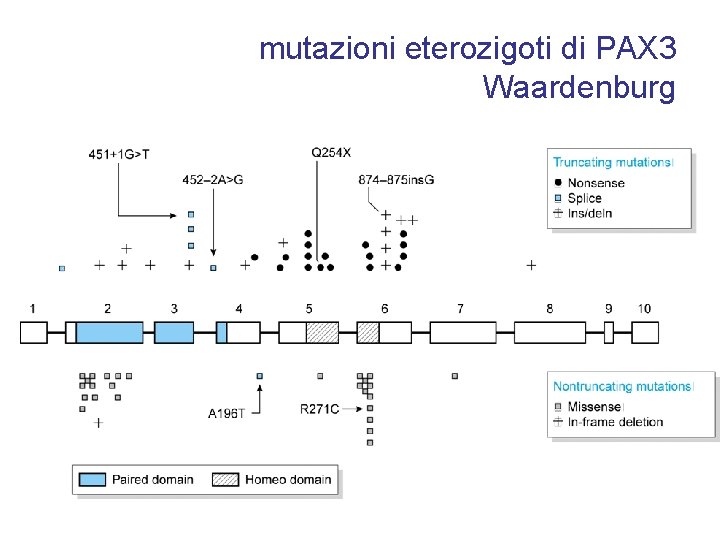
mutazioni eterozigoti di PAX 3 Waardenburg

mutazioni eterozigoti di PAX 3 Waardenburg • sordità (o deficit uditivo di vario livello) bilaterale, • modifiche nella pigmentazione, sia dei capelli (albinismo parziale, in genere piebaldismo) che della pelle, • anomalie nello sviluppo dei tessuti derivati dalla cresta neurale • lateralizzazione del canto mediale • diverso colore degli occhi (eterocromia), di solito uno marrone e l'altro blu


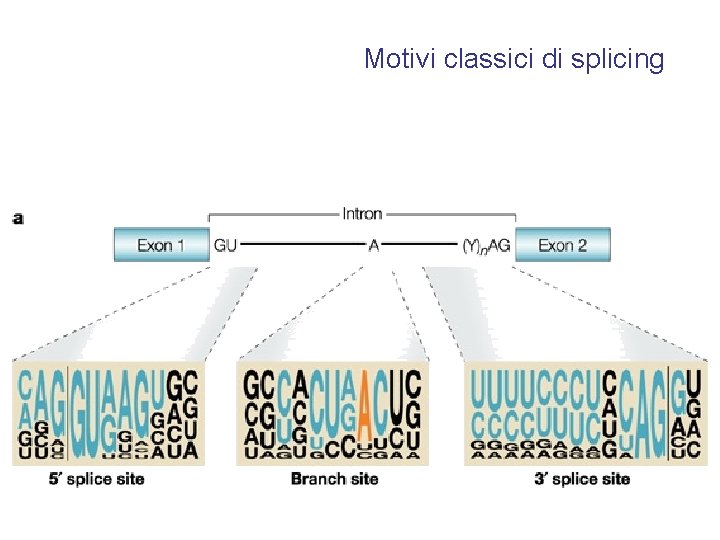
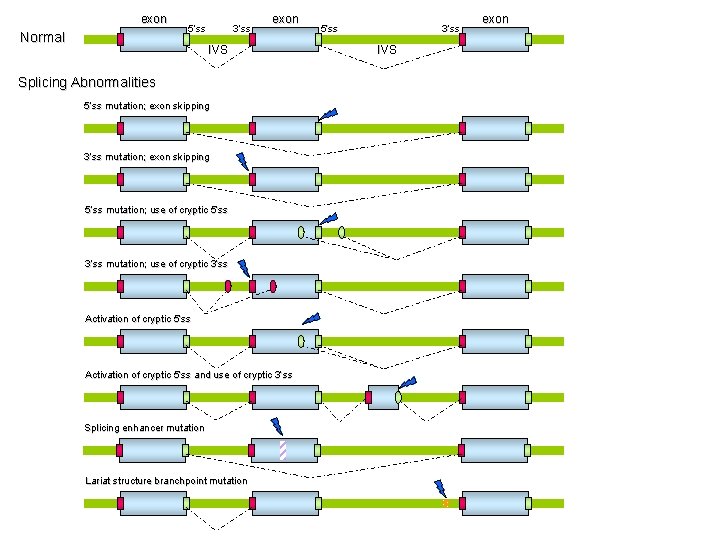
Motivi classici di splicing

Nucleotidi intronici fiancheggianti • inizio dell’introne: il numero dell’ultimo nucleotide dell’esone che precede, il segno + e la posizione nell’introne, ad esempio 77+1 G, 77+2 T, oppure quando il numero dell’esone è noto ed univoco IVS 1+1 G, IVS 1+2 T • fine dell’introne: il numero del primo nucleotide del esone seguente, il segno – e la posizione a monte dell’esone, ad esempio 78 -2 A, 78 -1 G, oppure quando il numero dell’esone è noto ed univoco, IVS 1 -2 A, IVS 1 -2 G


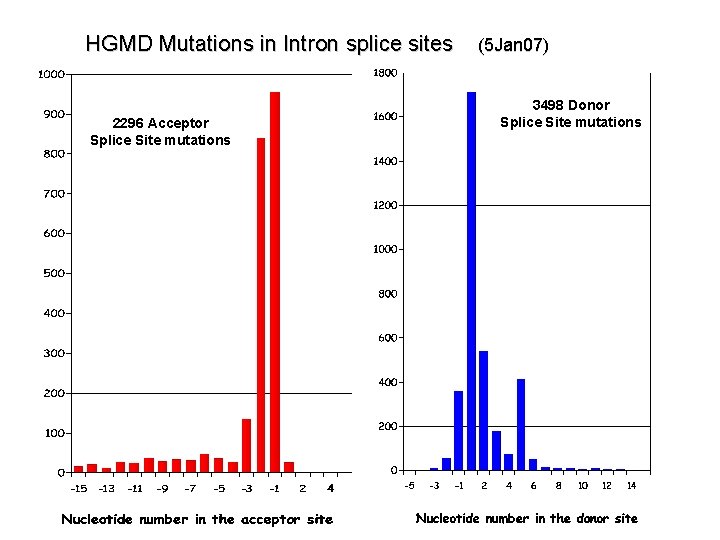
HGMD Mutations in Intron splice sites (5 Jan 07) 2296 Acceptor Splice Site mutations 3498 Donor Splice Site mutations

exon Normal 5’ss 3’ss exon IVS Splicing Abnormalities 5’ss mutation; exon skipping 3’ss mutation; exon skipping 5’ss mutation; use of cryptic 5’ss 3’ss mutation; use of cryptic 3’ss Activation of cryptic 5’ss and use of cryptic 3’ss Activation of cryptic 5’ss Splicing enhancer mutation Lariat structure branchpoint 5’ss 3’ss IVS exon

Progeria Hutchinson-Gilford • invecchiamento precoce • bassa statura, pelle rugosa • calvizie, assenza di tessuto adiposo • aterosclerosi ed infarto

Progeria Hutchinson-Gilford • nuova mutazione in eterozigosi del gene lamina A • la mutazione è in eterozigosi • de novo • cromosoma 1 q 23 • La mutazione non cambia l'aminoacido glicina G 608 G, ma introduce un sito donor di splicing GGT che fa perdere 50 aminoacidi alla proteina • sperimentazione con inibitori di farnesil-trasferasi


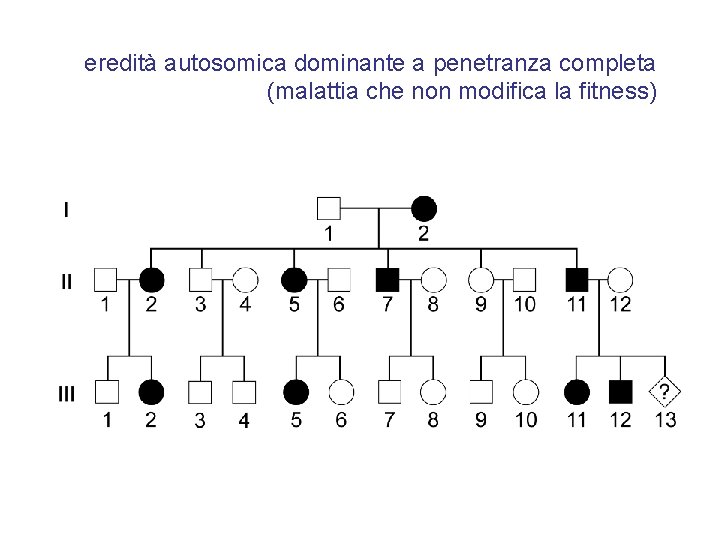
Malattie genetiche da mutazione in 1 allele Le mutazioni monoalleliche possono causare disordini a trasmissione dominante o recessiva legata all’X negli uomini • • • Se la malattia a trasmissione dominante è grave in età fertile e pertanto limita o annulla la capacità riproduttiva (bassa fitness), le mutazioni monoalleliche sono nuove e spesso distribuite in modo casuale Se la malattia dominante non è grave in età fertile e non limita in alcun modo la capacità riproduttiva (normale fitness), le mutazioni monoalleliche sono ereditate da un genitore e spesso si tramandano da molte generazioni Se la malattia è recessiva legata all’X ed è letale ha una vita media di tre generazioni, perché le donne trasmettono gli alleli mutati in eterozigosi e gli uomini li eliminano

eredità autosomica dominante a penetranza completa (malattia che non modifica la fitness)

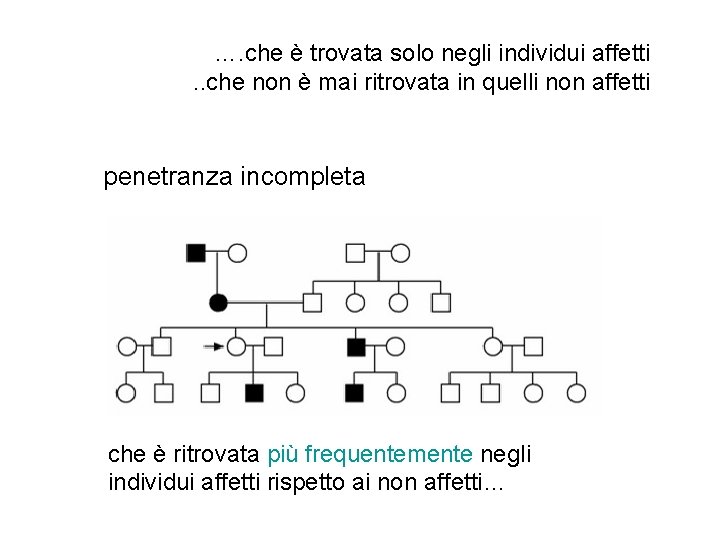
Cos’è una mutazione causativa? Una variazione della sequenza del DNA …. • . . che è trovata solo negli individui affetti • . . che non è mai ritrovata in quelli non affetti • . . che spiega il processo patologico • . . che, quando corretta per tempo, fa recuperare un fenotipo normale

…. che è trovata solo negli individui affetti. . che non è mai ritrovata in quelli non affetti penetranza incompleta che è ritrovata più frequentemente negli individui affetti rispetto ai non affetti…

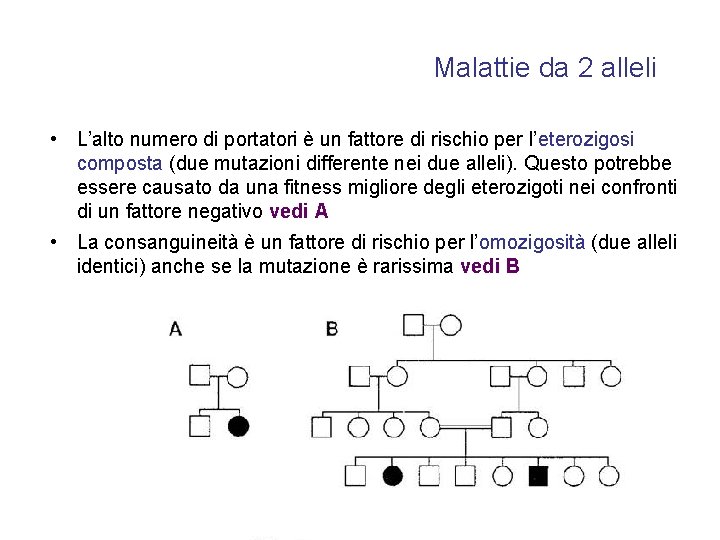
Malattie genetiche da mutazione in 2 alleli Le mutazioni bialleliche possono causare disordini a trasmissione autosomica recessiva • • • Se la malattia a trasmissione recessiva è grave in età fertile e limita o annulla la capacità riproduttiva (bassa fitness), le mutazioni non si estinguono comunque perché i portatori sani sono 10 -10. 000 volte più numerosi degli affetti Le mutazioni in genere si trasmettono da 100 -1000 generazioni, mentre le nuove mutazioni sono rare Solo se la malattia è biallelica le mutazioni hanno una firma etnica che caratterizza una località di origine e un fondatore comune eterozigote sano

Malattie da 2 alleli • L’alto numero di portatori è un fattore di rischio per l’eterozigosi composta (due mutazioni differente nei due alleli). Questo potrebbe essere causato da una fitness migliore degli eterozigoti nei confronti di un fattore negativo vedi A • La consanguineità è un fattore di rischio per l’omozigosità (due alleli identici) anche se la mutazione è rarissima vedi B


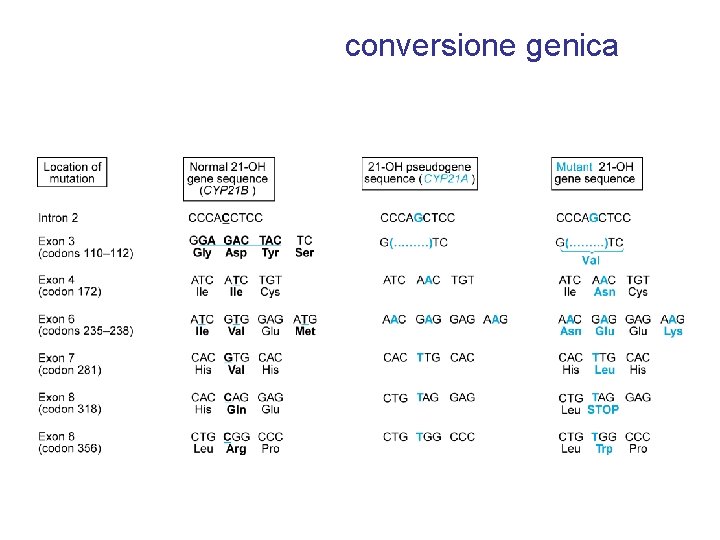
conversione genica
 Nigro & nigro pc
Nigro & nigro pc Mutazioni puntiformi
Mutazioni puntiformi Mutazioni puntiformi
Mutazioni puntiformi Potenziale elettrico formula
Potenziale elettrico formula Poem about team
Poem about team Mazzoldi nigro voci meccanica e termodinamica
Mazzoldi nigro voci meccanica e termodinamica Mutazioni genetiche
Mutazioni genetiche Poliploidia
Poliploidia Mutazioni genetiche
Mutazioni genetiche Mutazioni geniche
Mutazioni geniche Poliploidia
Poliploidia Poliglutamine
Poliglutamine Mutazioni geniche
Mutazioni geniche Inversione paracentrica
Inversione paracentrica Doenas
Doenas Patologia sociale
Patologia sociale Patologia vegetale molecolare
Patologia vegetale molecolare Hymenolepis nana morfologia
Hymenolepis nana morfologia Patologia
Patologia Posição ortostática
Posição ortostática Q es la patologia
Q es la patologia Patologia sperimentale unibo
Patologia sperimentale unibo Patologia
Patologia Tabela perímetro cefálico por idade
Tabela perímetro cefálico por idade Patologia
Patologia Acincia
Acincia Informe de patología estructural
Informe de patología estructural Imune
Imune Patologia usac
Patologia usac Patologia
Patologia Patologia hemolinfatica
Patologia hemolinfatica Patologia sociale
Patologia sociale Patologia
Patologia Corpusculi mallory
Corpusculi mallory Patologia
Patologia Brahiesofag
Brahiesofag Nablacion
Nablacion Robert s wright
Robert s wright Vincenzo liberatore
Vincenzo liberatore Vincenzo liberatore
Vincenzo liberatore Pars flaccida piccolo omento
Pars flaccida piccolo omento Vincenzo civerchio
Vincenzo civerchio Vincenzo vagnoni
Vincenzo vagnoni Vincenzo pisciuneri
Vincenzo pisciuneri Vincenzo vigna
Vincenzo vigna Vincenzo liberatore
Vincenzo liberatore Vincenzo quotes italian
Vincenzo quotes italian Itc benini melegnano
Itc benini melegnano Vincenzo pisciuneri
Vincenzo pisciuneri Grv to
Grv to Vincenzo lucchese
Vincenzo lucchese Docs. google. com/forms
Docs. google. com/forms Boracchi polimi
Boracchi polimi