MORT CELLULAIRE NECROSE APOPTOSE AUTOPHAGIE desousasophia inra fr





































- Slides: 57

MORT CELLULAIRE NECROSE APOPTOSE AUTOPHAGIE desousa@sophia. inra. fr

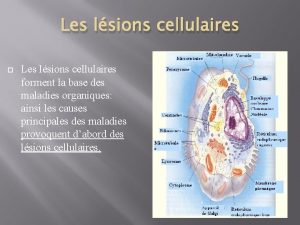
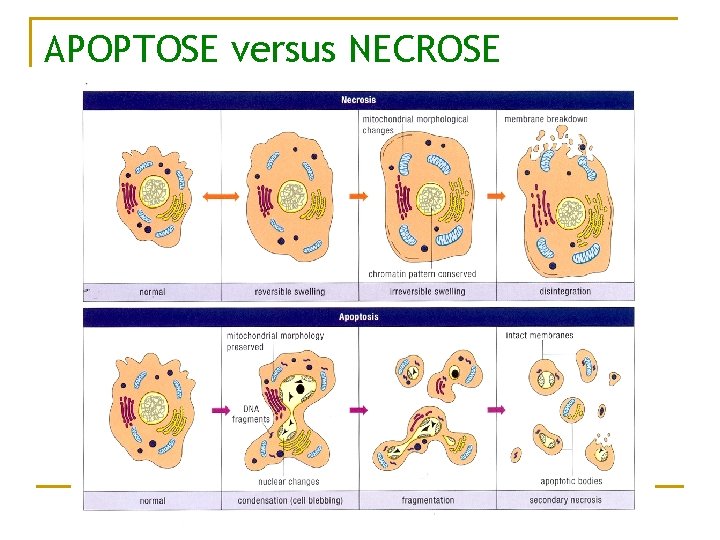
NECROSE n n La nécrose est une mort anormale de la cellule (cf. apoptose) Causes possibles : q q n Perte de l’homéostasie cellulaire Réduction de l’afflux sanguin Trop peu d’oxygène dans le sang Toxines, trauma, radiation, T°, etc. . Conséquences : q q Les cellules gonflent, éclatent et relarguent leurs contenus dans les espaces interstitiels Importante réaction inflammatoire

Fonctions cellulaires altérées q q q Dérégulation de la perméabilité membranaire et donc influence les mécanismes de transport Réduction du métabolisme cellulaire Plus de synthèse protéique Dommage aux lysosomes : fuite d’enzyme dans le cytoplasme Destruction des organelles cellulaires

MECANISMES MOLECULAIRES n L’absence d’oxygène entraine : q q q n Déplétion d’ATP Synthèse et dégradation des phospholipides Fuite de calcium de la mitochondrie Ischémie/Reperfusion q Création d’oxygène radicalaire O 2 - (anion superoxyde), OH- (radical hydroxyde), H 2 O 2 n n Molécules très réactive qui attaquent tous les constituants cellulaires Les capacités de détoxication de ces molécules sont très limités au niveau cellulaire

Atteinte cellulaire liée aux ROS n n n Anion Superoxyde formés par les P 450 et détoxication par les superoxyde dismutase Hydrogène peroxyde détoxication par : catalase et glutathion peroxydase Radical hydroxyl. OH initie une peroxydation lipidique et les atteintes à l’ADN

Comment les radiations tuent les cellules n n Radiolyse de l’eau H 20 donne. H et. OH Dommage aux membranes Dommage à l’ADN Les cellules non proliférative sont aussi tuées mais les doses doivent être plus importante

1ère Mise en évidence de l’apoptose Date du milieu du XIXème siècle puis remis au jour dans les années 30 pour vraiment être caractérisé en 1972 Décrit chez les organismes pluricellulaires: - Embryogenèse - Métamorphose n

Apoptose histoire n n 1842 : Carl Vogt remarque la disparition de noyaux dans certains tissus 1951 décrit la mort de certaines cellules dans les tissus embryonnaire 1964 Première utilisation du terme mort programmée 1972 Kerr Wyllie et Currie utilise le terme d’apoptose

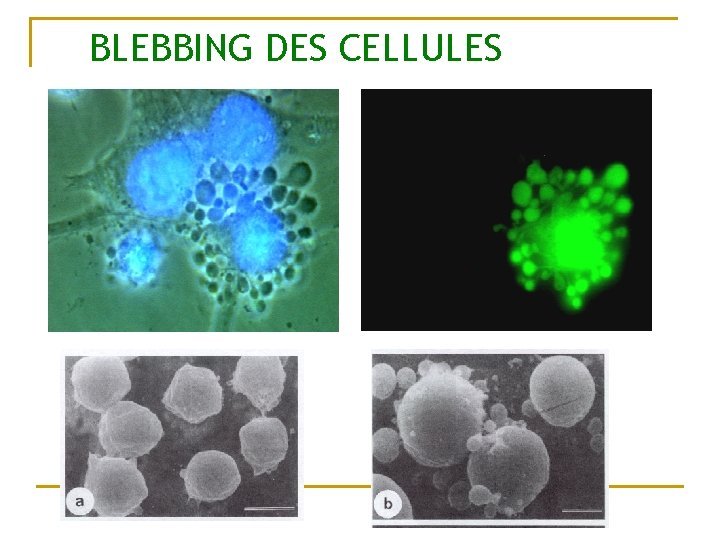
APOPTOSE n Caractéristiques d’une cellule en apoptose q q q n Condensation cellulaire Condensation de la chromatine Fragmentation de l’ADN « blebbing » de la membrane Exposition sur la membrane externe des phosphatidylserine Sécrétion de cytokines qui inhibe l’inflammation Ces caractéristiques sont régulés par des signaux

AUTOPHAGIE n n Sorte de « self » cannibalisme Manque de nutriment Digestion d’organelle intracellulaire Réarrangement de la membrane séquestration des composants dans des autophagosomes puis fusion avec lysozomes (dégradation enzymatique)

APOPTOSE versus NECROSE

FRAGMENTATION DE L’ADN

BLEBBING DES CELLULES

Pourquoi l’apoptose n n n Nombre de cellules dans l’organisme : 1014 Sans apoptose surplus de cellules, équilibre entre mort / prolifération Durée de vie d ’une cellule q q très variable selon l ’origine vie post-embryonnaire 200 types de cellules possédant toutes une durée de vie différente n n n Cell. Intestinales 1 semaines Erythrocyte 120 jours Cell. Hépatiques 1 ½ an Cell. Osseuses 25 -30 ans Neurones, Cell. Cardiaques, rétine, ne sont jamais remplacées Peau renouvelée tout les 23 jours

n Différenciation cellulaire q implique toute cellule soit au bon endroit au bon moment n Toutes cellules en trop doit disparaître n Période et vie cellulaire q q cellule en G 0 importance du réveil

Importance de l’apoptose n n n Homéostasie cellulaire, développement embryonnaire, synapse système immunitaire …(PCD : Programm cell death) Trop d’apoptose : maladie dégénératives Trop peu d’apoptose Cancer, maladies autoimmunes

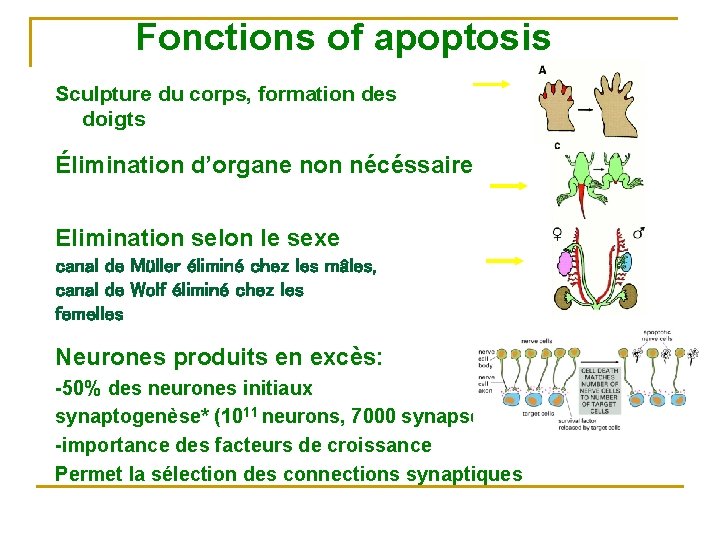
Fonctions of apoptosis Sculpture du corps, formation des doigts Élimination d’organe non nécéssaire Elimination selon le sexe canal de Müller éliminé chez les mâles, canal de Wolf éliminé chez les femelles Neurones produits en excès: -50% des neurones initiaux synaptogenèse* (1011 neurons, 7000 synapses) -importance des facteurs de croissance Permet la sélection des connections synaptiques

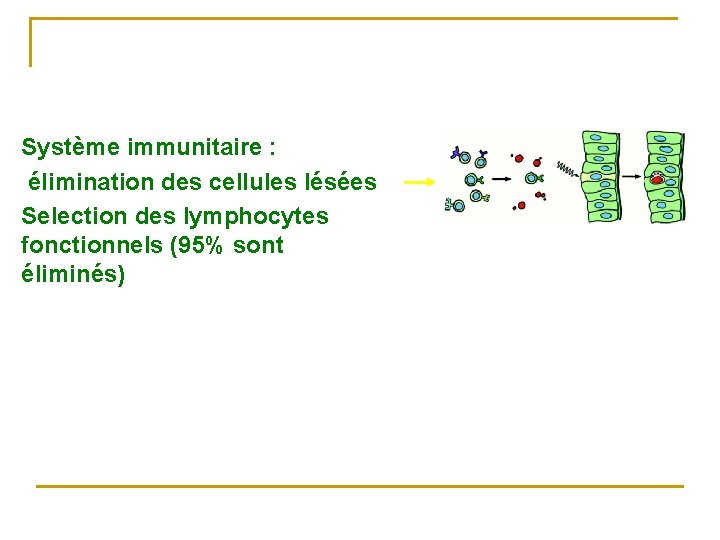
Système immunitaire : élimination des cellules lésées Selection des lymphocytes fonctionnels (95% sont éliminés)

Caenorhabditis elegans n n 1090 cellules somatiques 131 meurent par apoptose 116 de ces 131 appartiennent au système nerveux et à l’ectoderme 959 se développent chez l’adulte

MECANISMES GENERAUX n n Très conservés des espèces les plus élémentaires aux mammifères Premières études chez Caenorhabditis elegans q q 2 gènes ced-3 ced-4 «killer gene» 1 gènes ced-9 «survival gene»

Conservation des gènes Pro apop.


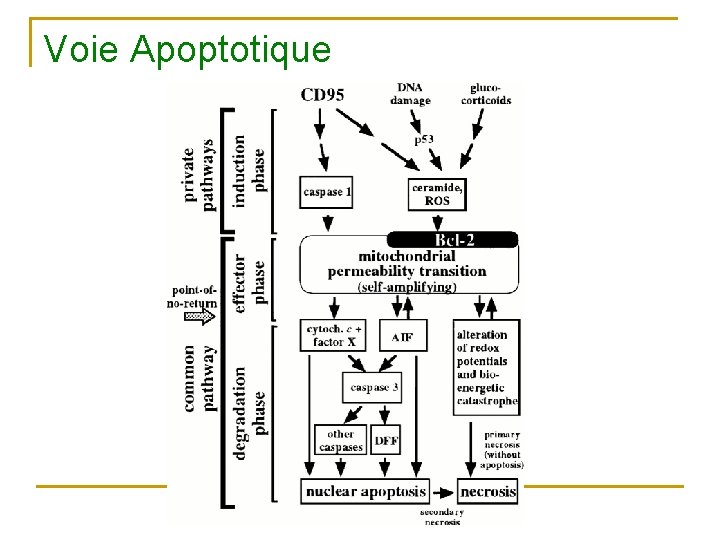
Voie Apoptotique

Différence entre les voies récepteur et mitochondriale n Mitochondriale q q q n Médié par le stress Synthèse de protéines 12 -24 heures Récepteur q q Pas de synthèse de prot Très rapide qqs heures

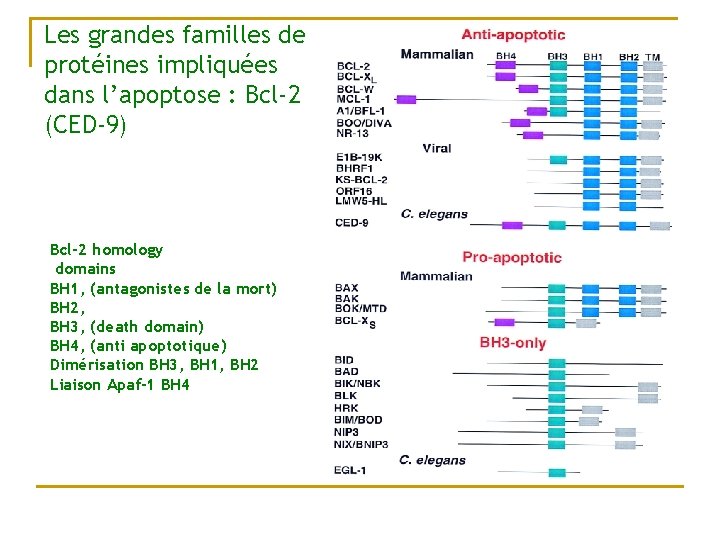
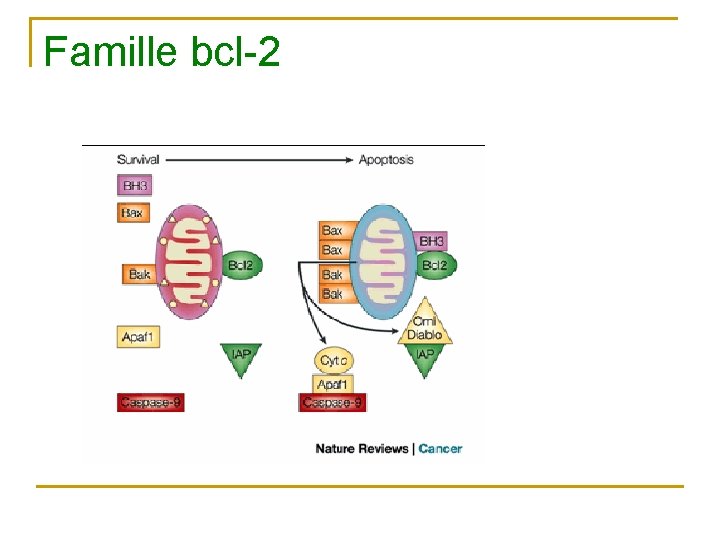
Les grandes familles de proteines impliquées dans l’apoptose : bcl-2 n n Homologue de ced-9 famille conservée, séquence homologue q n n BH domains (dimèrisation pour activation) Famille contenant des gènes pro et antiapoptotiques Homo et hétérodimère (balance entre mort et survie cellulaire)

Découverte de Bcl-2 n n En 1986 3 groupes indépendants clone ce que l’on appellera Bcl-2 oncogène En 1988 bcl-2 conduit a certain cancer en empêchant la mort cellulaire

Les grandes familles de protéines impliquées dans l’apoptose : Bcl-2 (CED-9) Bcl-2 homology domains BH 1, (antagonistes de la mort) BH 2, BH 3, (death domain) BH 4, (anti apoptotique) Dimérisation BH 3, BH 1, BH 2 Liaison Apaf-1 BH 4

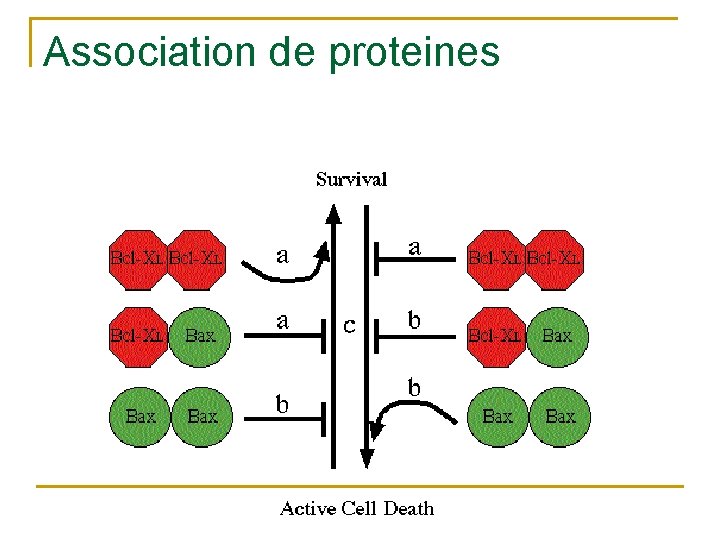
Association de proteines


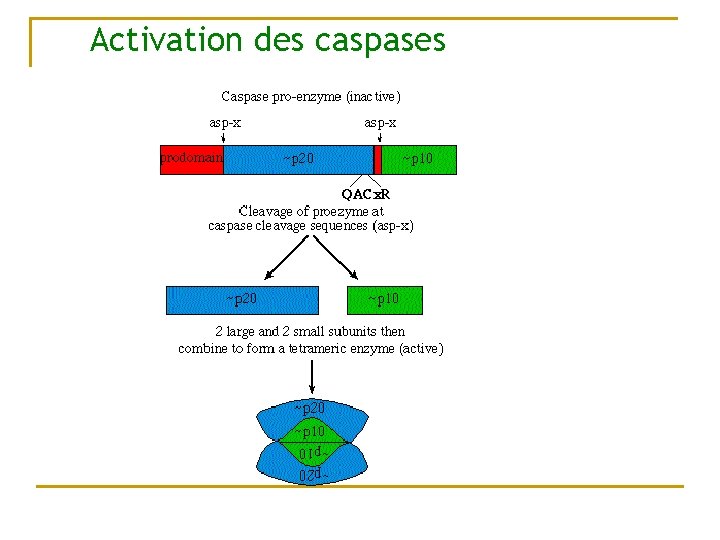
Les Caspase (CED-3) n n Cystéine protéase (17 membres) sont synthétisée à l’état de précurseur (QACXG) Comment les caspases tuent la cellule q n Destruction de protéines indispensable a la vie de la cellule Régulation des caspases : activation en cascade, IAP inhibiteur de caspase

Activation des caspases

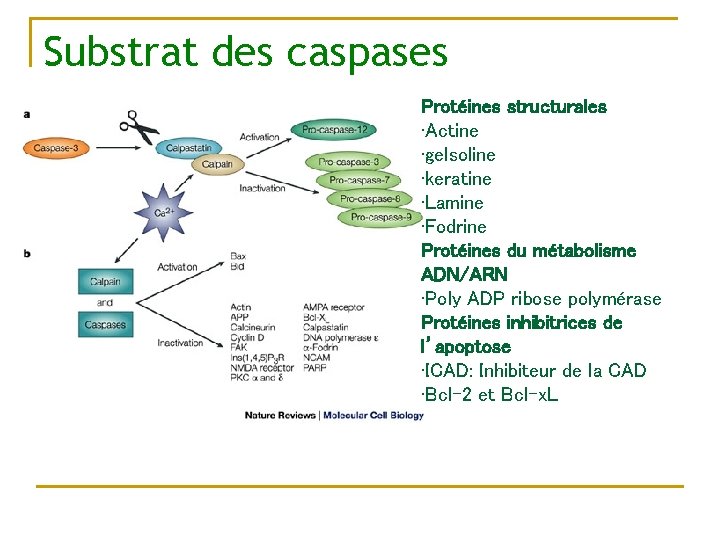
Proteines cibles des caspases FAK (focal adhésion kinase): l’inactivation de cette kinase pertube l’adhésion cellulaire, le détachement de la cellulae apoptotique de ses voisines. Lamines: Proteines de la membrane nucléaire, son clivage entraine la condensation du noyau. Proteines Structurales: actine, filaments intermédiares, etc. Destructuration cellulaire et formation des blebs. Endonuclease CAD: Fragmentation des chromosomes. CAD coupe L’AND en petit fragment. CAD est normalement inhibé. Les Caspases détruise la proteine inhibitrice et active CAD. Enzymes de réparation de l’ADN: pas de réparation si mort

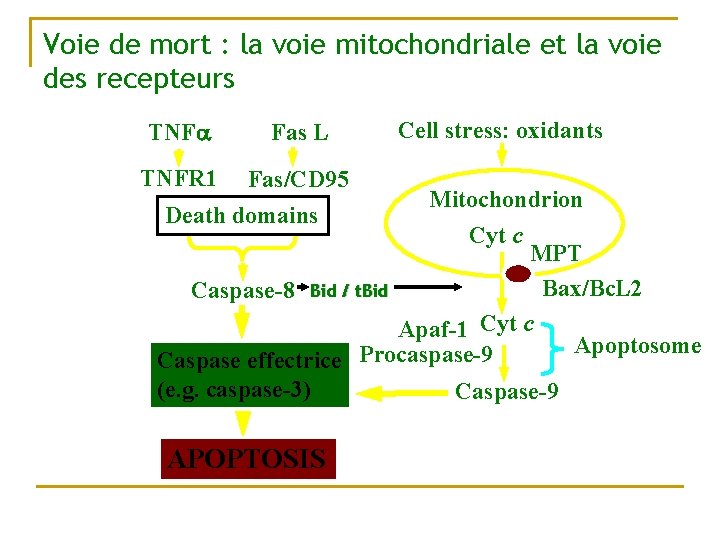
Voie de mort : la voie mitochondriale et la voie des recepteurs TNFa Fas L TNFR 1 Fas/CD 95 Death domains Cell stress: oxidants Mitochondrion Cyt c MPT Bax/Bc. L 2 Caspase-8 Bid / t. Bid Apaf-1 Cyt c Apoptosome Procaspase-9 Caspase effectrice (e. g. caspase-3) Caspase-9 APOPTOSIS

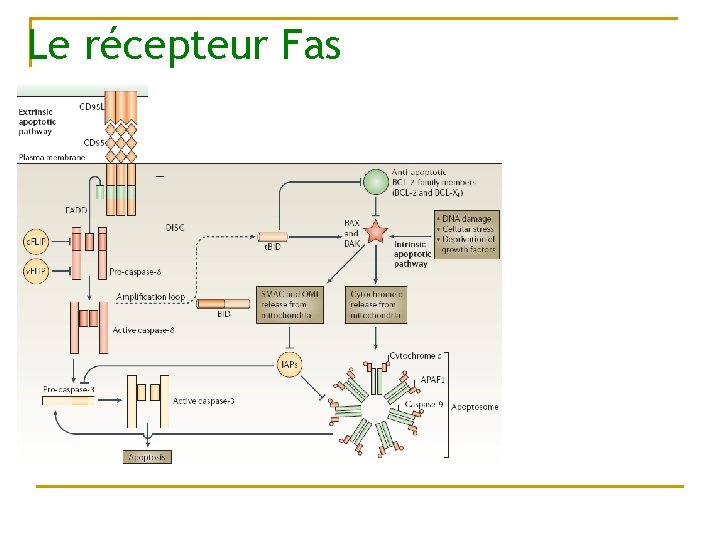
La voie de mort récepteur dépendant n n n Les récepteurs de mort : ils sont placés dans les membranes et détecte les signaux extracellulaire et initie rapidement la machinerie apoptotique Ils appartiennent à la famille des récepteurs au TNF car il contiennent un domaine extracellulaire riche en cystéine et dans le cytoplasme des séquences conservés appelés « Death Domain » Leurs ligands sont eux aussi très conservés

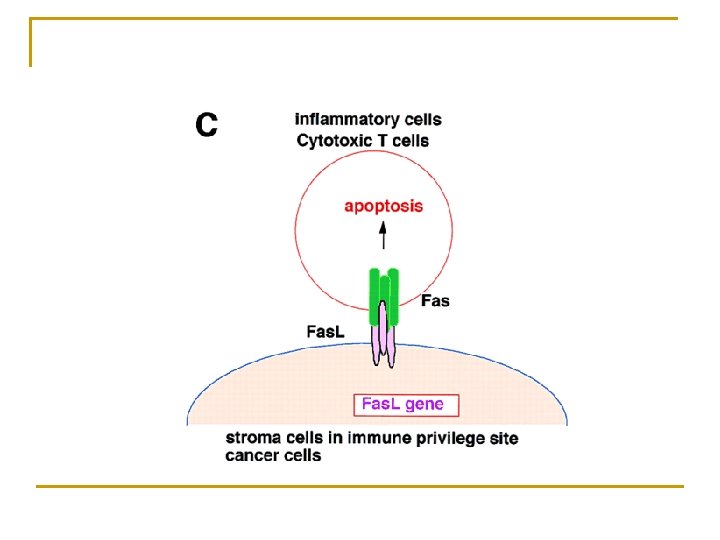
Le récepteur Fas

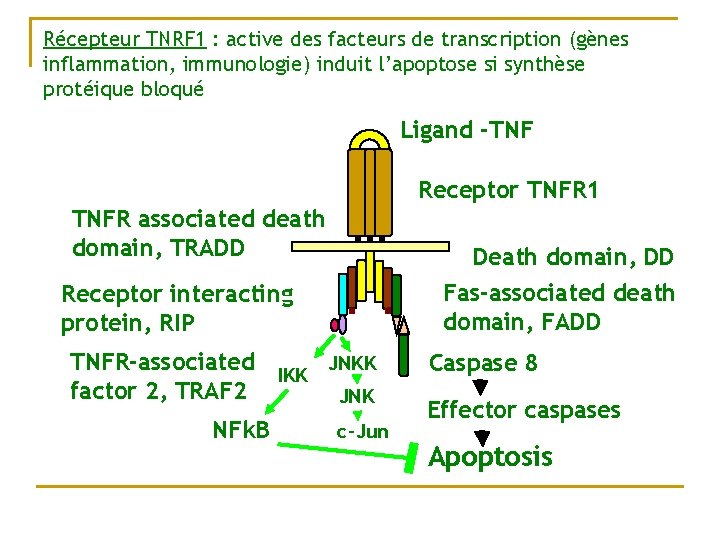
Récepteur TNRF 1 : active des facteurs de transcription (gènes inflammation, immunologie) induit l’apoptose si synthèse protéique bloqué Ligand -TNF Receptor TNFR 1 TNFR associated death domain, TRADD Death domain, DD Fas-associated death domain, FADD Receptor interacting protein, RIP JNKK TNFR-associated IKK factor 2, TRAF 2 JNK NFk. B c-Jun Caspase 8 Effector caspases Apoptosis

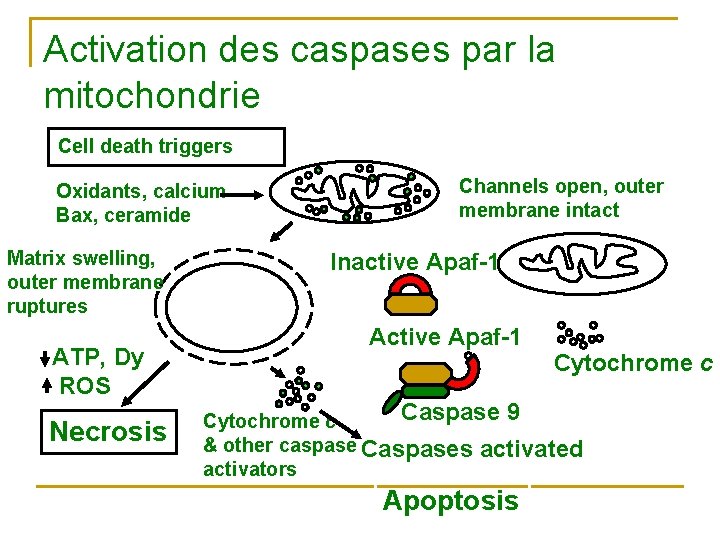
Mitochondrie et apoptose n n La mitochondrie est impliqué dans l’exécution et joue un rôle pivot 3 mécanismes q q q Dérégulation de la synthèse d’ATP Altération du statut REDOX Fuite de facteurs apoptogène qui vont activer des caspases

Famille bcl-2

Protéines activatrices des caspases n n n Cyt c va former l’apoptosome Procaspase 3 «dans certains tissus comme Apoptosis inducing factor AIF

Activation des caspases par la mitochondrie Cell death triggers Oxidants, calcium Bax, ceramide Matrix swelling, outer membrane ruptures ATP, Dy ROS Necrosis Channels open, outer membrane intact Inactive Apaf-1 Active Apaf-1 Cytochrome c Caspase 9 Cytochrome c & other caspase Caspases activated activators Apoptosis

Substrat des caspases Protéines structurales • Actine • gelsoline • keratine • Lamine • Fodrine Protéines du métabolisme ADN/ARN • Poly ADP ribose polymérase Protéines inhibitrices de l’apoptose • ICAD: Inhibiteur de la CAD • Bcl-2 et Bcl-x. L

Inhibitors of Apoptosis Proteins (IAPs) Identifié à partir de baculovirus n Consiste en diverses protéines : NAIP, MIAP XIAP, c. IAP 1, c. IAP 2, and survivine n Suppression de l’apoptose en empêchant l’activation des caspases et leur activités (caspase-3, -7, and 9) liaison spécifique n Expression of c. IAP 1/2 is stimulated by NF-κBmediated survival signals. n Negative regulators of IAPs: Smac/DIABLO, XAF 1, and OMI/HTRA 2 n

Apoptose et cycle cellulaire

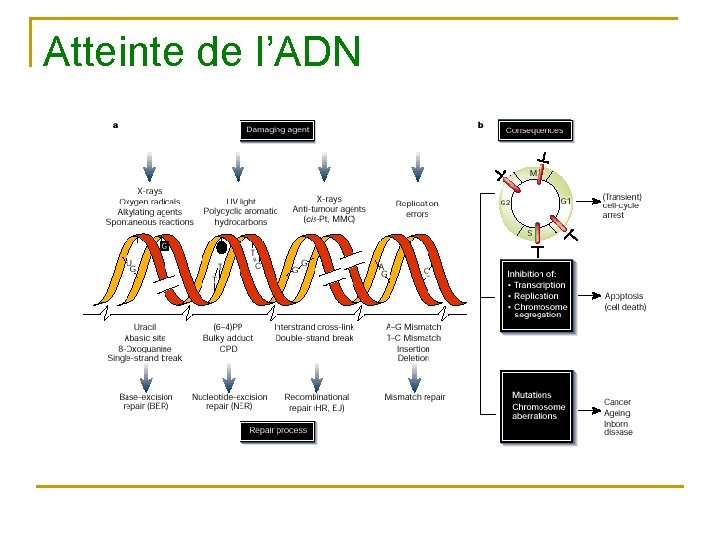
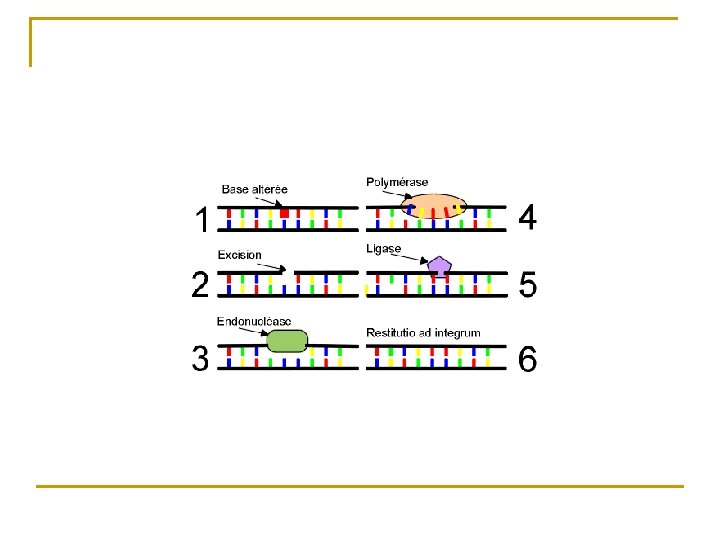
Atteinte de l’ADN


La proteine P 53 n n Dans les cellules normales (non stressé) la p 53 est rapidement dégradé après association avec son régulateur MDM 2. p 53 forme un complexe avec une proteine E 3 (ubiquitin ligase) et ainsi p 53 est guidé vers le proteasome. Dans les cellules normales p 53 est un régulateur positif de la prolifération cellulaire Dans les cellules « stressées » p 53 arrête la division par un phénomène apoptotique mitochondrial dépendant (Bax oligomérisation). Dans les cellules cancéreuses p 53 est un facteur de transcription (Puma, Noxa, p 53 AIP 1, Bax, Apa-1)

MDM 2 : Mouse Double Minute 2 n Le gène mdm 2 encode pour une protéine (zinc finger) qui inhibe p 53 pendant la phase de croissance dans une cellule normale q q n n Au niveau du noyau « bind » p 53 N-Terminal et masque son domaine de transactivation Au niveau du cytoplasme MDM 2 est responsable de l’ubiquitylisation et donc de la dégradation de p 53, MDM 2 est une cible de p 53 MDM 2 est une ubiquitin ligase (p 53 dégradation)

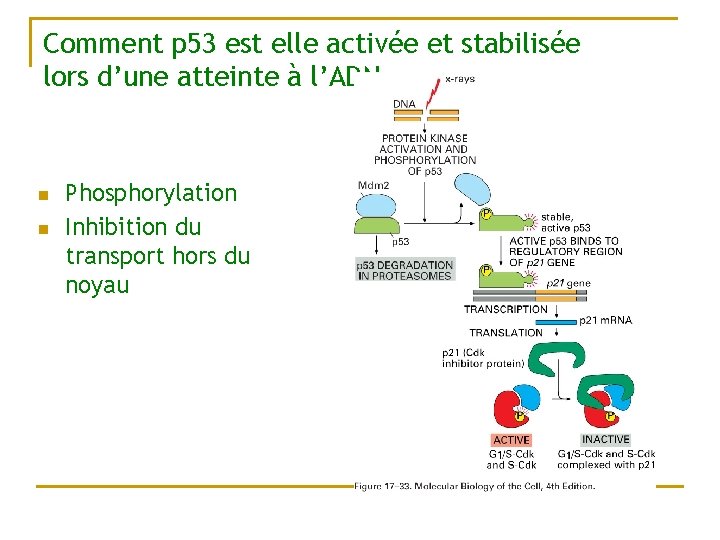
Comment p 53 est elle activée et stabilisée lors d’une atteinte à l’ADN n n Phosphorylation Inhibition du transport hors du noyau




Si p 53 est nécessaire pour supprimer des cellules altérées, que peut on attendre alors si p 53 est muté ? n Pas de blocage de la division cellulaire n Cancer

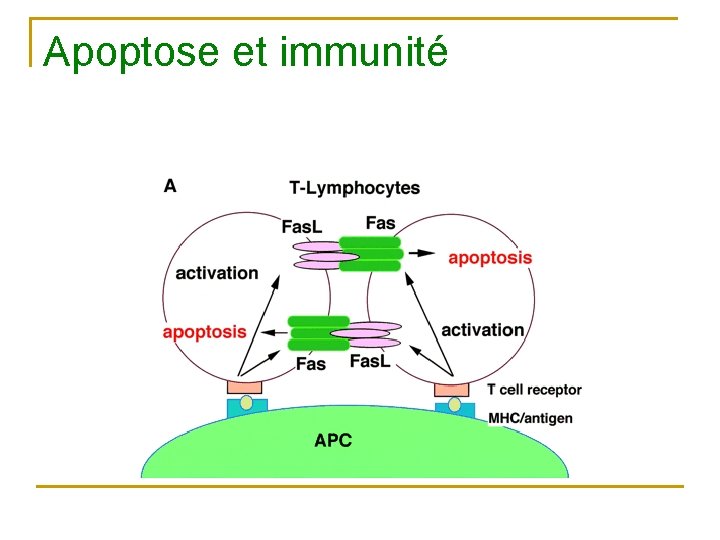
Apoptose et immunité



Foie : apoptose et nécrose n Ischémie : q q Baisse du niveau des IAPs No –Flow : affecte tous les hépatocytes Low Flow : affecte centrolobulaire vs périportal Ischémie reperfusion n Death recepteurs: * q q n n activation des cellules de Kupffler ; 1 -2 jours p. H > Nécrose (rapide) Aggrégation des récepteurs (Fas et TNFR) Sécrétion de Fas. L par lymphocyte cyto. (CTL) et le NK (hepatite alcoolique) Cholestase q Fas Médicementeuse q Acétaminophène : nécro/apoptose

n n n n • DED: death effector domain • CARD: caspase recruitment domain • CAD: caspase-activated deoxyribonuclease (CAD) • ICAD: CAD inhibitor • FADD: Fas-associated death domain • FLICE: Fas-like interleukin-1β converting enzyme • DISC: death inducing signaling complex • AIF: apoptosis inducing factor • Apaf-1: Apoptotic protease-activating factor-1 • Apoptosome: Cyt. C Apaf-1, d. ATP/ATP and pro-caspase 9 • Smac: second mitochondria-derived activator of caspase. • DIABLO: direct IAP binding protein with low p. I Bcl-2 proteins : B cell leukemia/lymphoma 2 Caspases : cysteine-dependent asparate-specific proteases; IAPs: Inhibitor of apoptosis
 Necrose e apoptose
Necrose e apoptose Apoptose exemplos
Apoptose exemplos Comparaison entre cellule animale et végétale tableau
Comparaison entre cellule animale et végétale tableau Oxydoreduction
Oxydoreduction Membrane cellulaire
Membrane cellulaire Psm culture cellulaire
Psm culture cellulaire Signalisation cellulaire
Signalisation cellulaire Christian dupraz inrae
Christian dupraz inrae Dr lawrence inra
Dr lawrence inra Inra
Inra Necrose tubulaire aigue
Necrose tubulaire aigue Tipos necrose
Tipos necrose Polype pulpaire définition
Polype pulpaire définition Seriacion troponina
Seriacion troponina La nécrose pulpaire
La nécrose pulpaire Pycnose reversible
Pycnose reversible Necrose liquefeita
Necrose liquefeita Maxalexis mort
Maxalexis mort La peine de mort argument contre
La peine de mort argument contre La mort des oiseaux
La mort des oiseaux Felix nussbaum autoportrait au passeport juif
Felix nussbaum autoportrait au passeport juif Solomon linda le lion est mort ce soir
Solomon linda le lion est mort ce soir Pierre bachelet décès
Pierre bachelet décès Heros de la mort aux trousses
Heros de la mort aux trousses Nestrien
Nestrien Comment est mort pouchkine
Comment est mort pouchkine Mort janklow
Mort janklow Saule pleureur mort
Saule pleureur mort Doris lussier texte sur la mort
Doris lussier texte sur la mort X360ca
X360ca Chapitre xxiii le dernier jour d'un condamné
Chapitre xxiii le dernier jour d'un condamné Tatiana schlossberg rose schlossberg
Tatiana schlossberg rose schlossberg Orphée et eurydice corot
Orphée et eurydice corot Quand est né et mort euclide
Quand est né et mort euclide Radu cinamar
Radu cinamar Maurice tillet mort
Maurice tillet mort Pam mort
Pam mort Vincenzo camuccini la mort de césar
Vincenzo camuccini la mort de césar Anverten
Anverten Aripi membranoase
Aripi membranoase Le triomphe de la mort
Le triomphe de la mort Couchée avec mes soeurs j'ai la tete rouge reponse
Couchée avec mes soeurs j'ai la tete rouge reponse Slidetodoc
Slidetodoc Words with the root vit
Words with the root vit La mort nest rien
La mort nest rien