Modell Hairpin gir RNA pol pause og induserer


























- Slides: 59

Modell • Hairpin gir RNA pol pause og induserer konformasjonsendring i enzymet • RNA henger nå bare svakt i templat via d. A-r. U og dissosierer – Forutsagt effekt av GTP til ITP (svekket terminering) • Flere andre forhold bidrar • Kontekst, NTP, supercoil osv

Assistert terminering - rho-avhengig • Noen termineringsseter krever en hjelpefaktor (rho -faktor) • Rho = Hexamer NTPase som katalyserer opptvinning (unwinding) • Modell – Rho binder RNA oppstrøms via et spesifikt motiv, migrerer mot polymerasen, opptvinning, frigjøring av transkript

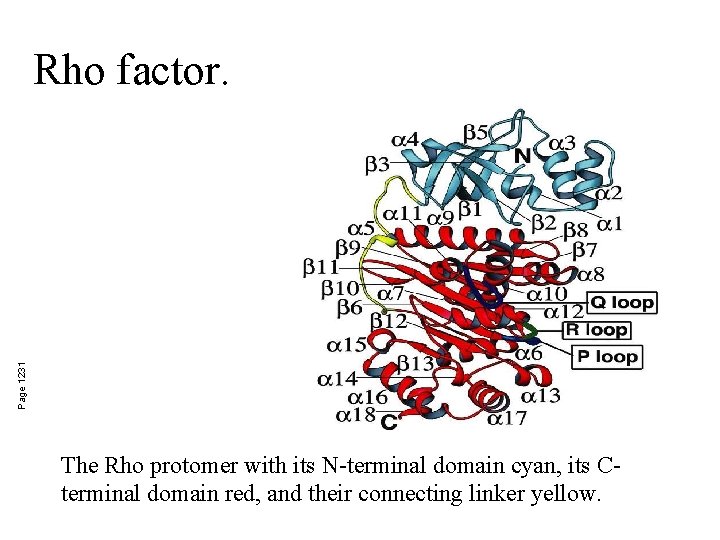
Page 1231 Rho factor. The Rho protomer with its N-terminal domain cyan, its Cterminal domain red, and their connecting linker yellow.

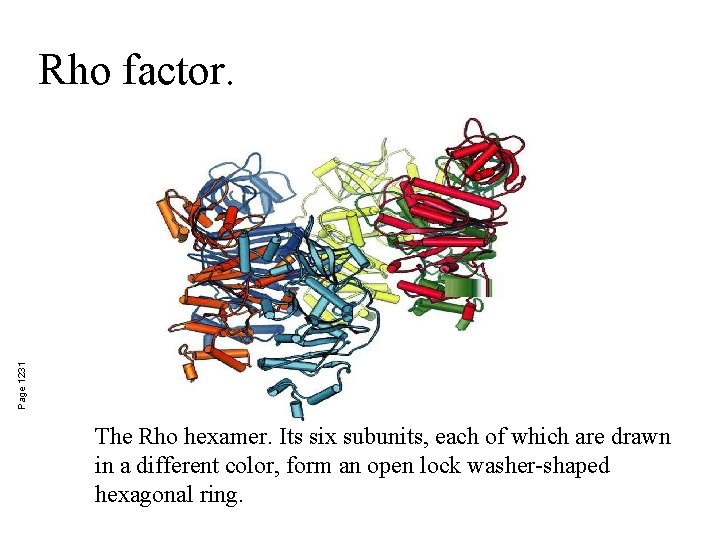
Page 1231 Rho factor. The Rho hexamer. Its six subunits, each of which are drawn in a different color, form an open lock washer-shaped hexagonal ring.

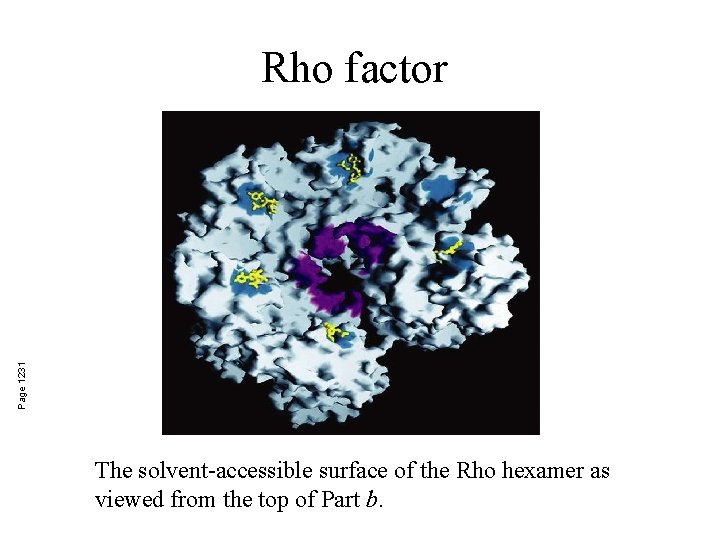
Page 1231 Rho factor The solvent-accessible surface of the Rho hexamer as viewed from the top of Part b.

Hvordan regulere transkripsjon? • Behov for regulering: – Nivå: Noen m. RNA trenges i store mengder, andre i små – Timing: Forskjellige m. RNA trenges til ulike tidspunkt – Respons: Noen m. RNA trenges bare i visse miljø (nærvær av ulike næringsstoffer) • Hvordan oppnås ulike transkripsjonsnivå?

Rask endring i prokaryoter • Koblet translasjon /transkripsjon gir respons innen minutter – Translasjon av m. RNA før transkripsjon er ferdig – m. RNAs levetid også kort (1 -3 min)

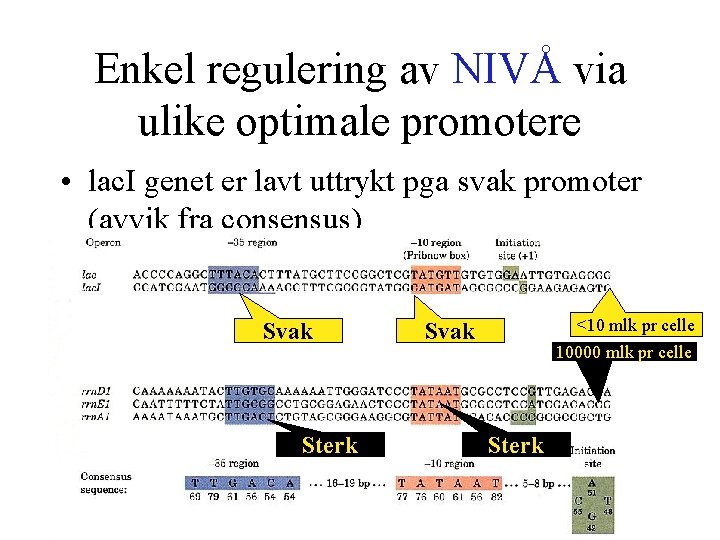
Enkel regulering av NIVÅ via ulike optimale promotere • lac. I genet er lavt uttrykt pga svak promoter (avvik fra consensus) Svak Sterk <10 mlk pr celle Svak 10000 mlk pr celle Sterk

Regulering av TIMING - serie av sigma-faktorer • Utvikling/differensiering = tidsmessig ordnet ekspresjon av gen-programmer • Fag-infeksjon - enkel modell for slik timing – Early - middle - late genes • B. subtilis infisert av fagen SP 01 – Early genes - via vertens RNAP/s - normale promotere – Et tidlig produkt = sgp 28 en ny s med endret promoterpreferanse – Middle genes - via vertens RNAP/sgp 28 - egne promotere – Late genes - via vertens RNAP/sgp 33/34 - egne promotere

Regulering av RESPONS - via repressorer (og aktivatorer) • Prokaryot transkripsjons-regulering via tre elementer: • Promotere gjenkjennes av RNA polymerasen • Operatorer gjenkjennes av repressorer • Positive kontrollelementer gjenkjennes av aktivatorer

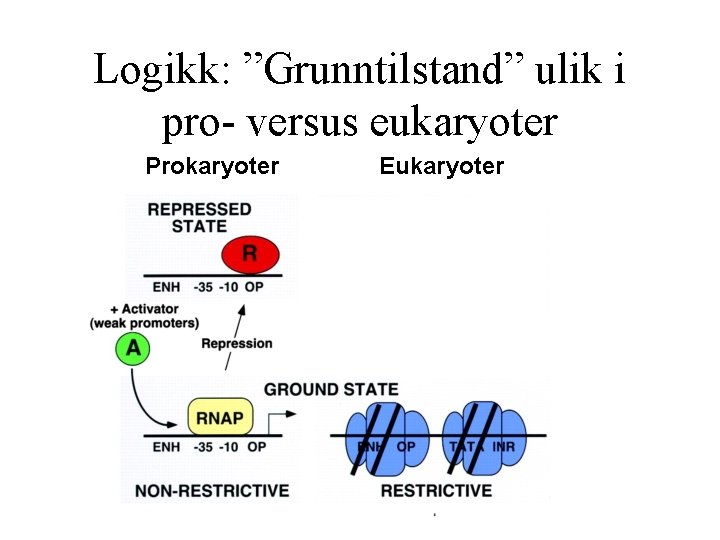
Logikk: ”Grunntilstand” ulik i pro- versus eukaryoter • Grunntilstand – Aktiviteten av en promoter in vivo i fravær av regulatoriske sekvenser og aktivatorer/repressorer • Prokaryoter: grunntilstand = åpen – Ingen begrensning på RNA polymerasens tilgang til promotere – Repressorer blokkerer tilgang – Aktivatorer kun nødvendige på visse svake promotere • Eukaryoter: grunntilstand = lukket (pga kromatin) – En sterk core-promoter alene er inaktiv i eukaryoter – De fleste eukaryote gener krever aktivatorer

Logikk: ”Grunntilstand” ulik i pro- versus eukaryoter Prokaryoter Eukaryoter

Lac operon Eksempel på repressorregulering av promotertilgang

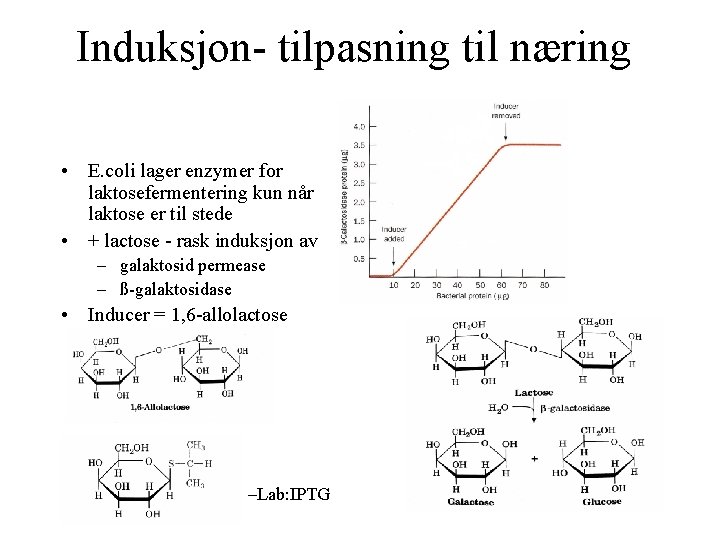
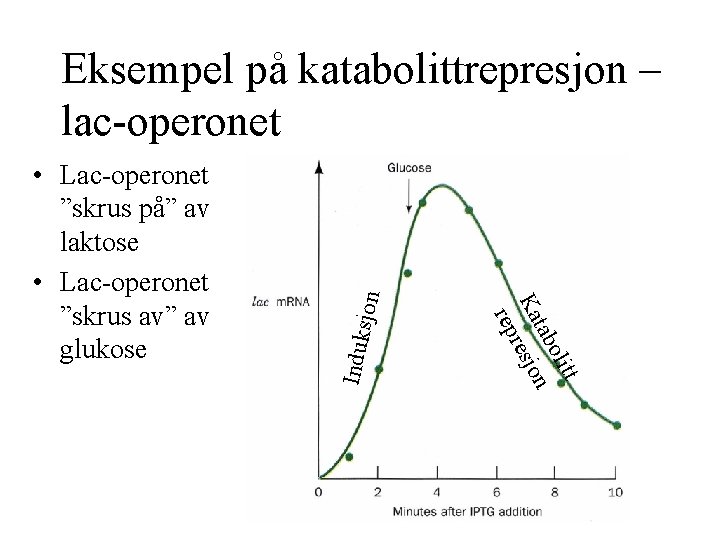
Induksjon- tilpasning til næring • E. coli lager enzymer for laktosefermentering kun når laktose er til stede • + lactose - rask induksjon av – galaktosid permease – ß-galaktosidase • Inducer = 1, 6 -allolactose –Lab: IPTG

Genetisk kartlegging • Operon • Strukturelle gener – Proteinkodende • Operator – Bindingssekvens for regulator • Promoter – Bindingssekvens for RNA polymerase

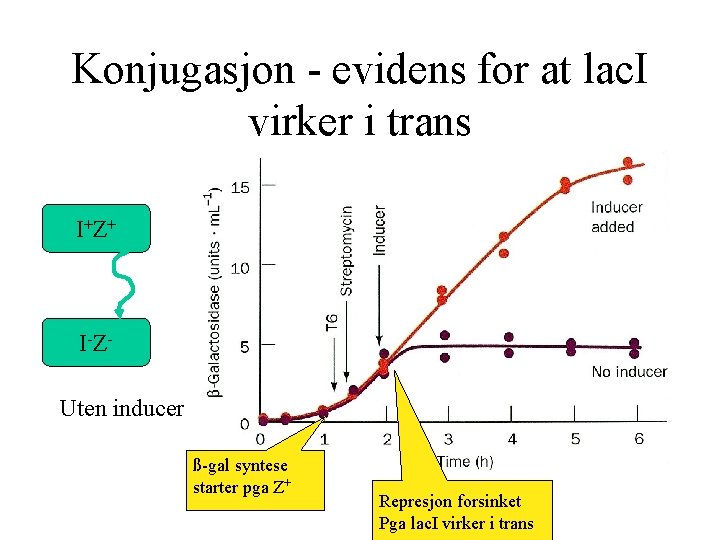
Evidens for repressor • Konstitutive mutasjoner – lac-operonet aktivt i fravær av inducer • Mutasjon mappet til lac. I genet – lac. I ligger utenfor operonet • lac. I virker i trans via kodet protein-produkt (lac repressor) • Oc mutasjoner samme effekt men virker i cis (operator mutasjon)

Konjugasjon - evidens for at lac. I virker i trans I+ Z + I -Z Uten inducer ß-gal syntese starter pga Z+ Represjon forsinket Pga lac. I virker i trans

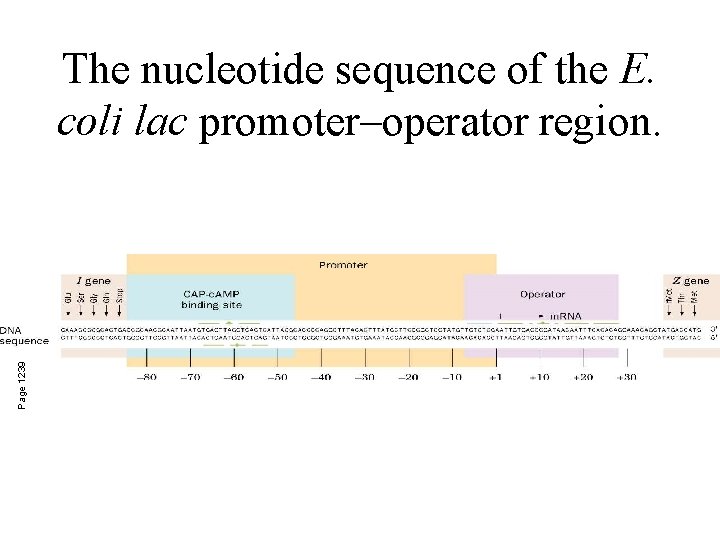
Page 1239 The nucleotide sequence of the E. coli lac promoter–operator region.

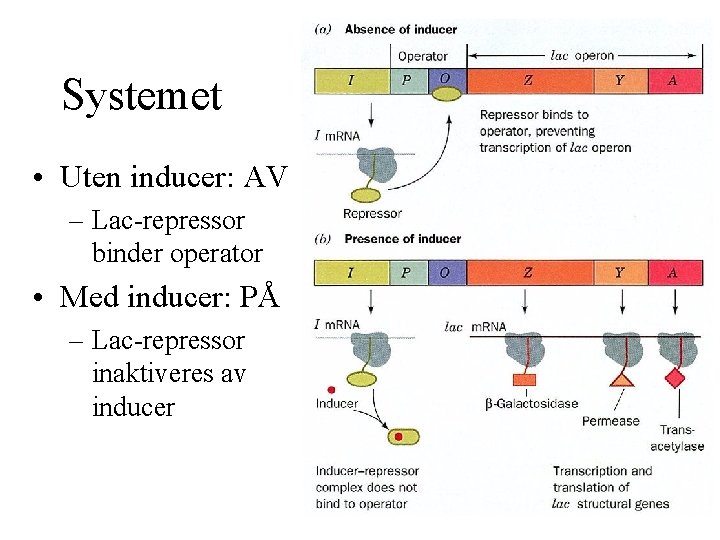
Systemet • Uten inducer: AV – Lac-repressor binder operator • Med inducer: PÅ – Lac-repressor inaktiveres av inducer

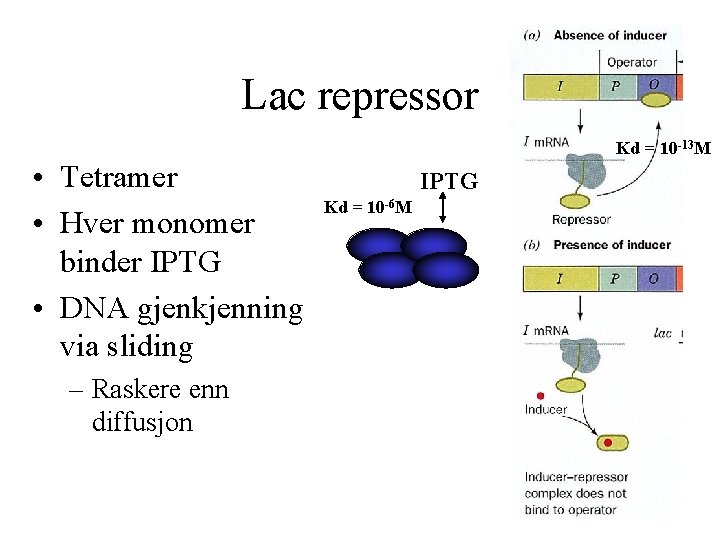
Lac repressor Kd = 10 -13 M • Tetramer • Hver monomer binder IPTG • DNA gjenkjenning via sliding – Raskere enn diffusjon IPTG Kd = 10 -6 M

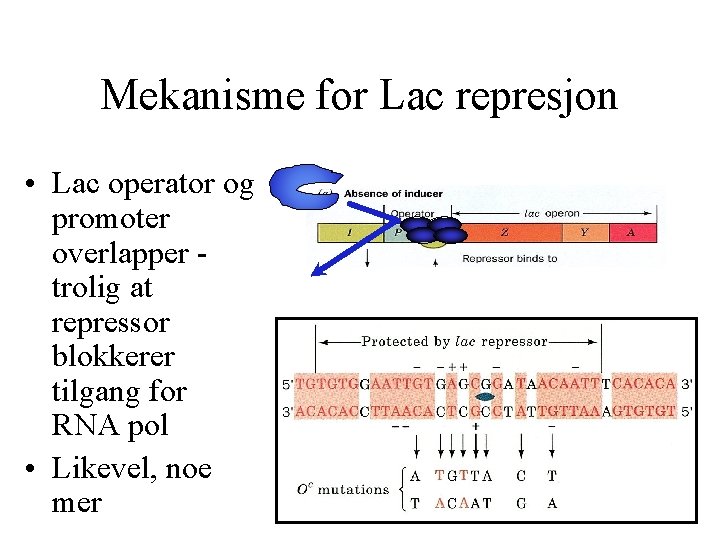
Mekanisme for Lac represjon • Lac operator og promoter overlapper trolig at repressor blokkerer tilgang for RNA pol • Likevel, noe mer

Page 1248 X-Ray structure of the lac repressor subunit.

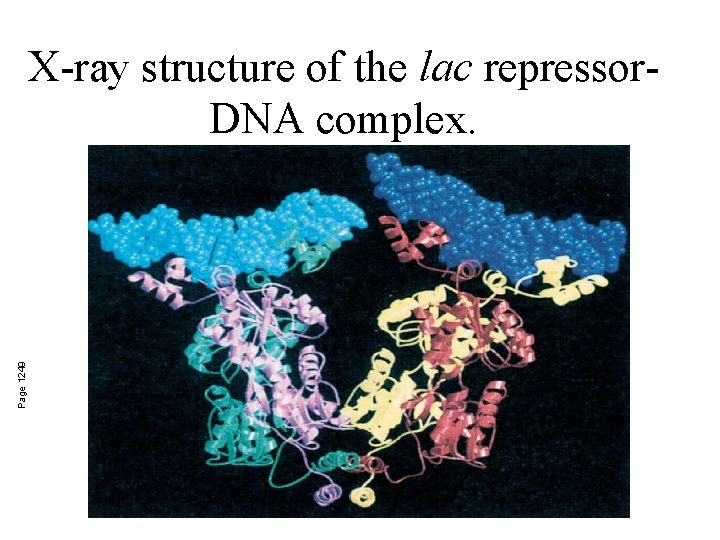
Page 1249 X-ray structure of the lac repressor. DNA complex.

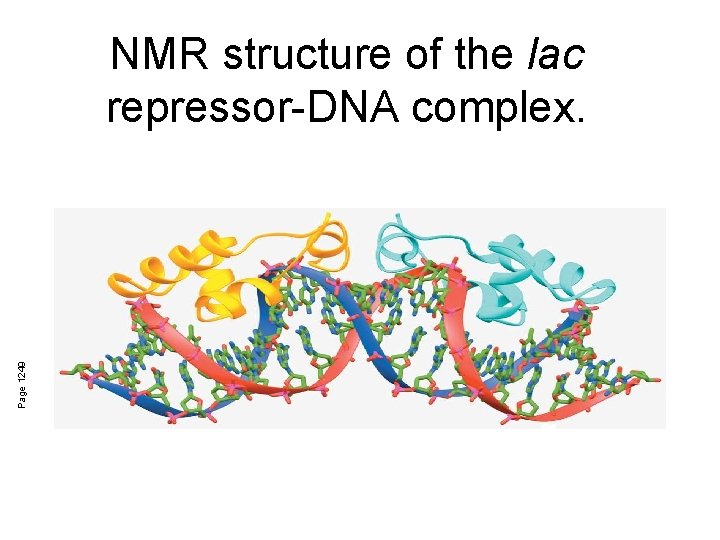
Page 1249 NMR structure of the lac repressor-DNA complex.

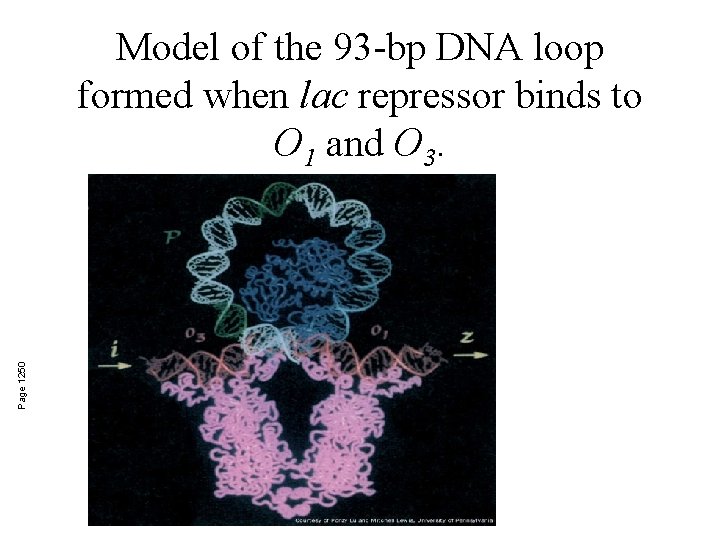
Page 1250 Model of the 93 -bp DNA loop formed when lac repressor binds to O 1 and O 3.

Katabolittrepresjon Eksempel på aktivering av gener via CAP-c. AMP komplekset

Glukoses forrang • Glukose foretrukket metabolitt • Katabolitt represjon – nærvær av glukose hindrer uttrykk av gener nødvendige for annen fermentering (laktose, arabinose, galaktose) • Hvordan senses høy/lav glukose? via c. AMP! – Glukose senker c. AMP-nivået

Induk tt oli tab n Ka resjo rep • Lac-operonet ”skrus på” av laktose • Lac-operonet ”skrus av” av glukose sjon Eksempel på katabolittrepresjon – lac-operonet

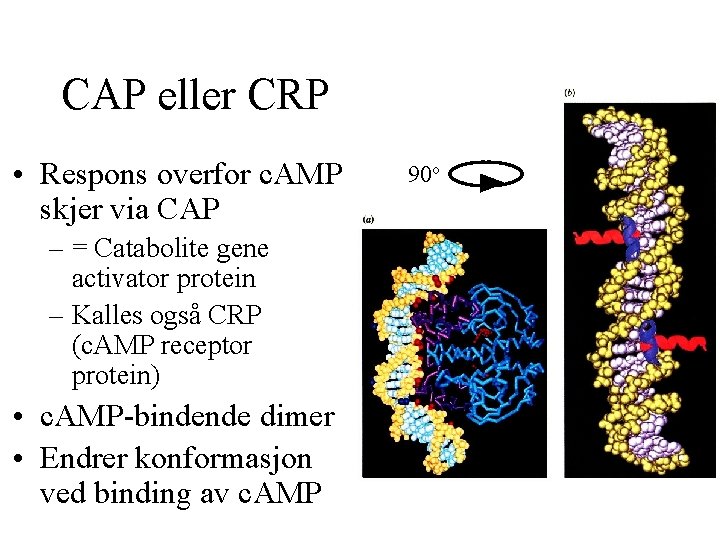
CAP eller CRP • Respons overfor c. AMP skjer via CAP – = Catabolite gene activator protein – Kalles også CRP (c. AMP receptor protein) • c. AMP-bindende dimer • Endrer konformasjon ved binding av c. AMP 90 o

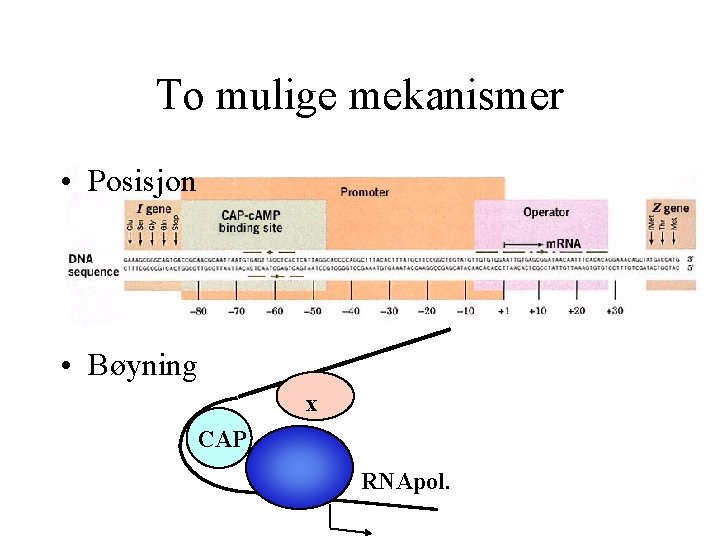
Mekanisme for aktivering? • CAP kan kontakte RNA pol direkte – CAP binder oppstrøms initieringskompleks – De to bindes i løsning • CAP endrer DNA-konformasjonen – 90 o indusert bøyning - endrede relative posisjoner – Major groove lukkes, minor åpnes – CAPs effekt gjenskapes av DNA-sekvenser som spontant bøyes

To mulige mekanismer • Posisjon • Bøyning x CAP RNApol.

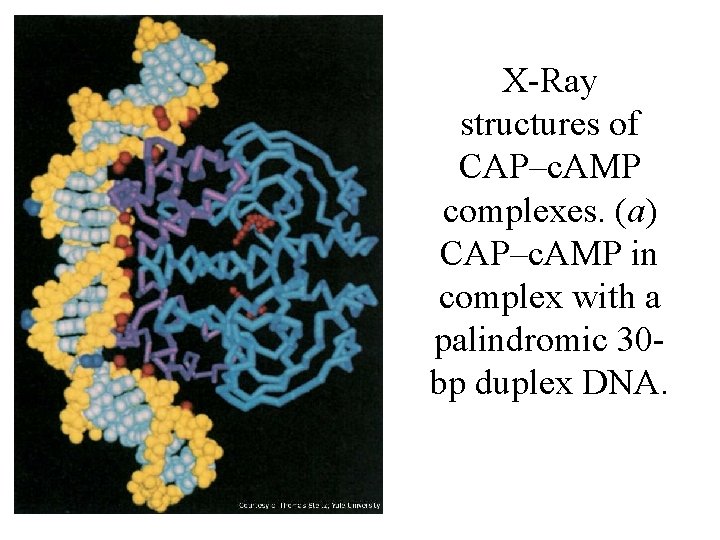
Page 1241 X-Ray structures of CAP–c. AMP complexes. (a) CAP–c. AMP in complex with a palindromic 30 bp duplex DNA.

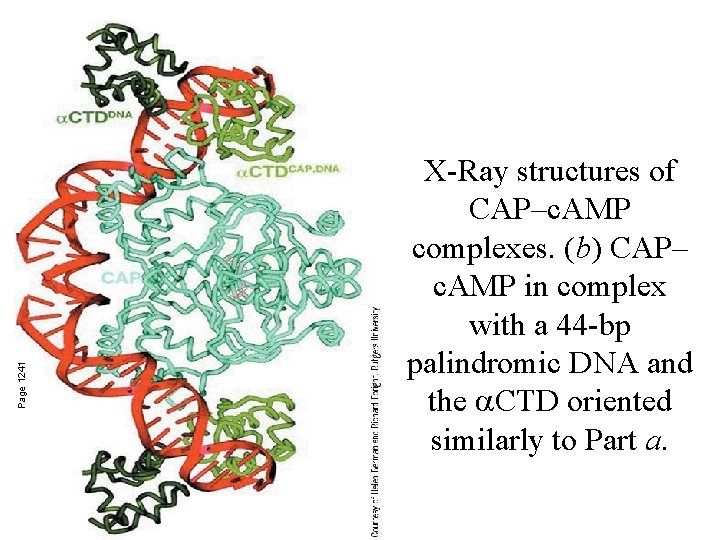
Page 1241 X-Ray structures of CAP–c. AMP complexes. (b) CAP– c. AMP in complex with a 44 -bp palindromic DNA and the CTD oriented similarly to Part a.

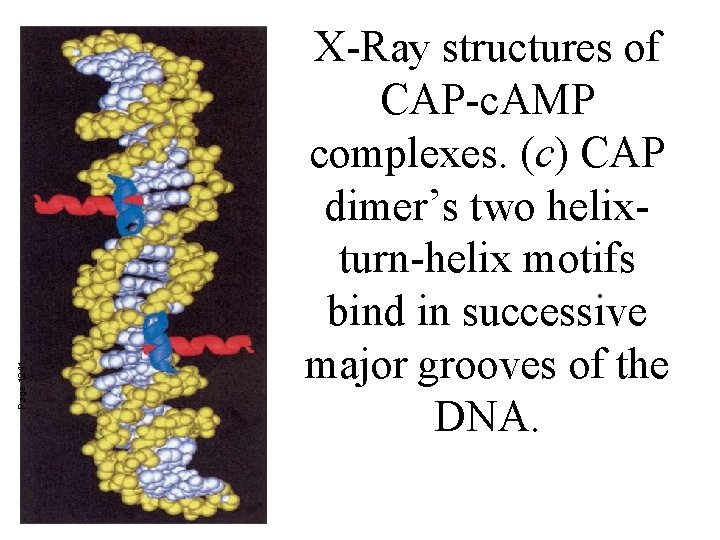
Page 1241 X-Ray structures of CAP-c. AMP complexes. (c) CAP dimer’s two helixturn-helix motifs bind in successive major grooves of the DNA.

Sekvens-spesifikk DNA-binding Prinsipper og eksempler

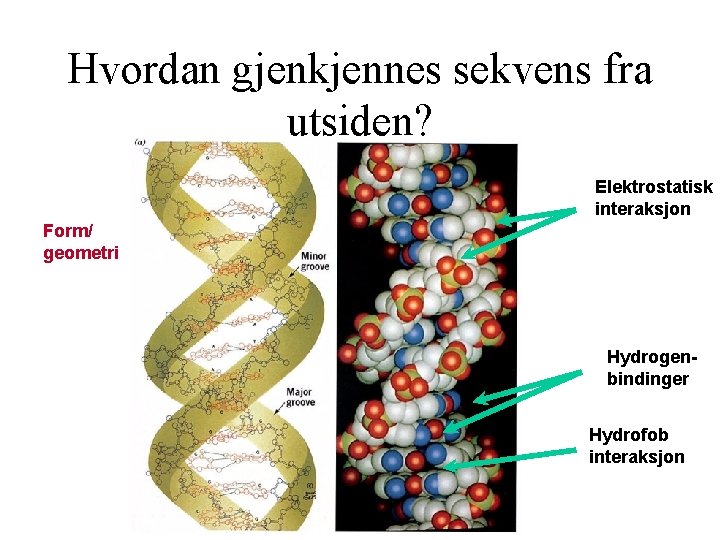
Hvordan gjenkjennes sekvens fra utsiden? Elektrostatisk interaksjon Form/ geometri Hydrogenbindinger Hydrofob interaksjon

Komplementære former • Dimensjonen til heliks passer dimensjonene i major groove av B-DNA • Sidegruppene peker utover og er gunstig plassert for dannelse av hydrogenbindinger

Gjenkjenning via hydrogenbinding • Hydrogenbinding er sentral for spesifikk gjenkjenning • Hydrogenbindingspotensialet er ikke uttømt i dupleks DNA, ledige seter peker ut mot major groove D A A

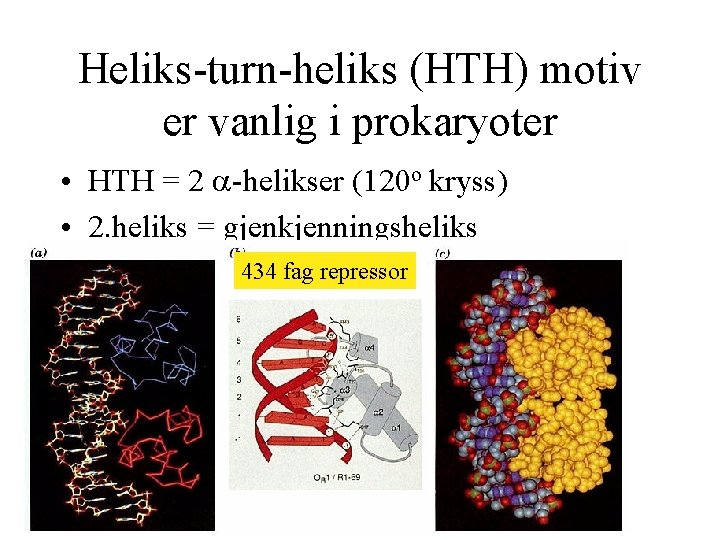
Heliks-turn-heliks (HTH) motiv er vanlig i prokaryoter • HTH = 2 -helikser (120 o kryss) • 2. heliks = gjenkjenningsheliks 434 fag repressor

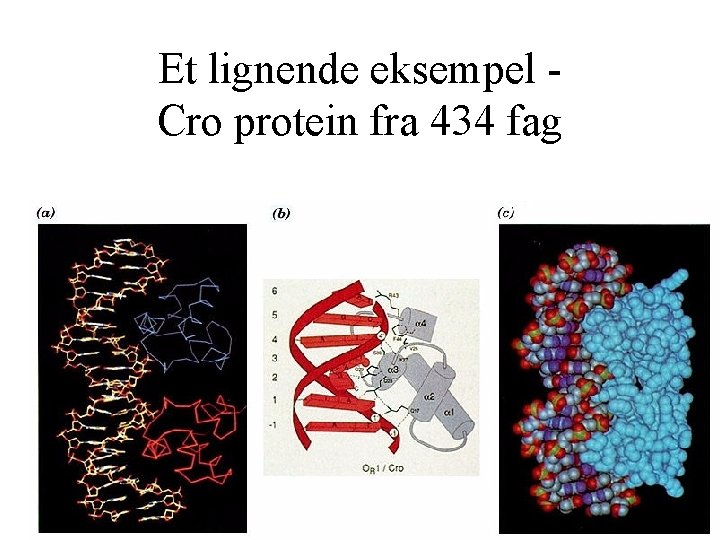
Et lignende eksempel Cro protein fra 434 fag

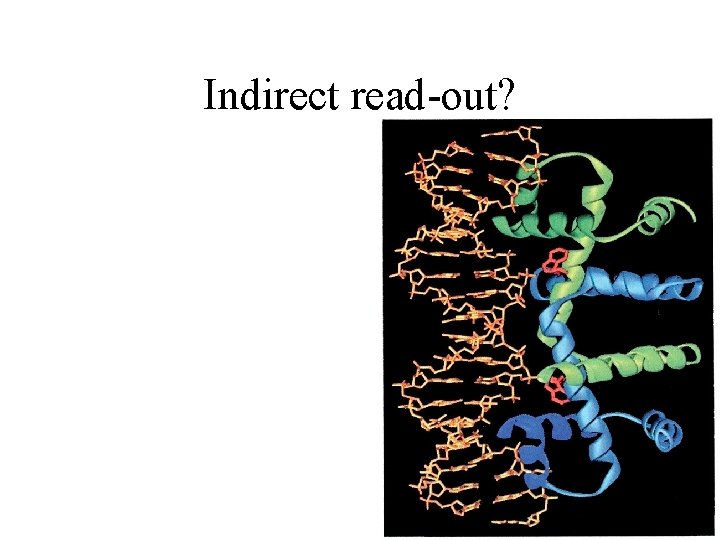
Indirect read-out?

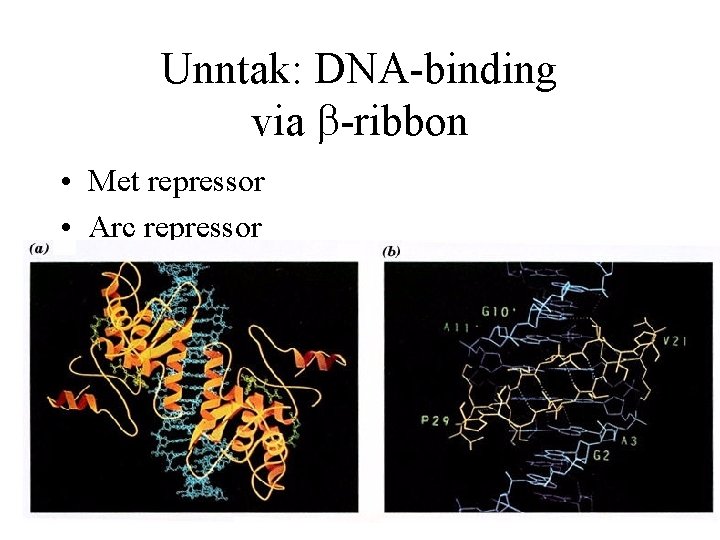
Unntak: DNA-binding via b-ribbon • Met repressor • Arc repressor

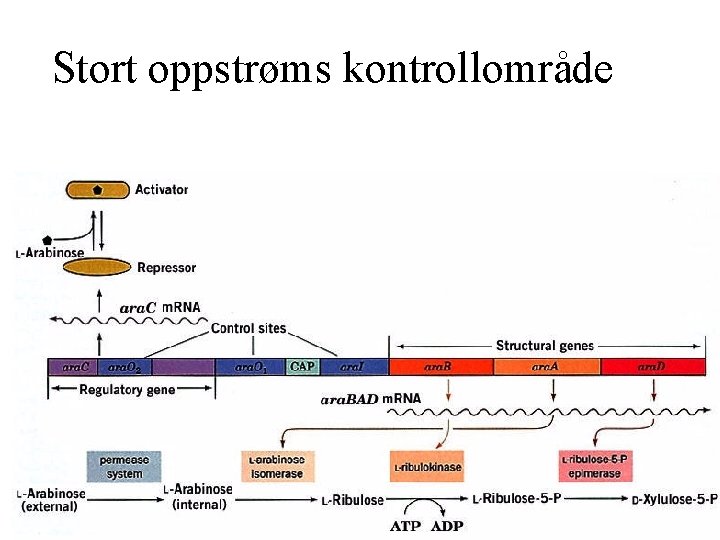
Arabinose-operonet

Stort oppstrøms kontrollområde

Kontroll via looping

Page 1247 X-Ray structure of E. coli Ara. C in complex with L-arabinose.

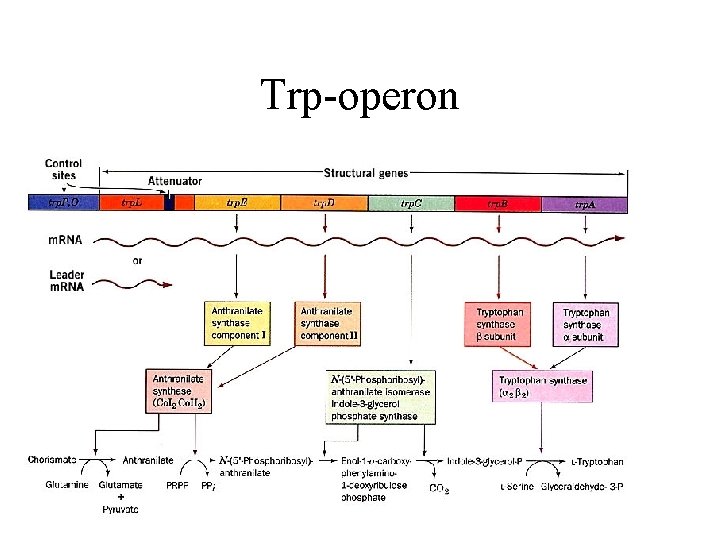
Trp operon Eksempel på regulering via både initiering og terminering

1. nivå: repressor + corepressor • Trp repressor binder tryptofan og styrkes som repressor – Logikk: når Trp tilgjengelig, reduseres syntese • Trp. R + Trp binder operator og inhiberer operonet 70 -fold • Trp = korepressor (økt represjon) – ≠ inducer som svekker represjon

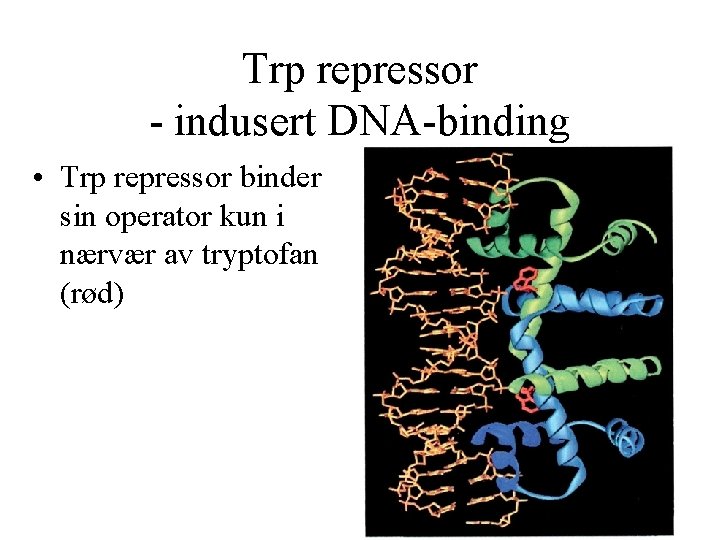
Trp repressor - indusert DNA-binding • Trp repressor binder sin operator kun i nærvær av tryptofan (rød)

Trp repressor

2. Nivå: regulering av terminering • Trp operon har en lang ledersekvens (trp. L)ds • Lav Trp: hele den polycistroniske m. RNA produseres • Høy Trp: en kort versjon lages, prematurt terminert i trp. L • Attenuator = kontrollelement ansvarlig for prematur terminering

Trp-operon

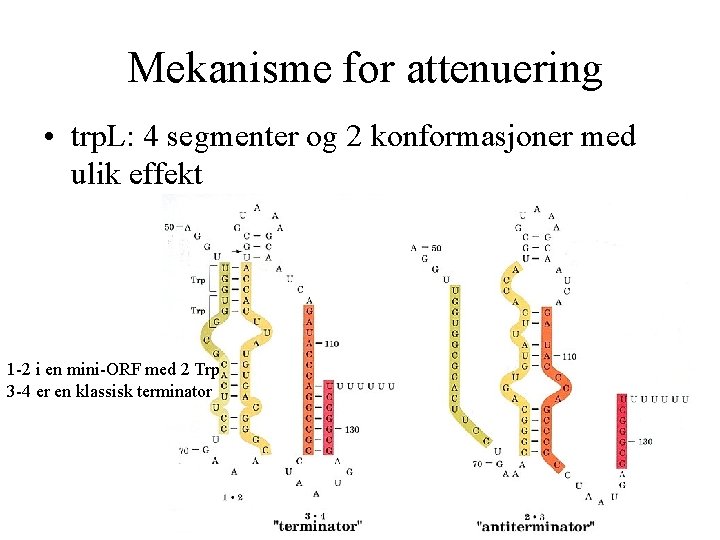
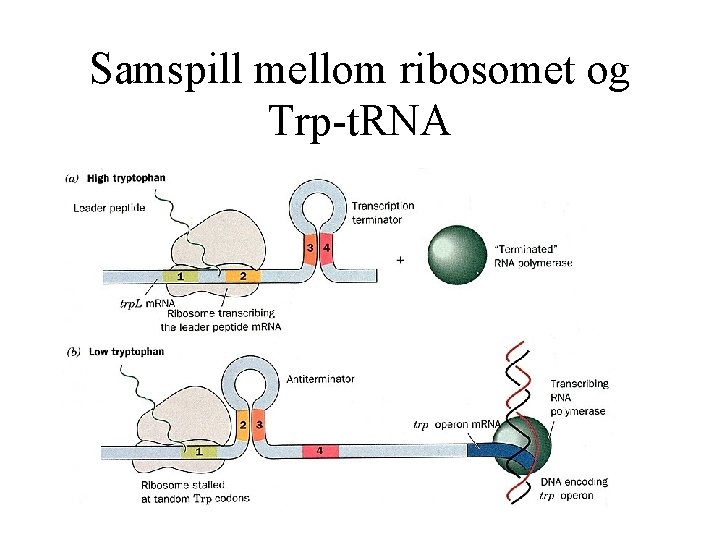
Mekanisme for attenuering • trp. L: 4 segmenter og 2 konformasjoner med ulik effekt 1 -2 i en mini-ORF med 2 Trp 3 -4 er en klassisk terminator

Modell • Mye Trp: ribosomet starter straks translasjon og fremmer konformasjonen som gir terminering • Lite Trp: ribosomets venting på Trp-t. RNA fremmer 2 -3 konformasjon som tillater videre transkripsjon

Samspill mellom ribosomet og Trp-t. RNA

Page 1253 Amino Acid Sequences of Some Leader Peptides in Operons Subject to Attentuation.

Gener for ribosomalt RNA i E. coli • r. RNA i bakterier: 23 S, 16 S og 5 S • Under optimale vekstforhold inneholder en bakteriecelle rundt 10000 ribosomer • Dersom det var bare en kopi av genene for r. RNA ville det kun være rundt 1200 ribosomer/celle • E. coli-genomet inneholder 7 separate r. RNAoperoner, hvert med en kopi av hvert gen

Stringent respons Eksempel på regulering via signalmolekyl som speiler vekstforhold

pp. Gpp • Stringent respons: ved aminosyremangel skjer en metabolsk tilpasning hvor r. RNAog t. RNA-syntesen reduseres 10 -20 fold • Ved stringent respons akkumuleres pp. Gpp • Syntese: rel. A (stringent faktor) – ATP + GTP = AMP + pp. Gpp • Degradering: spo. T