Mars 2011 ATELIER EPIGENETIQUE Utilisation de la QPCR











- Slides: 19

Mars 2011 ATELIER EPIGENETIQUE Utilisation de la Q-PCR pour analyser des données de Ch. IP ou de Me. DIP Emmanuèle Mouchel-Vielh

I. La PCR quantitative: principe et généralités II. Application aux expériences de Ch. IP et de Me. DIP

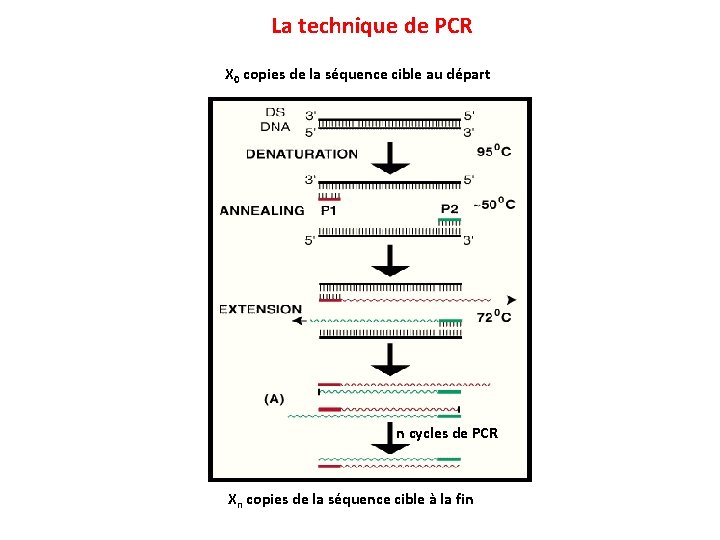
La technique de PCR X 0 copies de la séquence cible au départ n cycles de PCR Xn copies de la séquence cible à la fin

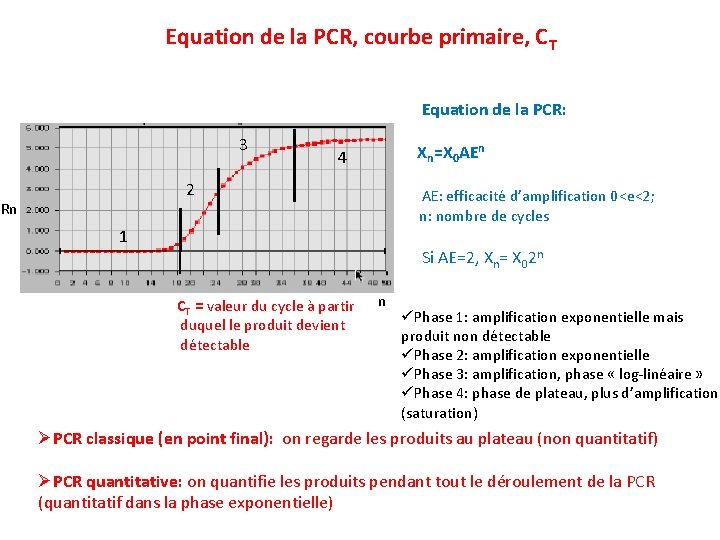
Equation de la PCR, courbe primaire, CT Equation de la PCR: 3 Xn=X 0 AEn 4 AE: efficacité d’amplification 0<e<2; 2 Rn n: nombre de cycles 1 Si AE=2, Xn= X 02 n CT = valeur du cycle à partir duquel le produit devient détectable n üPhase 1: amplification exponentielle mais produit non détectable üPhase 2: amplification exponentielle üPhase 3: amplification, phase « log-linéaire » üPhase 4: phase de plateau, plus d’amplification (saturation) ØPCR classique (en point final): on regarde les produits au plateau (non quantitatif) ØPCR quantitative: on quantifie les produits pendant tout le déroulement de la PCR (quantitatif dans la phase exponentielle)

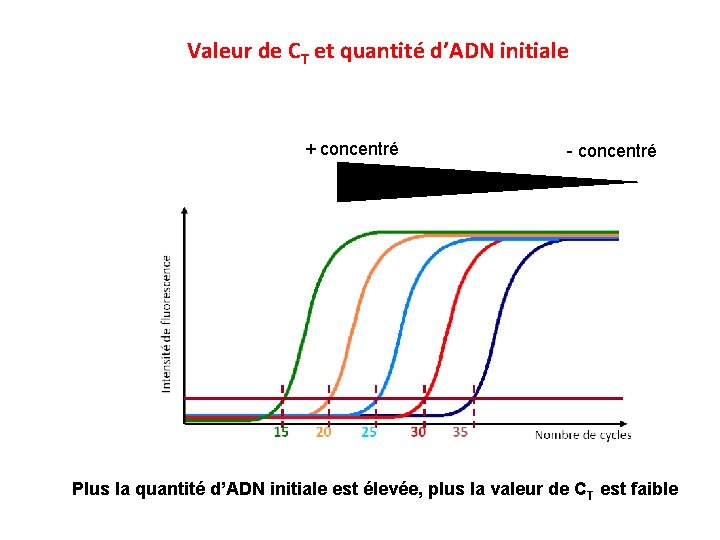
Valeur de CT et quantité d’ADN initiale + concentré - concentré Plus la quantité d’ADN initiale est élevée, plus la valeur de C T est faible

Propriétés générales de la PCR quantitative ü Détection à partir de 100 copies d’une séquence dans l’échantillon (environ 10 -15 g) ü Quantification absolue ou relative ü Très nombreuses applications (génotypage, étude de l’expression des gènes, ch. IP. . . ) üPour avoir la meilleur efficacité possible: amplicons de petite taille (70 à 250/300 bp) ü Principe général de détection: utilisation d’un (ou plusieurs) fluorochromes: l’émission de fluorescence est le reflet de la quantité d’ADN synthétisée à chaque cycle ü Principe des appareils de Q-PCR: un thermocycleur couplé à un fluorimètre qui mesure la fluorescence émise à chaque cycle (fluorimètre= laser pour exciter les fluorochromes+ système de détection de la fluorescence émise) üEtablissement de la courbe primaire de PCR : émission de fluorescence=f(n). Cette courbe permet de déterminer la valeur de CT , qui permet ensuite d’estimer (de manière absolue ou relative) la quantité X 0 présente au départ dans l’échantillon.

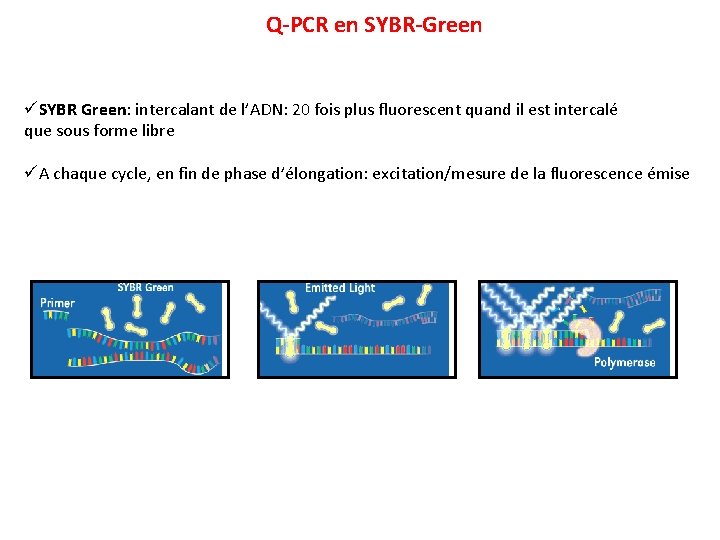
Q-PCR en SYBR-Green üSYBR Green: intercalant de l’ADN: 20 fois plus fluorescent quand il est intercalé que sous forme libre üA chaque cycle, en fin de phase d’élongation: excitation/mesure de la fluorescence émise

Avantages et inconvénients de la Q-PCR en SYBR-Green Avantages: üMéthode simple à mettre en œuvre, en particulier au niveau du choix des amorces üMéthode la moins chère Inconvénients: ü Ne permet pas de faire des PCR multiplex (=amplifications de plusieurs séquences dans le même tube) ü Moins sensible que d’autres méthodes (détection à partir de 1000 copies) ) üRisque possible d’aspécificité (production d’amplicons aspécifiques, quantifiés comme les amplicons spécifiques): pour vérifier, réalisation d’une courbe de fusion en fin de PCR d’’( 1 pic= 1 amplicon (si dimères d’amorces, 2ème pic avec fusion vers 50°C) Température

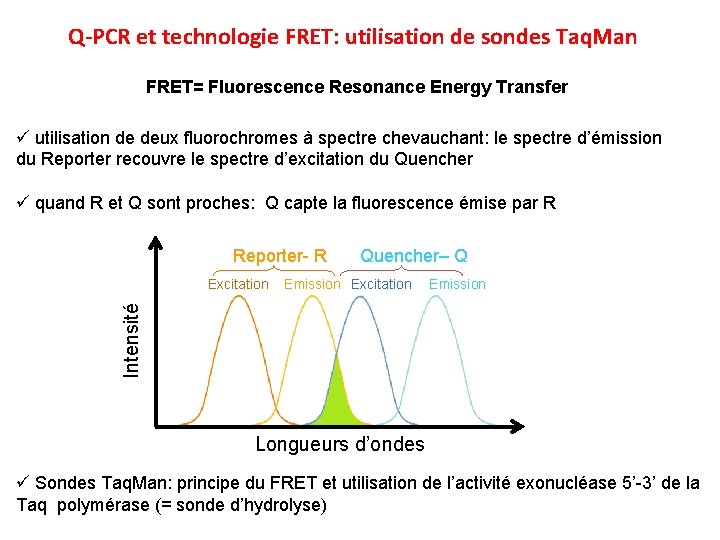
Q-PCR et technologie FRET: utilisation de sondes Taq. Man FRET= Fluorescence Resonance Energy Transfer ü utilisation de deux fluorochromes à spectre chevauchant: le spectre d’émission du Reporter recouvre le spectre d’excitation du Quencher ü quand R et Q sont proches: Q capte la fluorescence émise par R Reporter- R Emission Excitation Emission Intensité Excitation Quencher– Q Longueurs d’ondes ü Sondes Taq. Man: principe du FRET et utilisation de l’activité exonucléase 5’-3’ de la Taq polymérase (= sonde d’hydrolyse)

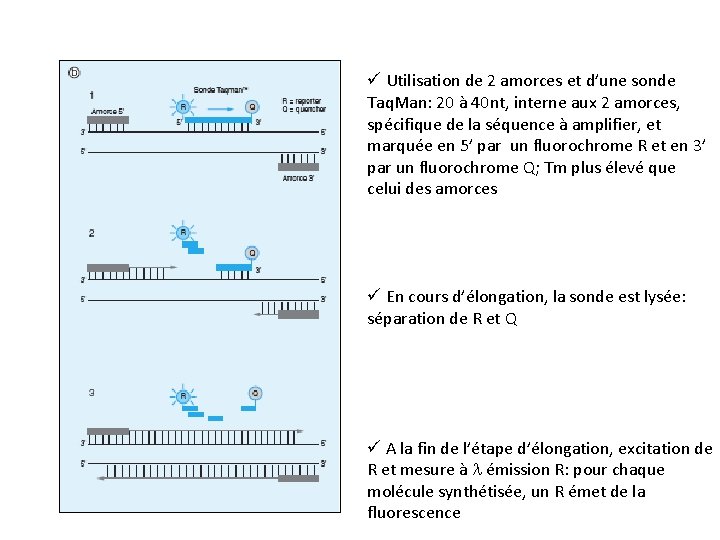
ü Utilisation de 2 amorces et d’une sonde Taq. Man: 20 à 40 nt, interne aux 2 amorces, spécifique de la séquence à amplifier, et marquée en 5’ par un fluorochrome R et en 3’ par un fluorochrome Q; Tm plus élevé que celui des amorces ü En cours d’élongation, la sonde est lysée: séparation de R et Q ü A la fin de l’étape d’élongation, excitation de R et mesure à l émission R: pour chaque molécule synthétisée, un R émet de la fluorescence

Avantages et inconvénients de la Q-PCR en sondes Taq. Man Avantages: ü permet les PCR multiplex (utilisation de différents couples Q-R) ü élimine le problème des hybridations aspécifiques ü plus sensible que la méthode SYBR-Green Inconvénients: üMoins facile à mettre en œuvre que la méthode SYBR-Green (qualité de la sonde Taq. Man) ü Plus cher que la méthode SYBR-Green

Exemples d’applications de la Q-PCR ü Analyse du niveau d’expression des gènes, mise en évidence de variants d’épissage… (QRT-PCR) ü Détection et quantifications d’agents pathogènes ü Génotypage, étude de polymorphismes ü Analyse d’expériences de Ch. IP ou de Me. DIP ü …. .

Interprétation des résultats de Ch. IP et Me. DIP par Q-PCR ü Choix du système de fluorescence ü Choix de la séquence à amplifier: petite taille des amplicons (50 à 150 bp) üChoix des amorces: critères classiques (absence de structure secondaire, pas d’appariement entre amorces, spécificité) ü Validation des amorces: réalisation d’une courbe de calibration pour vérifier leur efficacité: réactions de Q-PCR ( en triplicats) sur des dilutions en série d’un ADN contenant la séquence matrice à amplifier (ADNc, ADN génomique, plasmide…: non nécessaire de connaitre la concentration de la séquence matrice dans cet ADN): l’ efficacité doit être proche de 100%

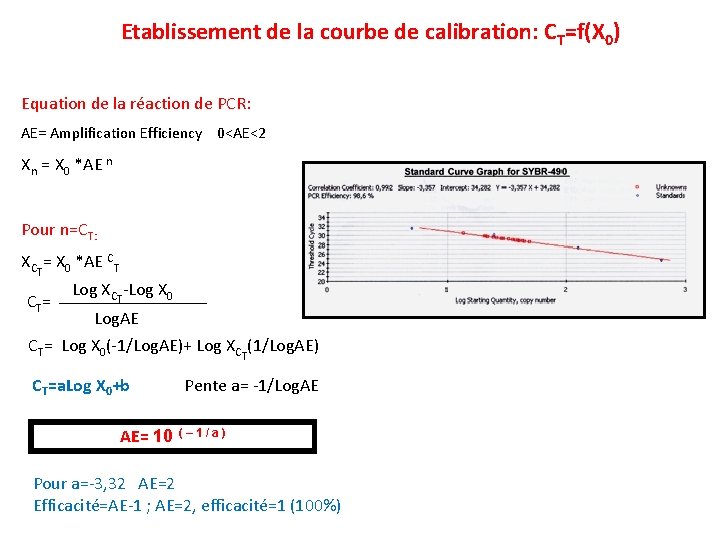
Etablissement de la courbe de calibration: CT=f(X 0) Equation de la réaction de PCR: AE= Amplification Efficiency 0<AE<2 Xn = X 0 *AE n Pour n=CT: XCT= X 0 *AE CT CT= Log XCT-Log X 0 Log. AE CT= Log X 0(-1/Log. AE)+ Log XCT(1/Log. AE) CT=a. Log X 0+b Pente a= -1/Log. AE AE= 10 ( – 1 / a ) Pour a=-3, 32 AE=2 Efficacité=AE-1 ; AE=2, efficacité=1 (100%)

Calculs pour les expériences de Ch. IP et Me. DIP Fd= facteur de dilution de l’input (ici: 50 pour le Ch. IP et 10 pour le Me. DIP) % (IP/ Total input)= AE^[Ctinput-(Log. Fd/Log AE)– Ct. IP]x 100% ou % input= AE^[(Ctinput – Ct. IP) x Fdx 100%] Pour Hoxc 8: AE=1, 96; efficacité=98%


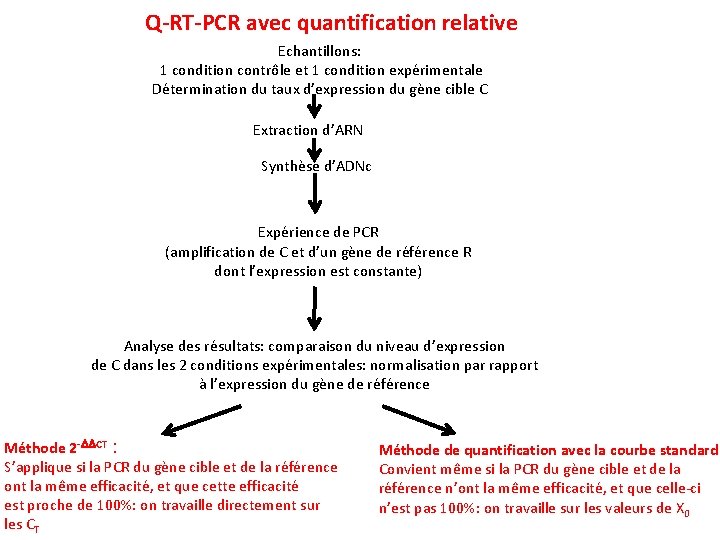
Q-RT-PCR avec quantification relative Echantillons: 1 condition contrôle et 1 condition expérimentale Détermination du taux d’expression du gène cible C Extraction d’ARN Synthèse d’ADNc Expérience de PCR (amplification de C et d’un gène de référence R dont l’expression est constante) Analyse des résultats: comparaison du niveau d’expression de C dans les 2 conditions expérimentales: normalisation par rapport à l’expression du gène de référence Méthode 2 -DDCT : S’applique si la PCR du gène cible et de la référence ont la même efficacité, et que cette efficacité est proche de 100%: on travaille directement sur les CT Méthode de quantification avec la courbe standard Convient même si la PCR du gène cible et de la référence n’ont la même efficacité, et que celle-ci n’est pas 100%: on travaille sur les valeurs de X 0

Quantification relative par méthode de Livak ( méthode 2 -DDCT) -A partir des courbes de PCR, détermination des CT dans les échantillons étudiés (condition contrôle et expérimentale) pour le gène cible et le gène de référence -Calcul de DCT=CT gène cible-CT gène de référence pour tous les échantillons - Calcul de DDCT=(DCT)contrôle-(DCT)expérimental -Calcul de 2 -DDCT: Si 2 -DDCT >1, gène sur-exprimé dans la condition expérimentale par rapport à la condition contrôle Si 2 -DDCT <1, gène sous-exprimé dans la condition expérimentale par rapport à la condition contrôle

Quantification relative par courbe standard -A partir des courbes de calibration, détermination de Log(X 0) dans les échantillons étudiés (condition contrôle et expérimentale) pour le gène cible et le gène de référence. -Normalisation par rapport à l’expression du gène de référence pour chaque échantillon (condition contrôle et expérimentale): Norm=X 0(gène cible)/X 0(gène référence) -On fait le rapport R= Norm(expé)/Norm(contrôle): Si R>1, gène sur-exprimé dans la condition expérimentale par rapport à la condition contrôle; Si R<1, gène sous-exprimé dans la condition expérimentale par rapport à la condition contrôle