Les nouvelles preuves de lvolution y accder en

























































- Slides: 71

Les nouvelles preuves de l’évolution: y accéder en classe PO 422 Phylogénétique Novembre 2009 1

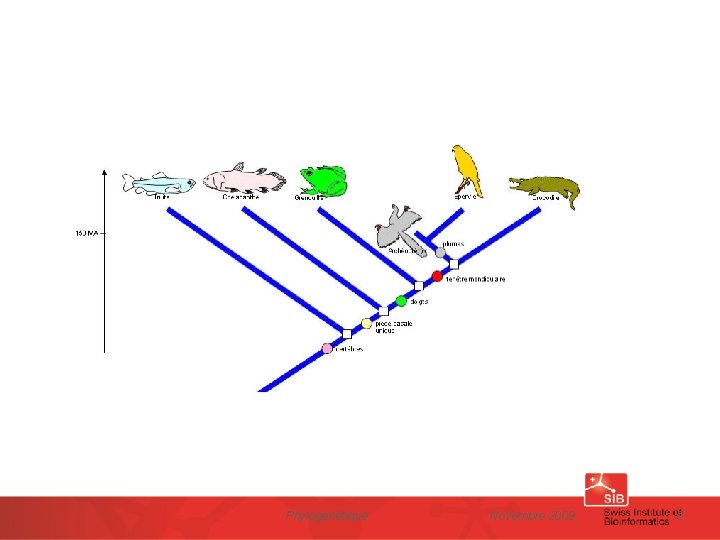
Phylogénie Etude des relations de parenté entre espèces. Une analyse phylogénétique permet d’estimer (modéliser) les relations évolutives qui existent entre les espèces grâce à un arbre: qui a un ancêtre commun - qui est le cousin de qui ? Phylogénétique Novembre 2009 2

http: //www. unige. ch/450/expositions/genome/presentation/slogans. html Phylogénétique Novembre 2009 3

Il est possible de construire un arbre phylogénétique à partir de différents types de données: – Les données morphologiques (écailles ou plumes, présence de certains os du crâne, forme des feuilles…). Il existe quelques centaines de caractères définis dans ce but par les spécialistes. – Les caractères physiologiques (température corporelle…) Phylogénétique Novembre 2009 4

Phylogénétique Novembre 2009 5

Il est possible de construire un arbre phylogénétique à partir de différents types de données: – les données morphologiques (écailles ou plumes, présence de certains os du crâne, , forme des feuilles…). Il existe quelques centaines de caractères définis dans ce but par les spécialistes. – Les caractères physiologiques (température corporelle…) Mais aussi…. – L’ordre des gènes (par exemple sur l’ADN des mitochondries) – les données moléculaires (séquences d’ADN ou de protéines). Des mutations modifient les séquences de l’ADN et par conséquent des protéines au cours de l’évolution. – toutes les données existantes…. (défi scientifique !) Phylogénétique Novembre 2009 6

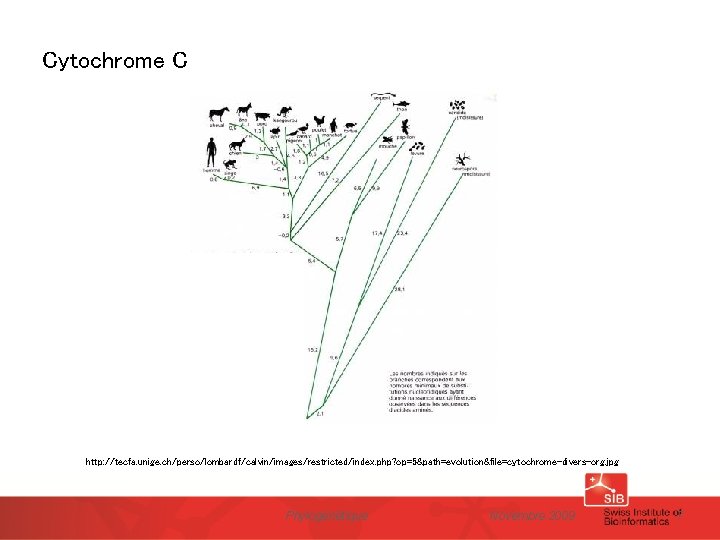
Cytochrome C http: //tecfa. unige. ch/perso/lombardf/calvin/images/restricted/index. php? op=5&path=evolution&file=cytochrome-divers-org. jpg Phylogénétique Novembre 2009 7

Comment construire des arbres phylogénétiques sur la base des séquences des protéines… Phylogénétique Novembre 2009 8

Actin-related protein 2 ARP 2_A ARP 2_B ARP 2_C ARP 2_D ARP 2_E MESAP---IVLDNGTGFVKVGYAKDNFPRFQFPSIVGRPILRAEEKTGNVQIKDVMVGDE MDSQGRKVIVVDNGTGFVKCGYAGTNFPAHIFPSMVGRPIVRSTQRVGNIEIKDLMVGEE MDSQGRKVVVCDNGTGFVKCGYAGSNFPEHIFPALVGRPIIRSTTKVGNIEIKDLMVGDE MDSKGRNVIVCDNGTGFVKCGYAGSNFPTHIFPSMVGRPMIRAVNKIGDIEVKDLMVGDE *: * **** ***. **: : ****: : *: : **: * Les différentes espèces sont: Caenorhabditis briggsae Drosophila melanogaster Homo sapiens Mus musculus Schizosaccharomyces pombe ? Quelle séquence ‘appartient’ à quelle espèce ? Phylogénétique Novembre 2009 9

• • • ARP 2_A ARP 2_B MESAP---IVLDNGTGFVKVGYAKDNFPRFQFPSIVGRPILRAEEKTGNVQIKDVMVGDE MDSQGRKVIVVDNGTGFVKCGYAGTNFPAHIFPSMVGRPIVRSTQRVGNIEIKDLMVGEE *: * **: **** ***: *****: *: : . . **: : ***: * • • • ARP 2_B ARP 2_C MDSQGRKVIVVDNGTGFVKCGYAGTNFPAHIFPSMVGRPIVRSTQRVGNIEIKDLMVGEE MDSQGRKVVVCDNGTGFVKCGYAGSNFPEHIFPALVGRPIIRSTTKVGNIEIKDLMVGDE ****: * *******: : *****: ******: * • • • ARP 2_C ARP 2_D MDSQGRKVVVCDNGTGFVKCGYAGSNFPEHIFPALVGRPIIRSTTKVGNIEIKDLMVGDE ****************************** • • • ARP 2_D ARP 2_E MDSQGRKVVVCDNGTGFVKCGYAGSNFPEHIFPALVGRPIIRSTTKVGNIEIKDLMVGDE MDSKGRNVIVCDNGTGFVKCGYAGSNFPTHIFPSMVGRPMIRAVNKIGDIEVKDLMVGDE ***: *: **********: : ****: . . *: *: ******** • • • ARP 2_A ARP 2_C MESAP---IVLDNGTGFVKVGYAKDNFPRFQFPSIVGRPILRAEEKTGNVQIKDVMVGDE MDSQGRKVVVCDNGTGFVKCGYAGSNFPEHIFPALVGRPIIRSTTKVGNIEIKDLMVGDE *: * **** ***. **: : *****: *: *. **: : ***** • • • ARP 2_D ARP 2_B MDSQGRKVVVCDNGTGFVKCGYAGSNFPEHIFPALVGRPIIRSTTKVGNIEIKDLMVGDE MDSQGRKVIVVDNGTGFVKCGYAGTNFPAHIFPSMVGRPIVRSTQRVGNIEIKDLMVGEE ****: * *******: : *****: ******: * Phylogénétique Novembre 2009 10

• Philo. Phylo ‘kit tout en 1’, mais avec un choix limité de protéines au départ… http: //education. expasy. org/philophylo • www. uniprot. org: banques de données de protéines (encyclopédie) contenant des informations biologiques et les séquences en acides aminés. • www. phylogeny. fr Phylogénétique Novembre 2009 11

Le principe • 1. Sélection: set de séquences de protéines ‘homologues’ • 2. Comparaison: alignement multiple • 3. Construction de l’arbre: ‘calculer les différences’ Phylogénétique Novembre 2009 12

Le principe • Sélection: set de séquences de protéines ‘homologues’ • Comparaison: alignement multiple • Construction de l’arbre: ‘calculer les différences’ Phylogénétique Novembre 2009 13

Philo. Phylogénétique Novembre 2009 14

www. uniprot. org • ‘query’ par nom de protéines ou nom de gènes Utiliser http: //www. uniprot. org/ Exemples: http: //education. expasy. org/cours/FLO/Liste_prot_evol. html • Blast Utiliser http: //www. expasy. org/tools/blast/ ou le Blast@Uni. Prot Phylogénétique Novembre 2009 15

Scénario 5: www. uniprot. org Phylogénétique Novembre 2009 16

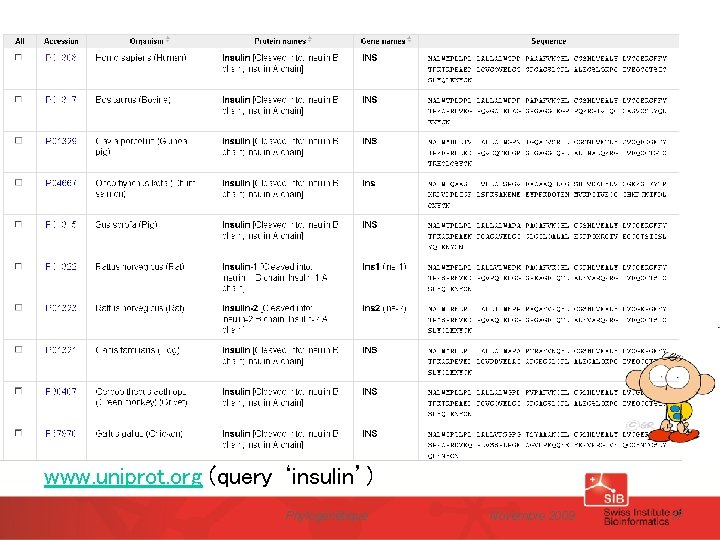
www. uniprot. org (query ‘insulin’) Phylogénétique Novembre 2009 17

Protein and gene name. S www. uniprot. org/ query FOLH 1 Phylogénétique Novembre 2009 18

Liste de protéines ‘candidats’ http: //education. expasy. org/cours/FLO/Liste_prot_evol. html Phylogénétique Novembre 2009 19

Le principe • Sélection: set de séquences de protéines ‘homologues’ • Comparaison: alignement multiple • Construction de l’arbre: ‘calculer les différences’ Phylogénétique Novembre 2009 20

Philo. Phylogénétique Novembre 2009 21

Alignement multiple des séquences en acides aminés de l’insuline de différentes espèces www. uniprot. org (query ‘insulin’, align) Phylogénétique Novembre 2009 22

http: //www. unige. ch/450/expositions/genome/presentation/slogans. html Phylogénétique Novembre 2009 23

• Les – – – • Les conséquences des mutations peuvent être des changements ‘mineurs’ (changement d’un acide aminé pour un autre) ou majeurs (duplication de gènes, délétion d’un bout de chromosomes, …) • Chez les mammifères, à chaque génération, il se produit en moyenne quelques dizaines de mutations dans le génome de chaque individu. mutations dans l’ADN peuvent être dûes à Des erreurs lors de la réplication de l’ADN Des agents chimiques ou physiques (fumée de cigarette, UV, …) Des virus – Si ces mutations se trouvent dans l’ADN des cellules sexuelles, elles seront transmises à la descendance. – La dérive génétique (‘genetic drift’) et/ou la sélection naturelle (favorisant la sélection des individus avec des mutations qui augmentent les chances de reproduction et de survie dans un environnement donné) fixeront les mutations dans une population puis une espèce donnée. Phylogénétique Novembre 2009 24

http: //www 3. interscience. wiley. com/cgi-bin/fulltext/117981248/HTMLSTART (i) FR 1 • The FRIGIDA (FR 1) gene has been shown to be a major determinant of flowering time in Arabidopsis thaliana. A majority of early-flowering ecotypes shows one or two deletions that generate a frameshift in the FR 1 open reading frame (ORF), suggesting that this phenotype has arisen at least twice. Le Corre, Roux & Reboud (2002) performed a population analysis on different ecotypes and confirmed that the loss of function mutations was associated with an early-flowering phenotype, these gene inactivations systematically evolved in a non-neutral fashion. Moreover, they confirmed that the gene inactivation was phenotypically linked to an early flowering ecotype adaptated to cold environments (Johanson et al. , 2000; Le Corre et al. , 2002). This represents a strong indication that environmental change has driven this genetic change. (ii) CCR 5 • This primate transmembrane receptor is a cellular gateway for the entry of HIV-1 and all strains of SIV. Human homozygotes for the CCR 5 null allele which has a 32 base pair (bp) deletion are highly resistant to HIV-1 infection. Another null allele (24 bp deletion) of CCR 5 has convergently evolved in sooty mangabeys (Cercocebus atys), a natural host of SIV. The occurrence of the mangabey null allele at an appreciable frequency (around 4%) could be explained by positive selection; null homozygotes are protected from SIV infection because the encoded protein is not transported to the cell surface (Palacios et al. , 1998). The null allele has been shown to be positively selected in humans (Galvani & Novembre, 2005). However, the exact nature of the selective pressure involved in the origin of the CCR 5 -{delta} 32 allele and its high prevalence in European populations (approximately 10%) is unclear as the HIV epidemic in humans is much more recent than the age of the null allele (about 700 years). However, both HIV and poxviruses enter leukocytes using chemokine receptors; it is plausible that the loss of the CCR 5 chemokine receptor originally conferred resistance against smallpox. This hypothesis is supported by a correlation between historical smallpox epidemics and allele geographic distribution (Galvani & Slatkin, 2003). Phylogénétique Novembre 2009 25

On ne peut pas ‘muter’ n’importe quoi…. Hélice alpha Peptide signal Hélice alpha Phylogénétique Novembre 2009 26

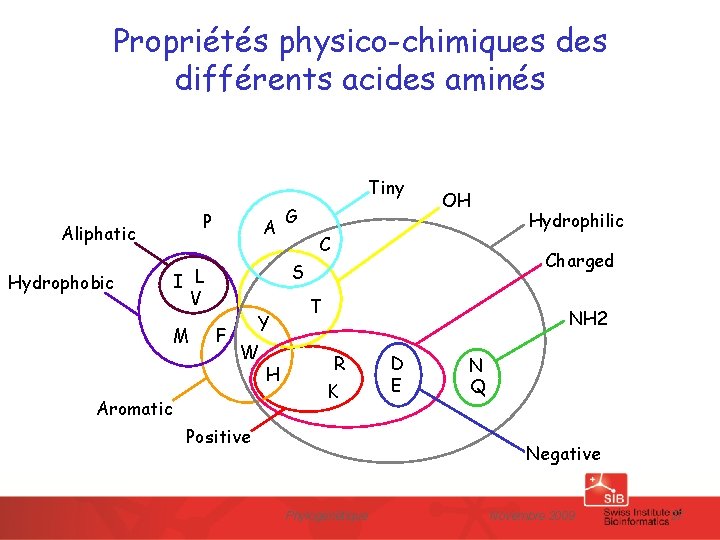
Propriétés physico-chimiques différents acides aminés Tiny P Aliphatic Hydrophobic A C F Y W Aromatic H Hydrophilic Charged S I L V M G OH T NH 2 R K Positive D E N Q Negative Phylogénétique Novembre 2009 27

www. uniprot. org (query ‘insulin’, align) Phylogénétique Novembre 2009 28

Les gènes (et les protéines) évoluent à des vitesses différentes L’histone H 4 n’a accumulé que 2 mutations ‘conservatives’ en 1. 5 milliard d’années Phylogénétique Novembre 2009 29

Les gènes (et les protéines) évoluent à des vitesses différentes Une protéine qui a évolué extrêmement rapidement: l’involucrin (une protéine de la peau): 10 % de changements en l’espace de 4 millions d’années (séparation des chimpanzés et de l’homme) Phylogénétique Novembre 2009 30

Le principe • Sélection: set de séquences de protéines ‘homologues’ • Comparaison: alignement multiple • Construction de l’arbre: ‘calculer les différences’ Phylogénétique Novembre 2009 31

• Un arbre phylogénétique est un modèle qui décrit les relations entre des unités taxonomiques Phylogénétique Novembre 2009 32

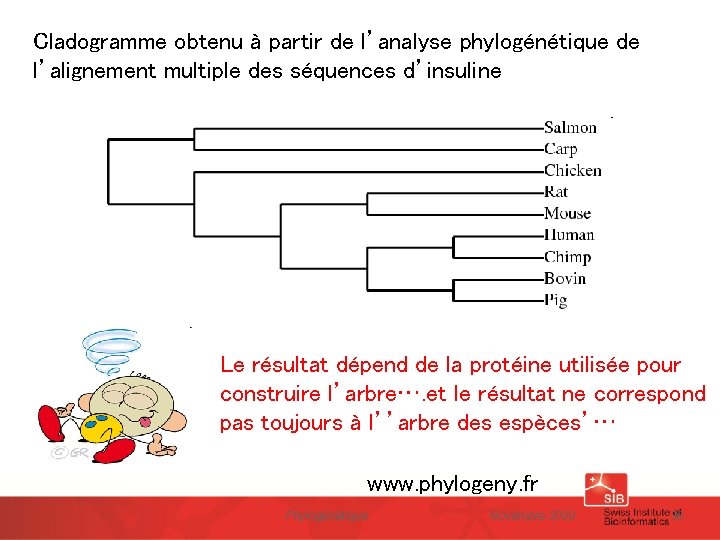
Le résultat dépend de la protéine utilisée pour construire l’arbre…. et le résultat ne correspond pas toujours à l’’arbre des espèces’… Phylogénétique Novembre 2009 33

www. phylogeny. fr • Introduire le set de séquences sélectionnées à www. uniprot. org (format ‘fasta’) • Utiliser les paramètres par défaut (‘one’ click) Phylogénétique Novembre 2009 34

www. phylogeny. fr Phylogénétique Novembre 2009 35

Cladogramme obtenu à partir de l’analyse phylogénétique de l’alignement multiple des séquences d’insuline Le résultat dépend de la protéine utilisée pour construire l’arbre…. et le résultat ne correspond pas toujours à l’’arbre des espèces’… www. phylogeny. fr Phylogénétique Novembre 2009 36

Les différents types d’arbres (1) Phylogénétique Novembre 2009 37

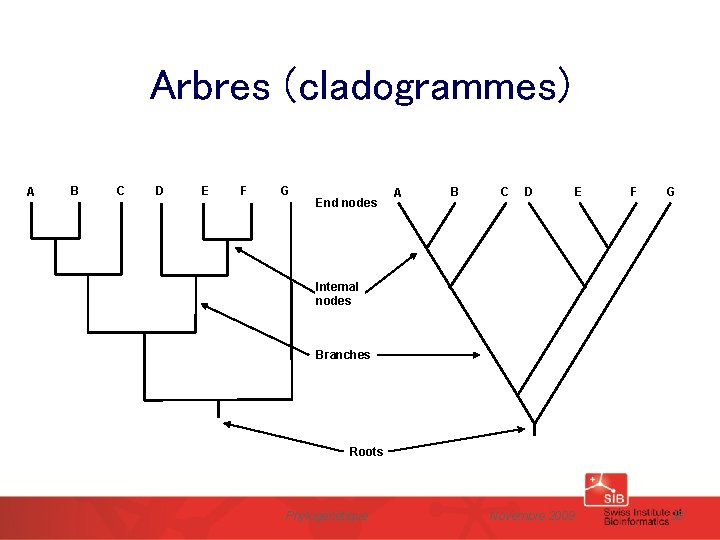
Arbres (cladogrammes) A B C D E F G End nodes A B C D E F G Internal nodes Branches Roots Phylogénétique Novembre 2009 38

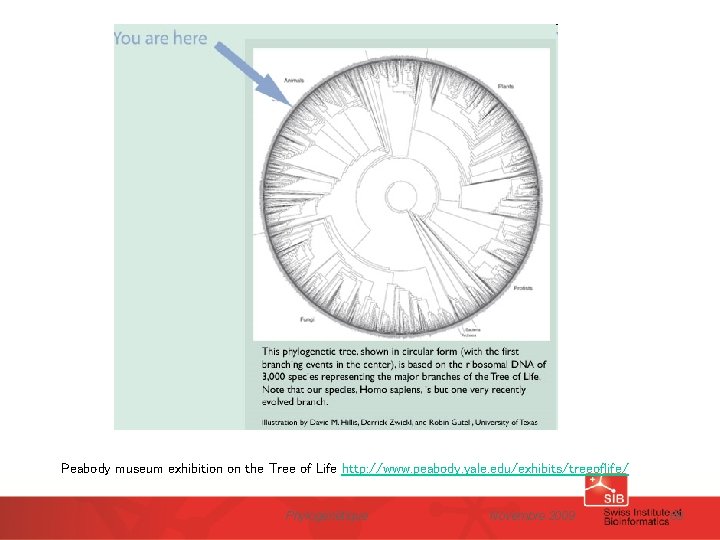
Peabody museum exhibition on the Tree of Life http: //www. peabody. yale. edu/exhibits/treeoflife/ Phylogénétique Novembre 2009 39

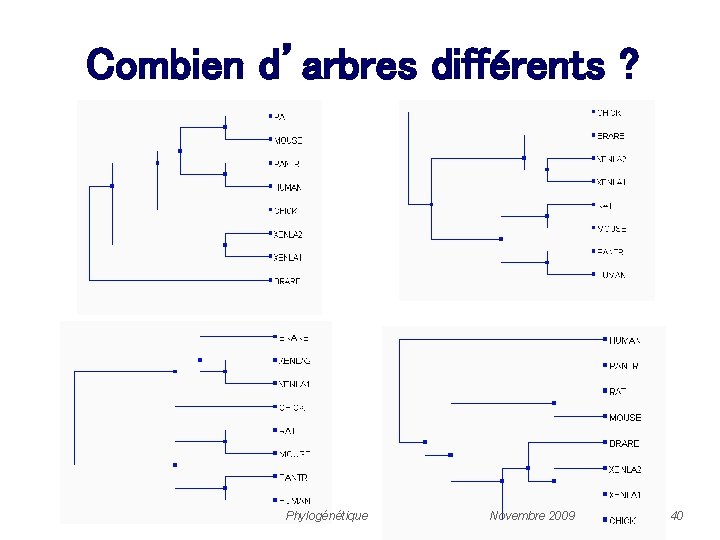
Combien d’arbres différents ? Phylogénétique Novembre 2009 40

Les différents types d’arbres (2) Phylogénétique Novembre 2009 41

Arbres phylogénétiques • Cladogramme Qui est le cousin de qui ? • Phylogramme La longueur des branches est proportionnelle aux différences. Cette longueur n’est pas toujours facile à interpréter ! Elle dépend entre autre de la vitesse d’évolution…(nombres de variations par site dans un laps de temps donné) Phylogénétique Novembre 2009 42

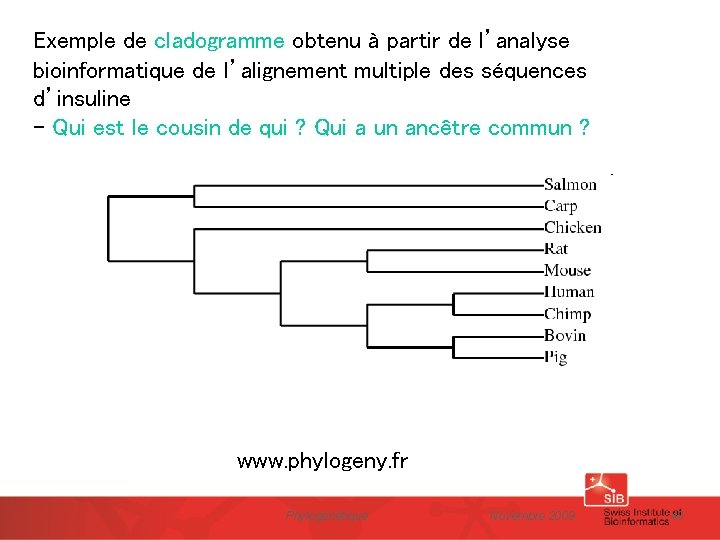
Exemple de cladogramme obtenu à partir de l’analyse bioinformatique de l’alignement multiple des séquences d’insuline - Qui est le cousin de qui ? Qui a un ancêtre commun ? www. phylogeny. fr Phylogénétique Novembre 2009 43

Exemple de phylogramme obtenu à partir de l’analyse bioinformatique de l’alignement multiple des séquences d’insuline - Quelle protéine a évolué plus rapidement ? Echelle: ‘expected number of changes per site’ Le temps qui s’écoule…. Phylogénétique Novembre 2009 44

Exemple de phylogramme obtenu à partir de l’analyse bioinformatique de l’alignement multiple des séquences d’insuline L’insuline de la carpe a ‘évolué’ (moins de changements dans sa séquences en acides aminés) moins rapidement que l’insuline des autres espèces… Probabilité que l’arbre ait la bonne configuration à chaque embranchement (> 0. 95: OK) Le temps qui s’écoule…. Phylogénétique Novembre 2009 45

Les embranchements • Spéciation • Duplication de gènes Phylogénétique Novembre 2009 46

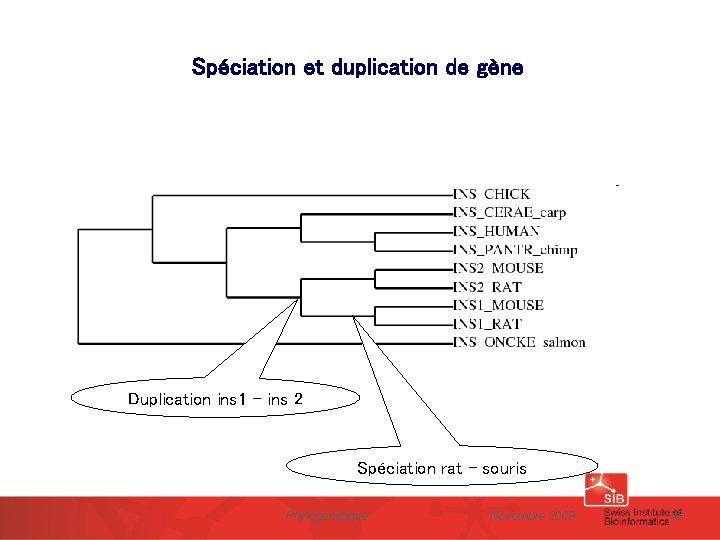
Spéciation et duplication de gène Frog gene A speciation Human gene A Orthologs speciation Mouse gene A Gene duplication Paralogs Mouse gene B Homologs speciation Ancestral gene speciation Human gene B Orthologs Frog gene B Drosophila gene AB Phylogénétique Novembre 2009 47

Spéciation et duplication de gène Duplication ins 1 – ins 2 Spéciation rat - souris Phylogénétique Novembre 2009 48

"Ce qui n'est pas entouré d'incertitudes ne peut pas être la vérité » R. Feynmann Phylogénétique Novembre 2009 49

A vous de jouer… Phylogénétique Novembre 2009 50

Ressources taxonomiques Phylogénétique Novembre 2009 51

http: //www. ncbi. nlm. nih. gov/guide/taxonomy/ Phylogénétique Novembre 2009 52

http: //www. uniprot. org/taxonomy/ Phylogénétique Novembre 2009 53

Animal Diversity Web (ADW) http: //animaldiversity. ummz. umich. edu/site/index. html Phylogénétique Novembre 2009 54

Perspectives et applications Phylogénétique Novembre 2009 55

Facteur d’élongation Les mitochondries et chloroplastes sont d’origine bactérienne ‘endosymbiotique’ http: //education. expasy. org/cours/FLO/Liste_prot_evol. html Phylogénétique Novembre 2009 56

Autres projets - ~200 protéines universelles - Arbre ‘global’ Applications - métagénomes (identifications de nouvelles espèces, de nouveaux gènes (20 mo), de nouveaux enzymes…. ) -1 ml d’eau de mer: 1 million de bactéries et 10 million de virus (C. Venter). - découvertes de nouvelles bactéries (la moitié de la biomasse) - médecine: identification de nouveaux pathogènes. Phylogénétique Novembre 2009 57

Références • http: //www. unige. ch/presse/Campus/campus 95. html Phylogénétique Novembre 2009 58

Liste de gènes intéressants ‘Conceptual bases for quantifying the role of the environment on gene evolution: the participation of positive selection and neutral evolution’ http: //www 3. interscience. wiley. com/cgi-bin/fulltext/117981248/HTMLSTART Phylogénétique Novembre 2009 59

http: //www 3. interscience. wiley. com/cgi-bin/fulltext/117981248/HTMLSTART (i) FR 1 • The FRIGIDA (FR 1) gene has been shown to be a major determinant of flowering time in Arabidopsis thaliana. A majority of early-flowering ecotypes shows one or two deletions that generate a frameshift in the FR 1 open reading frame (ORF), suggesting that this phenotype has arisen at least twice. Le Corre, Roux & Reboud (2002) performed a population analysis on different ecotypes and confirmed that the loss of function mutations was associated with an early-flowering phenotype, these gene inactivations systematically evolved in a non-neutral fashion. Moreover, they confirmed that the gene inactivation was phenotypically linked to an early flowering ecotype adaptated to cold environments (Johanson et al. , 2000; Le Corre et al. , 2002). This represents a strong indication that environmental change has driven this genetic change. (ii) CCR 5 • This primate transmembrane receptor is a cellular gateway for the entry of HIV-1 and all strains of SIV. Human homozygotes for the CCR 5 null allele which has a 32 base pair (bp) deletion are highly resistant to HIV-1 infection. Another null allele (24 bp deletion) of CCR 5 has convergently evolved in sooty mangabeys (Cercocebus atys), a natural host of SIV. The occurrence of the mangabey null allele at an appreciable frequency (around 4%) could be explained by positive selection; null homozygotes are protected from SIV infection because the encoded protein is not transported to the cell surface (Palacios et al. , 1998). The null allele has been shown to be positively selected in humans (Galvani & Novembre, 2005). However, the exact nature of the selective pressure involved in the origin of the CCR 5 -{delta} 32 allele and its high prevalence in European populations (approximately 10%) is unclear as the HIV epidemic in humans is much more recent than the age of the null allele (about 700 years). However, both HIV and poxviruses enter leukocytes using chemokine receptors; it is plausible that the loss of the CCR 5 chemokine receptor originally conferred resistance against smallpox. This hypothesis is supported by a correlation between historical smallpox epidemics and allele geographic distribution (Galvani & Slatkin, 2003). Phylogénétique Novembre 2009 60

Exemple de gènes / protéines • http: //education. expasy. org/cours/FLO/Liste_prot_evol. html Phylogénétique Novembre 2009 61

Divers - Simulation par ordinateur: avec 2 mécanismes, le hasard et la sélection, l’informaticien Karl Sim a généré une complexité fascinante: http: //www. archive. org/details/sims_evolved_virtual_crea tures_1994 Phylogénétique Novembre 2009 62

Phylogenetic servers • • • http: //www. phylogeny. fr/ http: //bioweb. pasteur. fr/seqanal/phylogeny/intro-uk. html http: //atgc. lirmm. fr/phyml/ http: //phylobench. vital-it. ch/raxml-bb/ http: //power. nhri. org. tw/power/home. htm Phylogénétique Novembre 2009 63

Un ancêtre commun Phylogénétique Novembre 2009 64

La construction d’arbre phylogénétique ne serait pas possible … sans la notion d’ancêtre commun… Les espèces ne sont pas immuables, mais issues d’autres espèces et peuvent elles-mêmes donner naissance à de nouvelles. La sélection naturelle est opportuniste et ne poursuit aucun but à long terme. Jean-Baptiste de Lamarck et Charles Darwin Phylogénétique Novembre 2009 65

• Cette notion d’ancêtre commun a été confirmée – par la découverte de l’ADN et du code génétique ‘quasi universel’* que se partagent tous les organismes vivants. *http: //www. ncbi. nlm. nih. gov/Taxonomy/Utils/wprintgc. cgi? mode=c – par la découverte de gènes très conservés entre les espèces (exemples: gène ‘engrailed’, histone H 4, ARN ribosomal) et de gènes ‘universels’ (~200). http: //education. expasy. org/cours/FLO/Liste_prot_evol. html Phylogénétique Novembre 2009 66

Peabody museum exhibition on the Tree of Life http: //www. peabody. yale. edu/exhibits/treeoflife/ Phylogénétique Novembre 2009 67

LUCA: Last Universal Common Ancestor LUCA est la ‘racine’ de l’arbre de la vie, mais probablement pas le premier organisme unicellulaire vivant. Il devait être déjà complexe et donc le résultat d’un long processus d’évolution Phylogénétique Novembre 2009 68

Peabody museum exhibition on the Tree of Life http: //www. peabody. yale. edu/exhibits/treeoflife/ Phylogénétique Novembre 2009 69

http: //www. peabody. yale. edu/exhibits/treeoflife/challenge. html Phylogénétique Novembre 2009 70

http: //www. sciencedaily. com/releases/2009/11/091104132706. htm Phylogénétique Novembre 2009 71