Les mthodes utilises en gnie gntique Module gnie



































































- Slides: 97

Les méthodes utilisées en : génie génétique Module : génie génétique Enseignante : Mme HAMDOUCHE

Plan: I- Préparation des acides nucléiques. Ø extraction des acides nucléiques ØLes enzymes agissant sur les AN II- Séparation des acides nucléiques Ø Electrophorèse

III- Détection, caractérisation et identification des AN Ø Marquage ØHybridation moléculaire ØSouthern blot IV- Amplification et sélection d’AN particuliers ØPCR ØClonage v les vecteurs

QU'EST-CE QUE LE GÉNIE GÉNÉTIQUE? Le génie génétique est le processus par lequel on identifie et isole l'ADN d'une cellule vivante ou morte pour l'introduire dans une autre cellule vivante. Comment les scientifiques peuvent-ils faire des manipulations génétiques? Ils utilisent la technologie de recombinaison de l'ADN. l'ensemble des outils et des techniques de la biologie moléculaire permettant, de manière contrôlée, l'étude de la modification des gènes : leur isolement, leur clonage, leur séquençage, leur découpage. . . dans un but de recherche fondamentale ou appliquée TECHNOLOGIE DE RECOMBINAISON DE L'ADN On appelle technologie de recombinaison de l'ADN les méthodes mises au point pour isoler, manipuler, amplifier, couper et épisser des séquences identifiables d'ADN. .

I- Préparation des acides nucléiques. 1 -1 extraction de l’ADN

2 - Les outils enzymatiques pour étudier les ADN

Plan: Les exonucléases 1 - Les nuleases Les endonucléases 2 - Les polymérases 3 - Enzymes modifiant l'ADN Les ligase

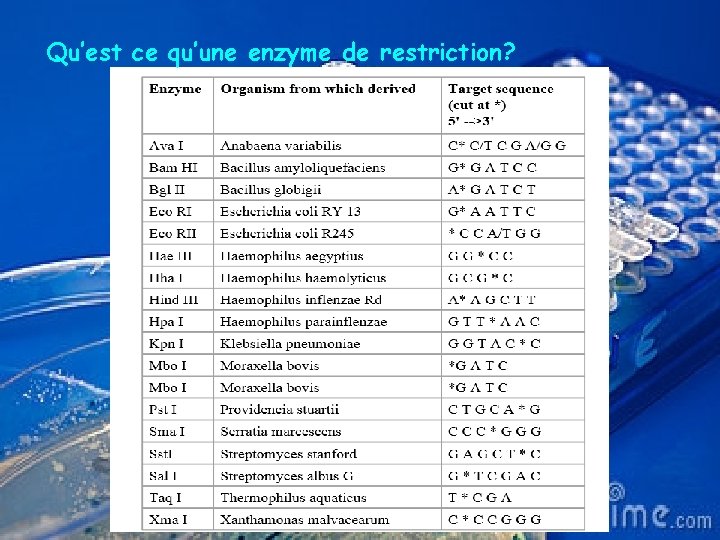
II- Les outils enzymatiques du génie génétique 1 - les enzymes de restriction Généralités • Découvertes à partir de 1973. Les enzymes de restriction sont capables de reconnaître spécifiquement une courte séquence, de 4 à 10 pb, et de cliver l'ADN au site reconnu.

Les enzymes de restriction appartiennent à la classe des endonucléases, c’est-à-dire des enzymes capables de cliver les liaisons phosphodiester entre deux nucléotides à l’intérieur d’un acide nucléique. Les endonucléases se différencient des exonucléases qui dégradent la molécule d’ADN à partir de l’une de ses extrémités (3’ ou 5’).

Origine des enzymes de restriction. Les enzymes de restriction sont extraites de micro-organismes, le plus souvent des bactéries. Les bactéries peuvent être parasitées par des virus à ADN. Les bactéries fabriquent des enzymes de restriction qui sont capables de cliver les ADN étrangers.

Séquences d’adn reconnues par les enzymes de restriction. • Les séquences de nucléotides reconnues par les enzymes de restriction sont habituellement des séquences dites palindromiques. Ces séquences palindromiques sont le plus souvent constituées de 4, 5 ou 6 paires de bases.

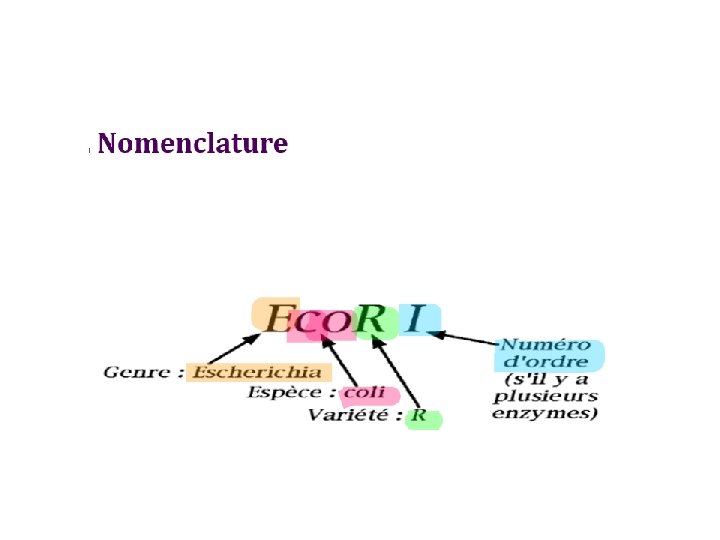
Nomenclature des enzymes de restriction. les enzymes de restriction présentent une nomenclature bien précise Example: Eco. R 1 genre: Escherichia espéce: coli la souche: R Order discovered: 1

Différents types de coupure Coupure franche Ø Certains enzymes coupent le site en son milieu et produisent deux fragments dont les extrémités sont franches. Coupure décalée Ø Cependant, la plupart réalisent une coupure dissymétrique : on parle dans ce cas d'extrémités cohésives (chaque fragment possède une chaîne qui dépasse l'autre de quelques bases)

Les enzymes de restriction de type II provoquent 2 types de coupure : • coupures a ≪ bouts francs ≫. Les enzymes coupent exactement au même niveau les 2 brins d'ADN : exemple Puu II qui coupe ↓ 5' GGCC 3' 3' CCGG 5‘ ↑ • coupures a ≪ bouts cohésifs ≫. Dans ce cas, les coupures sont décalées l'une par rapport a l'autre. -Les parties simple brin complémentaire peuvent s'apparier C'est une propriétés très utilisée dans les recombinaisons génétiques in vitro

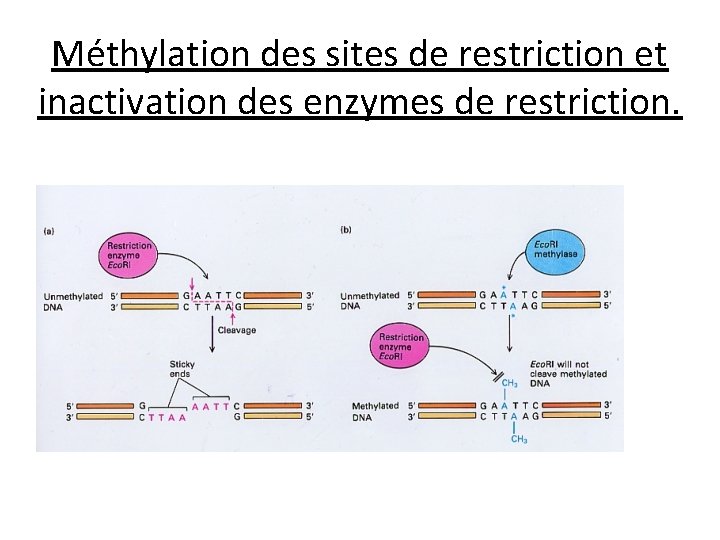
Méthylation des sites de restriction et inactivation des enzymes de restriction.

Les types des enzymes de restriction Ø 1 - enzymes de type I Ø 2 - enzymes de type II : plus utilisées aux laboratoires Ø 3 - enzymes de type III

Type I : l'enzyme reconnait sa séquence puis se déplace sur l'ADN et s'arrete de manière aléatoire 1000 a 5000 paires plus loin et libère quelques dizaines de nucléotides. Type II : une fois la séquence reconnue, l'enzyme coupe l'ADN au niveau de cette séquence (les plus courante en biologie moléculaire). Type III : âpres reconnaissance de la séquence spécifique, ces enzymes découpent l'ADN une vingtaine de nucléotides plus loin.

Utilisations des enzymes de restriction. Ø Les utilisations des enzymes de restriction sont très nombreuses en biologie moléculaire. Ø elles permettent de fractionner l’ADN en multiples fragments susceptibles d’être séparés par les techniques d’électrophorèse. Ø Les enzymes de restriction peuvent être utilisées pour préparer un fragment d’ADN d’un gène donné (insert) à être inséré dans un vecteur comme un plasmide ADN recombinant. Ø Les enzymes de restriction sont utilisées couramment pour recher des mutations dans le génome.

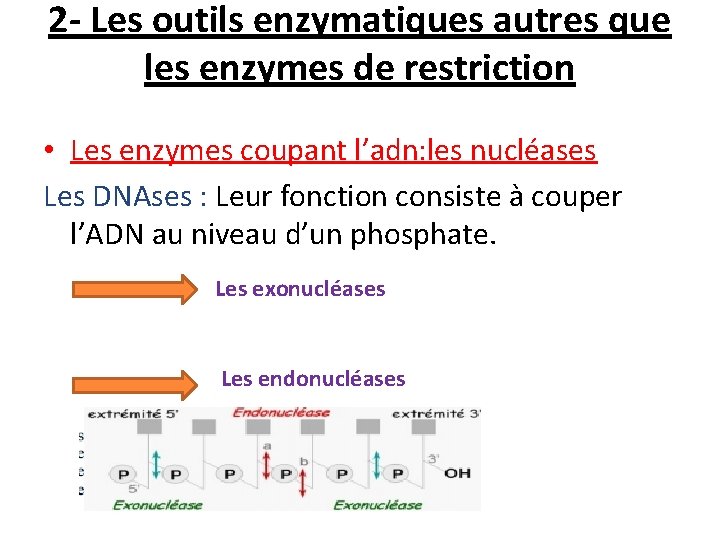
2 - Les outils enzymatiques autres que les enzymes de restriction • Les enzymes coupant l’adn: les nucléases Les DNAses : Leur fonction consiste à couper l’ADN au niveau d’un phosphate. Les exonucléases Les endonucléases

1. Les nucléases Définition : les nucléases dégradent les molécules d'ADN en rompant les liaisons phosphodiesters liant un nucléotide au suivant. Il existe 2 types de nucléases : 1. 1. Exonucléases Ces enzymes éliminent des nucléotides un a partir d'une extrémité de l'ADN. exemple : ü la Bal 31 (obtenue de cultures d'Alieromonas espejana) éliminant des nucléotides apartir des 2 extrémités des 2 brins, ü l'exonuclease 3 (obtenue a partir de cultures d'E. coli) catalysant l'hydrolyse séquentielle des nucléotides d'un ADN a partir d'une extrémité 3' libre.

les exonucléases v exonucléase III : 3’-5’ exonu v. Exonucléase I : 3’- 5’ exonu (ADN simple brin) v. Bal 31: 5’ – 3’ exonu + activité endonu

1. 2. Endonucléases a)a clivages non spécifiques Ces enzymes sont capables de rompre des liaisons phosphodiesters internes. • Exemple: Ø La DNAse 1 extraite du pancréas, coupe préférentiellement âpres une base pyrimidique. Ø La nucléase S 1, extraite de culture d'Aspergillus oryzae dégrade seulement les acides nucléiques monocaténaires.

2. Les polymérases et enzymes apparentées Les polymérases d'acides nucléiques sont des enzymes qui synthétisent un nouveau brin d'ADN complémentaire a une matrice d'ADN ou ARN. 2 types de polymérases sont couramment utilisées en génie génétique : 2. 1. ADNpolymérase I Extraite d'Escherichia coli, elle joue un rôle majeur dans la réparation de l'ADN. L'enzyme se fixe a une région monobrin et synthétise totalement le brin complémentaire, . L'activite polymerasique s'effectue dans le sens 5' vers 3' a partir d'une amorce 3'OH. 2. 2. Transcriptase réverse Codée par les gènes Pol des rétrovirus, cette enzyme transcrit ARN en ADN complémentaire (c. ADN). Comme toutes les polymérases, elle travaille dans le sens 5' vers 3'.

Les polymérases: q activité polymérisante des AN q L’ ADN polymérase I q Le fragment de Klenow q La Taq ploymérase q La transcriptase inverse q L’ARN polymérase

La digestion de la DNA polymérase I par une protéase (subtilisine) donne deux fragments : celui de 76 k. D (fragment de Klenow) possède encore deux des activités catalytiques de la polymérase : 5’→ 3’ polymérase et 3’→ 5’ exonucléase. Ce fragment peut-être utilisé pour synthétiser le deuxième brin à partir d’un ADN simple brin et d’une amorce. .

Le fragment de Klenow est utilisé pour : Øla synthèse du deuxième brin, complémentaire d’un c. DNA ; Øle marquage des extrémités 5’ sortantes du DNA double brin ; Øle marquage du DNA par la technique des amorces aléatoires ; Øle séquençage du DNA par la technique des didésoxynucléotides ; Øla mutagénèse dirigée à partir d’oligonucléotides synthétiques


3. Enzymes modifiant l'ADN De nombreuses enzymes modifient l'ADN par addition ou suppression de groupes chimiques spécifiques. 1 - les enzymes enlevant les groupes phosphates – la phosphatase alcaline : elle retire le phosphate en 5' sur l'ADN, l'ARN et les nucléotides libres. L'elimination des phosphates en 5' empêche toute action des ligases. 2 - les enzymes ajoutant les groupes phosphates – la T 4 polynucleotide kinase : elle transfère le phosphate d'un ATP sur le phosphate en 5' d'un polynucleotide. – les topopisomerases : Les topo isomérases sont capables de changer la conformation de l’ADN. - Les méthylases :

4. Ligases Ce type d'enzyme permet de relier les 2 fragments d'ADN double brins C'est une enzyme capitale puisqu'elle permet la construction de molécules d'ADN recombinées. 4. 1. ADN ligase L'enzyme n'agit que si les 2 ADN sont associes par des extrémités cohésives. Cette ligase assure la formation des liaisons phosphodiesters entre une extrémité 3'OH et une extrémité 5' phosphate. 4. 2. T 4 ADN ligase Elle est capable d'effectuer des ligations entre 2 ADN a bouts francs.

LA CARTE DE RESTRICTION • Une carte de restriction montre l'arrangement linéaire des sites de reconnaissance de l’endonucléase de restriction le long d’une molécule d’ADN.

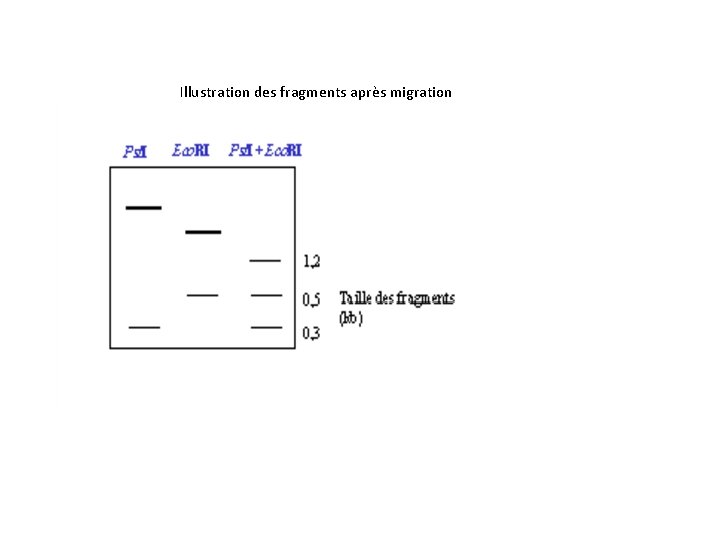
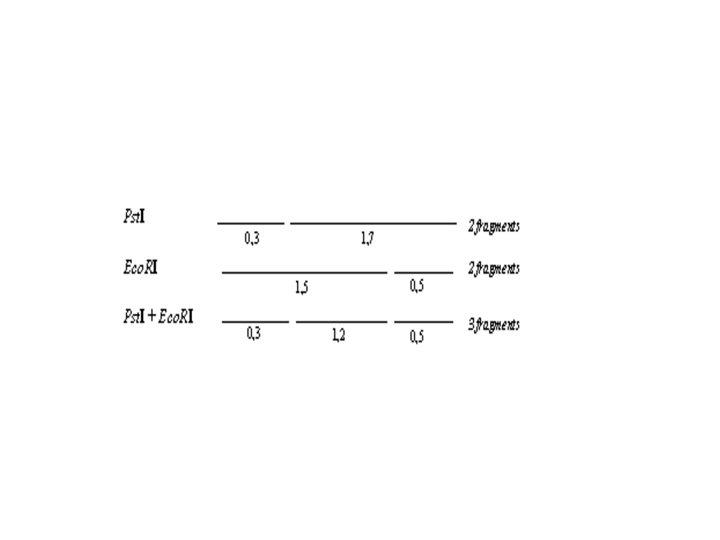
• Carte de restriction de l’ADN. Les enzymes de restriction coupent l’ADN au niveau de séquences parfaitement définies. Il est donc possible d’établir une véritable carte d’un gène donné. Cette carte porte le nom de carte de restriction. En pratique, l’ADN à étudier est réparti en différentes fractions. Chaque fraction est traitée par une enzyme de restriction ou un couple d’enzymes de restriction. Ainsi dans l’illustration ci-dessous. Dans le puits 1, on dépose l’ADN digéré par Pst. I seule; dans le puits 2, on dépose l’ADN digéré par Eco. RI seule et dans le puits 3, l’ADN digéré par Eco RI et Pst. I. La détection des différents fragments est réalisée à l’aide d’une sonde marquée du gène étudié.

Illustration des fragments après migration



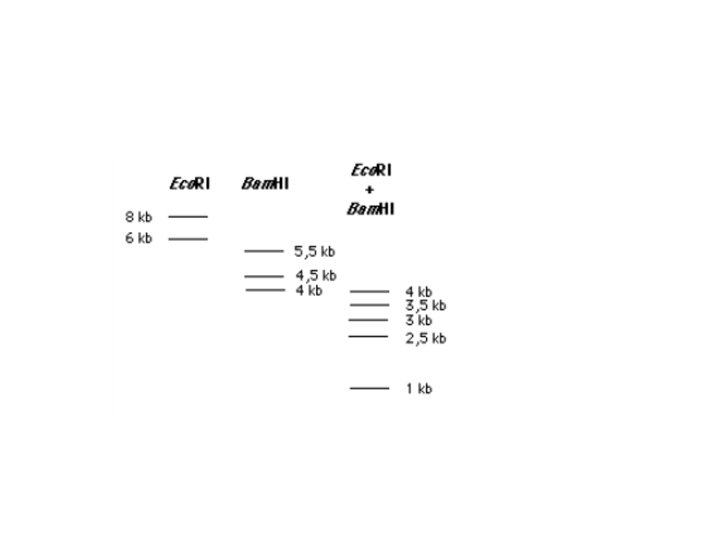
Exercice 01: Un des plasmides recombinants contenant le gène M (appelé p. BM 1) est digéré par les enzymes de restriction Bam HI et Eco RI. Après migration et séparation des fragments d'ADN sur gel d'agarose puis coloration au bromure d'éthidium, on obtient les profils de restriction suivants: Donnez la carte de restriction du plasmide recombinant p. BM 1.



b) a clivages spécifiques : enzymes de restriction v Elles coupent de manière définie et reproductible l'ADN bicentenaire. v Ce sont des enzymes produites par de très nombreuses bactéries au moment des infections lysogeniques v. L'analyse de leurs coupures de l'ADN montre qu'elles se font en des sites spécifiques. v Plus de 1200 différentes enzymes de restriction ont été caractérisées et elles sont classes en 3 types :

II- Séparation des acides nucléiques Ø Electrophorèse













Exercice 1 Soient les enzymes de restriction Bam. HI, Pst I, Xho. I et Mbo I dont les sites reconnus sont : • Bam. HI : 5' G/GATCC 3' ; • Pst I : 5' CTGCA/G 3' ; • Xho. I : 5' C/TCGAG 3' ; • Mbo. I : 5' /GATC 3'. Recopier la séquence de l’ADN et encadrer les sites de restriction en indiquant la position des coupures. 5’ ATACGGGATCCGAGCTCTCGATCGTCTGCAGAAATTCC 3’ TATGCCCTAGGCTCGAGAGCTAGCAGACGTCTTTAAGG 5’

Exercice 2: Un fragment d’ADN linéaire 1. Coupé par l’enzyme Eco. RI donne un fragment de 8 Kb, un de 4 Kb et un de 3 Kb 2. coupé par l’enzyme Sma. I donne un fragment de 9 Kb et un de 6 Kb. 3. Coupé par les deux enzymes Eco. RI et Sma. I produit un fragment de 6 Kb, un de 4 Kb, un de 3 Kb et un de 2 Kb.

Exercice 3: Un plasmide recombinant contenant le gène M (appelé p. BM 1) est digéré parles enzymes de restriction Bam HI et Eco RI. Après migration et séparation des fragments d'ADN sur gel d'agarose puis coloration au bromure d'éthidium, on obtient les profils de restriction suivants: • Donnez la carte de restriction du plasmide recombinant p. BM 1.

III- Détection, caractérisation et identification des AN Ø Marquage Ø Hybridation moléculaire Ø Southern blot

• Certains types d’expériences (Southern, northern, hybridation in situ, . . . ) nécessitent de visualiser une hybridation. Pour cela, il est nécessaire de marquer un acide nucléique (ADN ou ARN simple brin) qui prendra le nom de sonde.


Les différents types de marquage - Marquage radioactif dit chaud - Marquage froid dit biochimique

1 -Marquage radioactif L’atome ou le groupement incorporé est RADIOACTIF (ajout d’un radio isotope tel que 32 P, 35 S, 3 H). Marquage interne Marquage externe

a- Marquage Interne : * Nick translation ou déplacement de cassures Utilisation de 2 enzymes : - DNAse I pour générer quelques coupures simple brin dans le fragment d'intérêt -DNA pol I pour dégrader l'ADN dans sens 5'-3' au niveau de ces coupures et repolymériser en présence d'un nucléotide chaud. L’ADN double brin traité par la DNAse I est clivé au hasard. La réparation des coupures réalisées par la Dn. Ase I nécessite l'action de l'ADN polymérase I en présence de désoxynucléosides triphosphates marqués au phosphore radioactif (32 P).

*amorçage au hasard (Random Priming) • un ou plusieurs des oligonucléotides s’hybrident avec la séquence en 5’ du fragment d’intérêt (ADN simple brin) et servent d’amorces pour la synthèse d’un deuxième brin.

B- Marquage externe Ø marquage en 5' T 4 polynucléotide kinase ajoute un P 32 en 5' (sonde peu radioactive) Ø marquage en 3‘ v avec une ADN polymérase: ADN Pol I ou T 4, Taq pol (PCR) (sondes très radioactives) v avec une exonucléase v avec une terminal transférase

Vecteurs et Clonage moléculaire

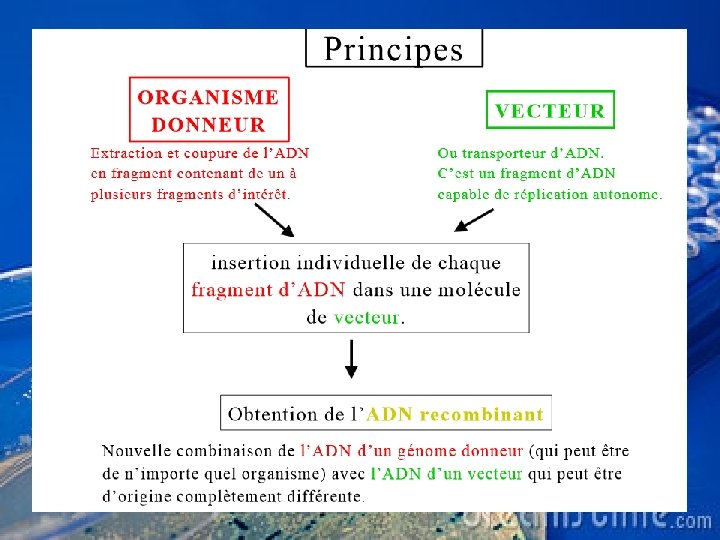
Clonage moléculaire Définition: Cloner un fragment d'ADN consiste a: • isoler physiquement ce fragment et l’ inséré dans un vecteur pour augmenter leur nombre de copie U n clone est un ensemble de cellules qui ont toutes le même génome et qui ont une origine commune

Clonage moléculaire Principe: Le clonage consiste a: 1) insérer un fragment d'ADN étranger a cloner (insert) dans un vecteur de manière a obtenir un vecteur recombinant. 2) introduire le vecteur recombinant dans une cellule hôte. 3) amplifier le vecteur recombinant par division de la cellule hôte, afin d'obtenir un clone recombinant.

Clonage moléculaire

Clonage moléculaire Ø comment obtenir l’ADN de l’organisme donneur? Ø Qu’est ce qu’une enzyme de restriction? ØQuels sont les vecteurs de clonage? ØComment l’ADN d’intérèt et le vecteur sont –ils liés? ØLes cellules hôtes? ØComment l’ADN recombinant est introduit dans une cellules hôtes? ØLes intérêts du clonage?

Øcomment obtenir l’ADN de l’organisme donneur?

Qu’est ce qu’une enzyme de restriction?

ØQuels sont les vecteurs de clonage?

Un vecteur est une séquence d'ADN permettant la propagation, la sélection d'une séquence d'ADN d'intérêt.

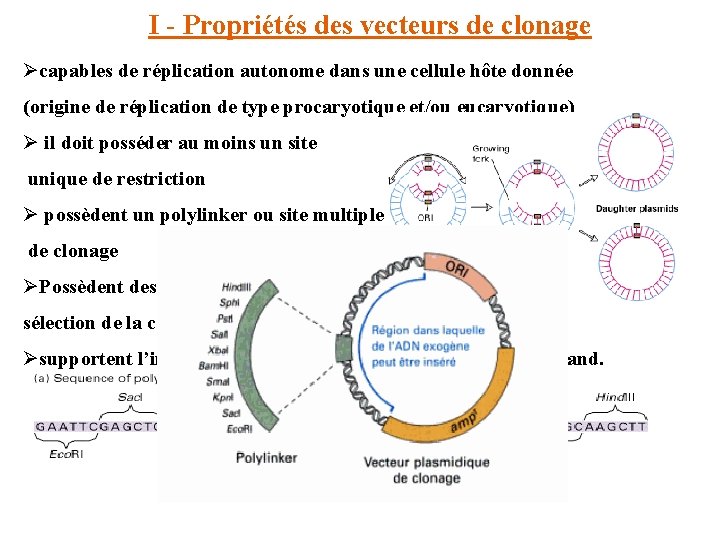
I - Propriétés des vecteurs de clonage Øcapables de réplication autonome dans une cellule hôte donnée (origine de réplication de type procaryotique et/ou eucaryotique) Ø il doit posséder au moins un site unique de restriction Ø possèdent un polylinker ou site multiple de clonage ØPossèdent des propriétés permettant la sélection de la cellule hôte (résistance ATB) Øsupportent l’insertion d’un fragment d’ADN plus ou moins grand.

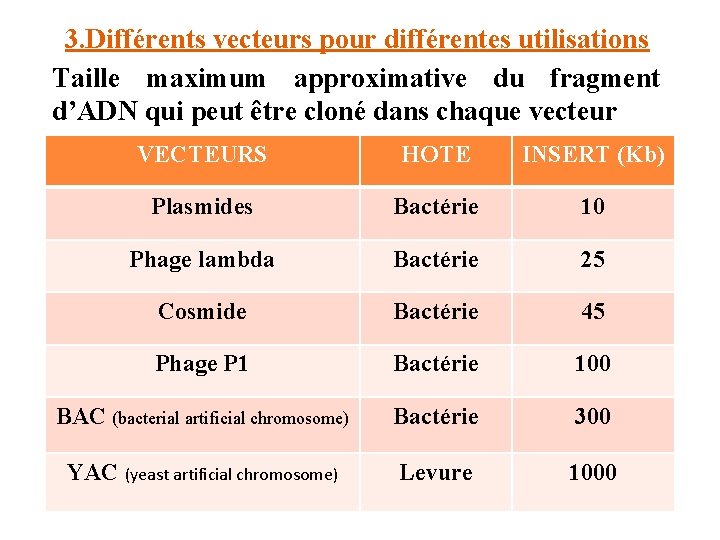
3. Différents vecteurs pour différentes utilisations Taille maximum approximative du fragment d’ADN qui peut être cloné dans chaque vecteur VECTEURS HOTE INSERT (Kb) Plasmides Bactérie 10 Phage lambda Bactérie 25 Cosmide Bactérie 45 Phage P 1 Bactérie 100 BAC (bacterial artificial chromosome) Bactérie 300 YAC (yeast artificial chromosome) Levure 1000

4. Les plasmides comme vecteurs de clonage ØMolécule d’ADN de taille réduite, d'origine bactérienne ØMolécule d’ADN circulaire – taille 2 kb à 5 kb ØPeut accepter jusqu’à 10 kb d’ADN exogène ØPeut être considérée comme un minichromosome capable de réplication autonome ØConfère la résistance à un ou plusieurs antibiotiques= sélection

4. Les plasmides comme vecteurs de clonage ØMolécule d’ADN de taille réduite, d'origine bactérienne, extrachromosomique (épisome) ØMolécule d’ADN circulaire – taille 2 kb à 5 kb ØPeut accepter jusqu’à 10 kb d’ADN exogène ØPeut être considérée comme un minichromosome capable de réplication autonome ØConfère la résistance à un ou plusieurs antibiotiques= sélection

Les plasmides de première génération: (exp: Col. E 1, Psc 101, RSF 2124) C'est avec ces plasmides qu'ont été effectué les 1 er clonages (clonage génique). se sont des plasmides à l’état naturel, non modifies au laboratoire. Mais ils avaient besoin d'être améliorés. •

Plasmide de deuxième génération: (exp: la serie p. BR 312 à p. BR 322) dérivé de plasmides naturels: plasmides artificiels Le plasmide p. BR 322 est constitué de 4, 4 kb et possède deux gènes de résistance : un pour la tétracycline (Tc. R), l’autre pour l’ampicilline (Ap. R) il possède, 20 sites uniques pour les endonucléases de restriction dont 11 localisés sur deux gènes de résistance. •

Plasmide de troisième génération: (exp: famille p. UC) dérivé de p. BR 322, plasmide de 2 nd génération. Leur plus petite taille (2, 6 kb) permet d'insérer un fragment d'ADN étranger plus grand. Possède les gènes de résistance à l’ampicilline et Lac. Z

ØComment l’ADN d’intérèt et le vecteur sont –ils liés? ØLes cellules hôtes? ØComment l’ADN recombinant est introduit dans une cellules hôtes? ØLes intérêts du clonage?

Utilisation d’un vecteur 1 - préparation du vecteur: coupure du vecteur à l’endroit exact ou l’on désire insérer la séquence à étudier.

2 - traitement par phosphatase alcaline

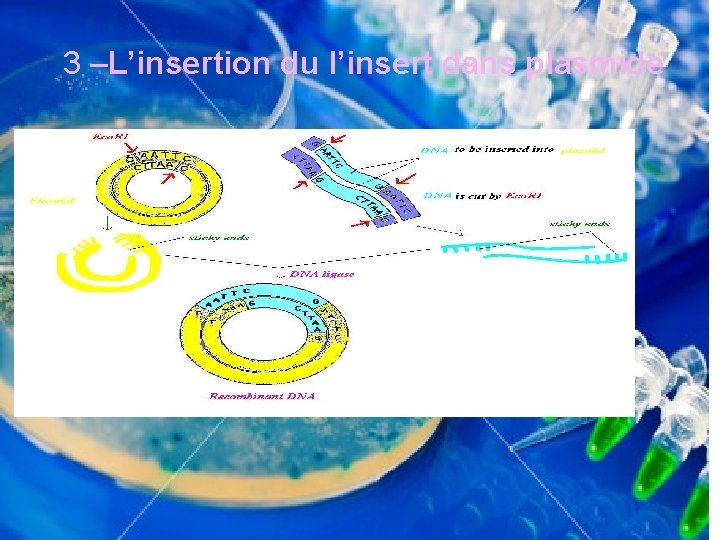
3 –L’insertion du l’insert dans plasmide

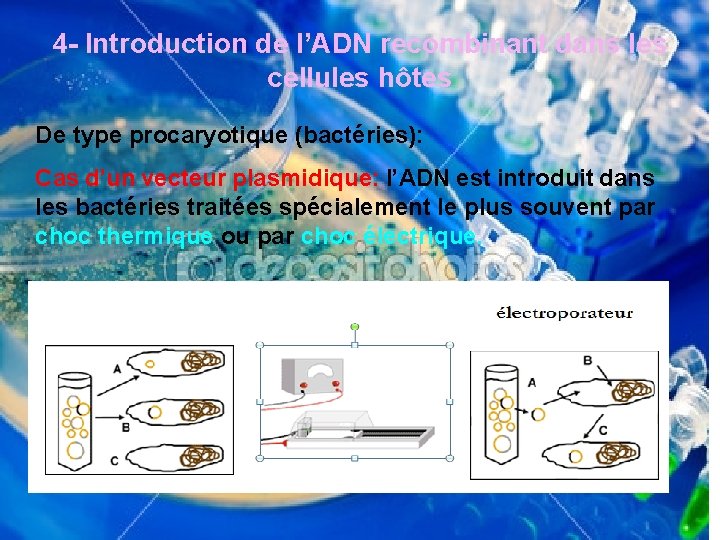
4 - Introduction de l’ADN recombinant dans les cellules hôtes De type procaryotique (bactéries): Cas d’un vecteur plasmidique: l’ADN est introduit dans les bactéries traitées spécialement le plus souvent par choc thermique ou par choc électrique.

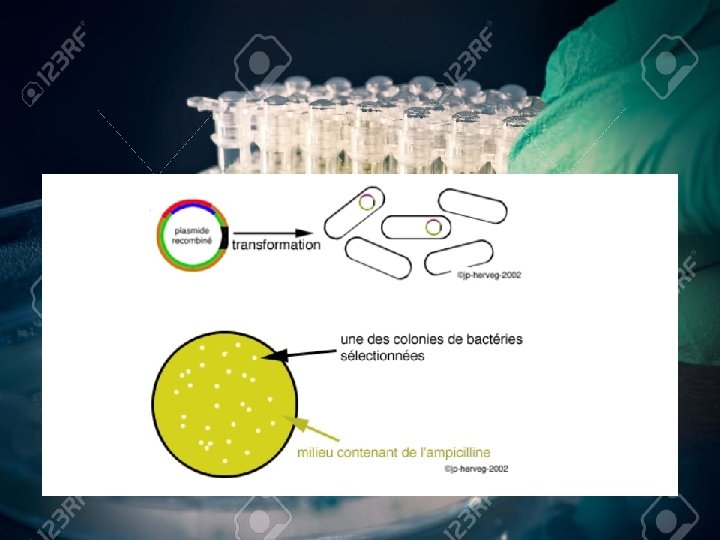
5 - la sélection des bactéries transformés:

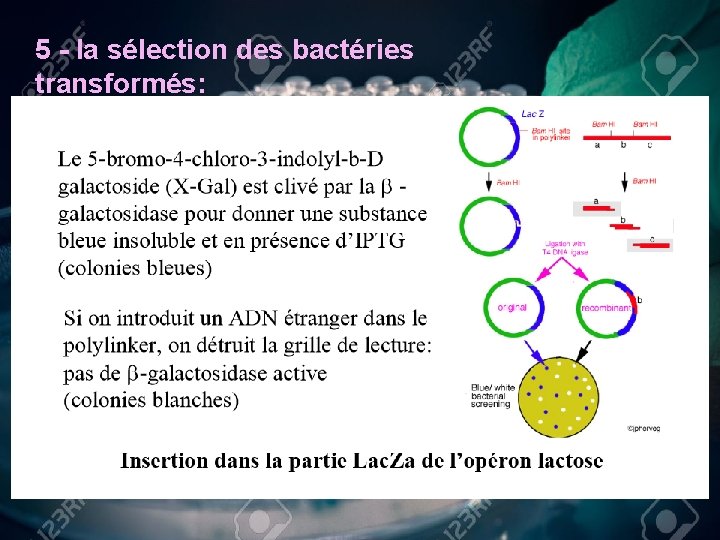
5 - la sélection des bactéries transformés:

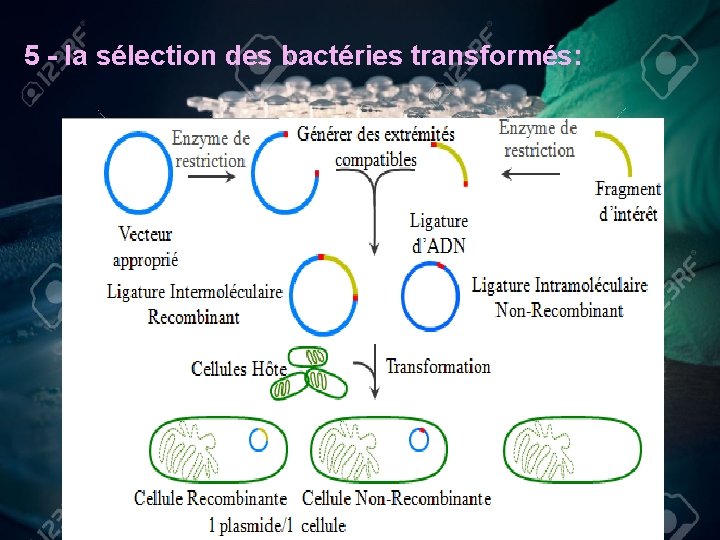
5 - la sélection des bactéries transformés:


ØLe taux de transformation généralement très faible. des bactéries est ØSi on utilise un gène de résistance dans plasmide on obtient une sélection séparer les bactéries comportant des plasmides et les bactéries sans plasmides car ces dernières sont tuées. Ø cette méthode permet pas de distinguer les bactéries avec plasmides intacts des bactéries avec plasmides recombinants. ØOn utilise pour cela une résistance à un second antibiotique , ou plasmide a un gène code (Lac. Z) pour un protéine β galactosidase.

5 - la sélection des bactéries transformés:



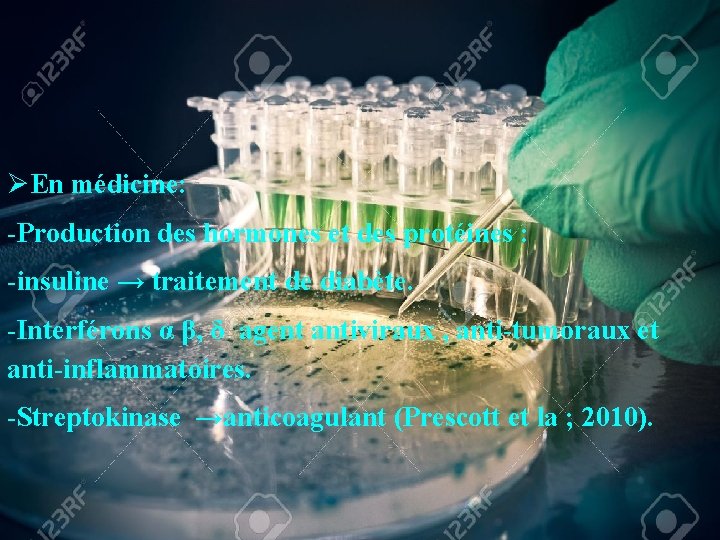
ØEn médicine: -Production des hormones et des protéines : -insuline → traitement de diabète. -Interférons α β, δ agent antiviraux , anti-tumoraux et anti-inflammatoires. -Streptokinase →anticoagulant (Prescott et la ; 2010).




