LE SEQUENCAGE OPTIMISATION DE LA RACTION DE SEQUENCE
































- Slides: 66

LE SEQUENCAGE








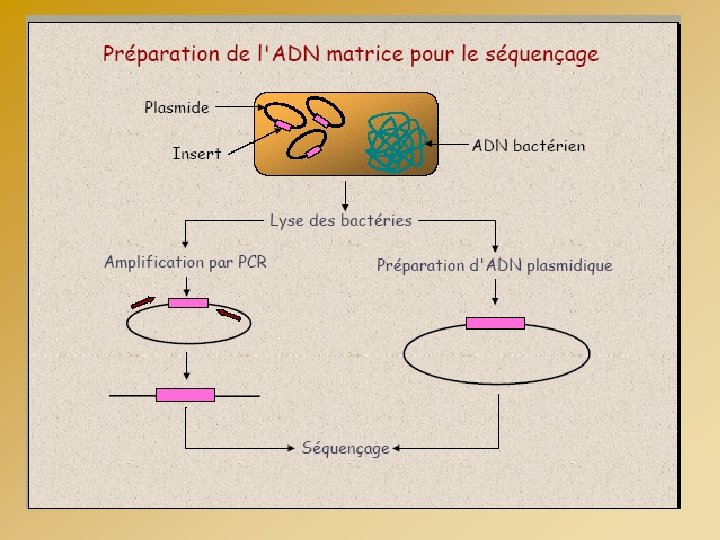
OPTIMISATION DE LA RÉACTION DE SEQUENCE QUALITE DE LA MATRICE - Produit de PCR : Purification (élimination des amorces, d. NTP) - Kit de purification ( colonnes Qiagen – Sigma …) - Traitement à l’Exo. SAP Mélange de 2 enzymes : Exonucléase 1 (élimine les amorces) Phosphatase Alcaline (élimine les d. NTP) - Plasmide : Purification - Kit de purification - Quantification - Dépôt sur gel d’agarose avec marqueur de taille - Nanodrop

OPTIMISATION DE LA RÉACTION DE SEQUENCE REACTION DE SEQUENCE - Instructions du fournisseur - Quantité d’ADN - Volume du mélange réactionnel - Quantité d’amorce - Cycles 94°C 3 min (96°C 10 sec- 50°C 5 sec - 60°C 4 min) 25 x Volume total = 25 µl ADN (PCR) = 5 ng Amorce = 3 pmoles Tampon 5 X = 2, 5 µl Kit Big Dye = 1 µl H 2 O = qsp 25 µl Kit Big Dye contient : - ADN Polymérase - Tampon - Mg. Cl 2 - d. NTP - dd. NTP*

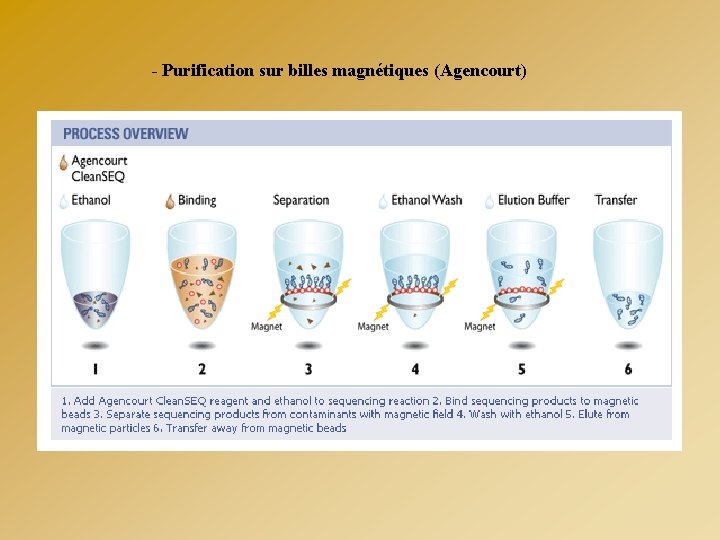
OPTIMISATION DE LA RÉACTION DE SEQUENCE PRECIPITATION DES SEQUENCES - Instructions du fournisseur - Précipitation éthanolique Na. Ac 3 M p. H 4, 6 + Et. OH 95% Lavage Et. OH 70% - Purification sur billes magnétiques (Agencourt)

- Purification sur billes magnétiques (Agencourt)

SEQUENCEUR CAPILLAIRE





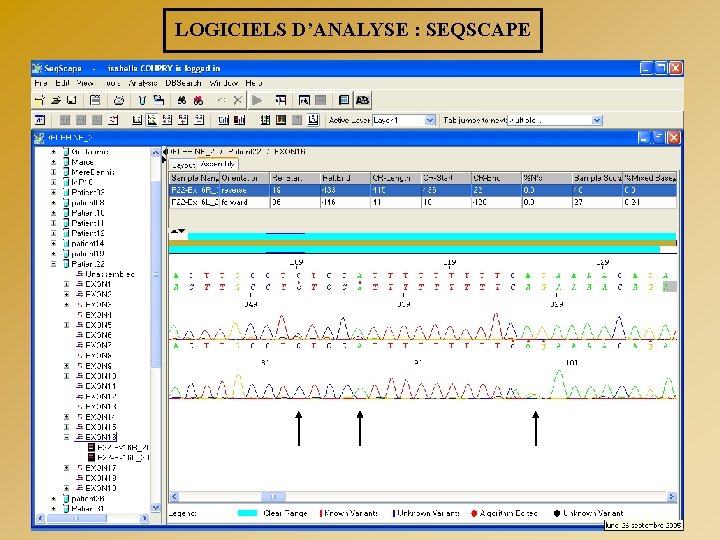
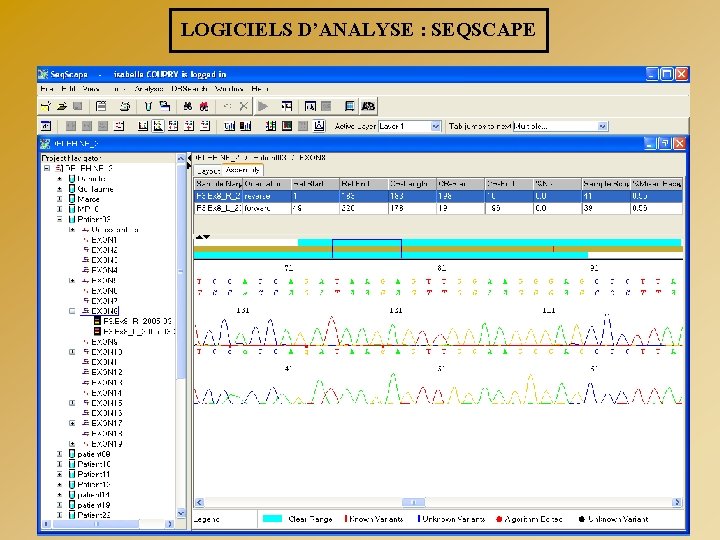
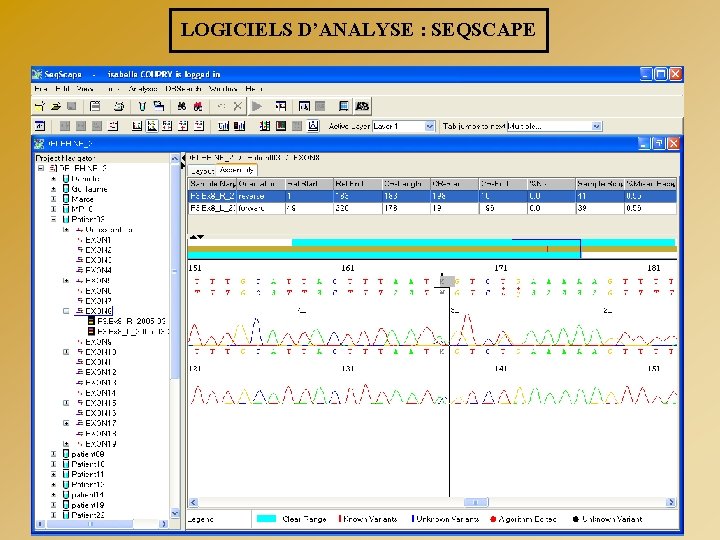
LOGICIELS D’ANALYSE : SEQSCAPE

LOGICIELS D’ANALYSE : SEQSCAPE

LOGICIELS D’ANALYSE : SEQSCAPE

LOGICIELS D’ANALYSE : SEQSCAPE

LOGICIELS D’ANALYSE : SEQSCAPE

LOGICIELS D’ANALYSE : SEQSCAPE

POURQUOI ANALYSER UNE SEQUENCE ? - A partir de la structure d’un gène, déterminer la séquence codante et en déduire la séquence protéique - Trouver des homologies : - Au niveau des gènes d’une même famille - Au niveau d’un même gène dans différentes espèces - Trouver des différences : - Polymorphismes - Mutations Alignement de séquences (Logiciel Seq. Scape)

LOGICIELS D’ANALYSE : SEQSCAPE

LOGICIELS D’ANALYSE : SEQSCAPE

LOGICIELS D’ANALYSE : SEQSCAPE

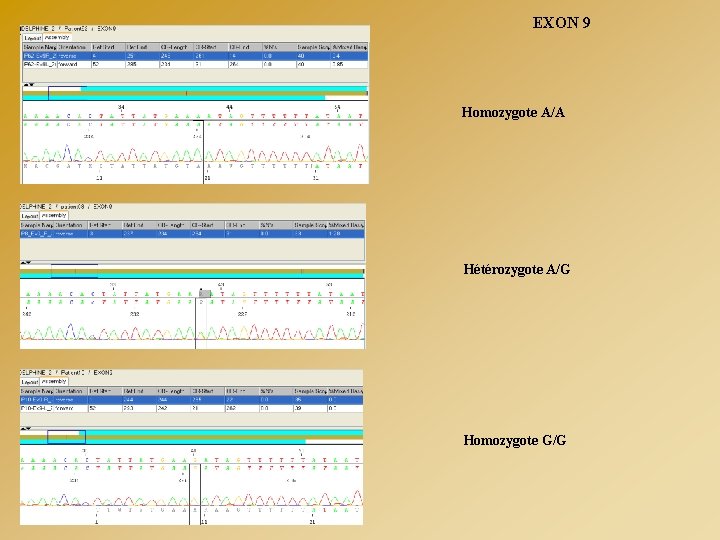
EXON 9 Homozygote A/A Hétérozygote A/G Homozygote G/G


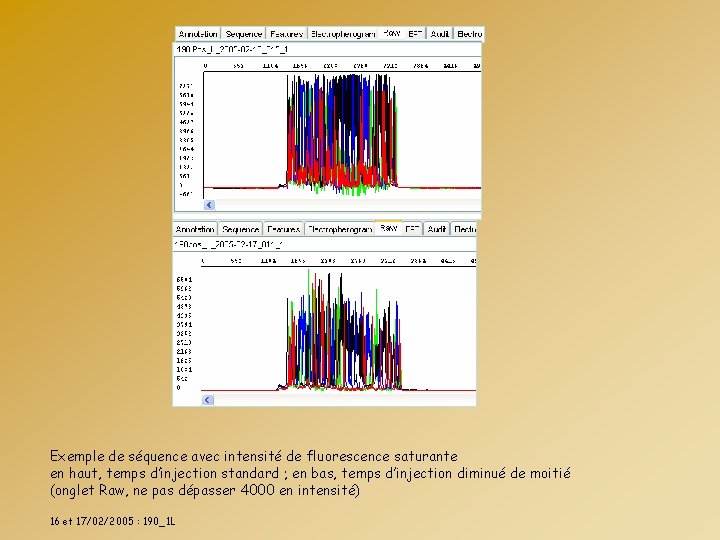
Exemple de séquence avec intensité de fluorescence saturante en haut, temps d’injection standard ; en bas, temps d’injection diminué de moitié (onglet Raw, ne pas dépasser 4000 en intensité) 16 et 17/02/2005 : 190_1 L

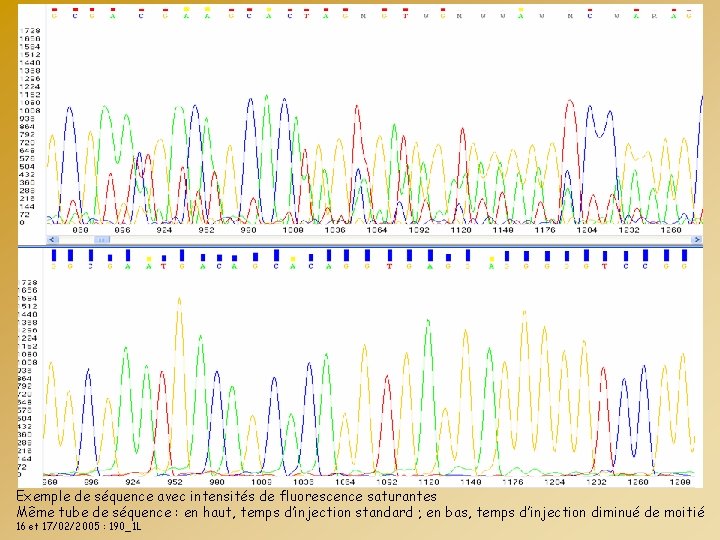
Exemple de séquence avec intensités de fluorescence saturantes Même tube de séquence : en haut, temps d’injection standard ; en bas, temps d’injection diminué de moitié 16 et 17/02/2005 : 190_1 L

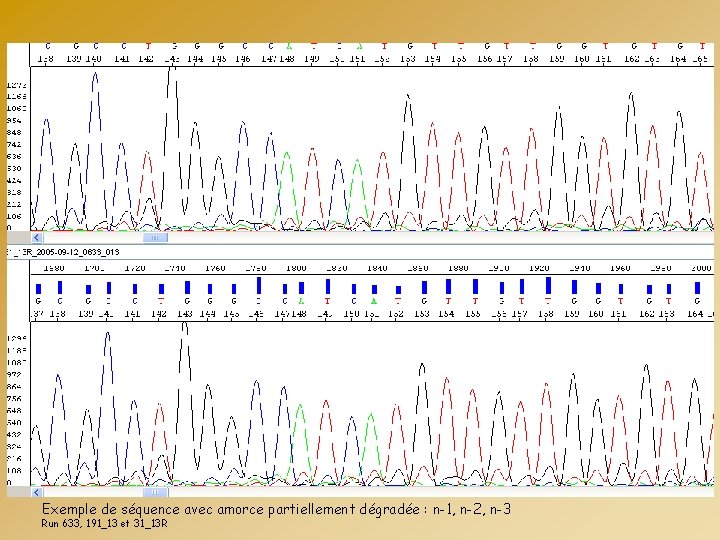
Exemple de séquence avec amorce partiellement dégradée : n-1, n-2, n-3 Run 633, 191_13 et 31_13 R

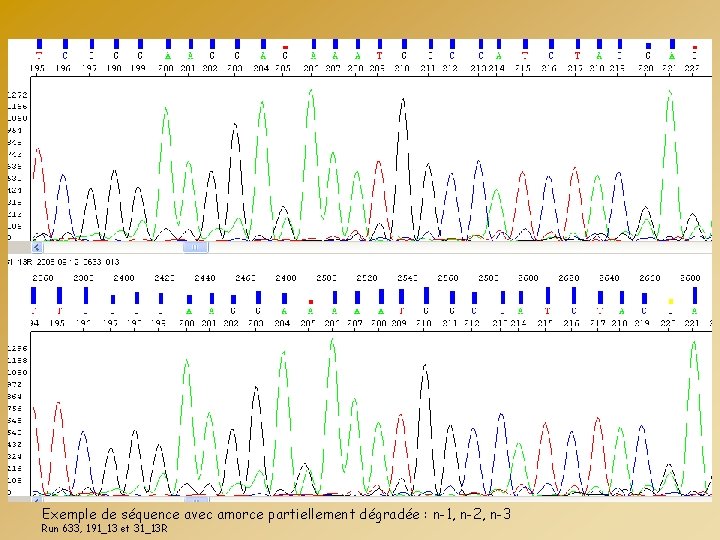
Exemple de séquence avec amorce partiellement dégradée : n-1, n-2, n-3 Run 633, 191_13 et 31_13 R

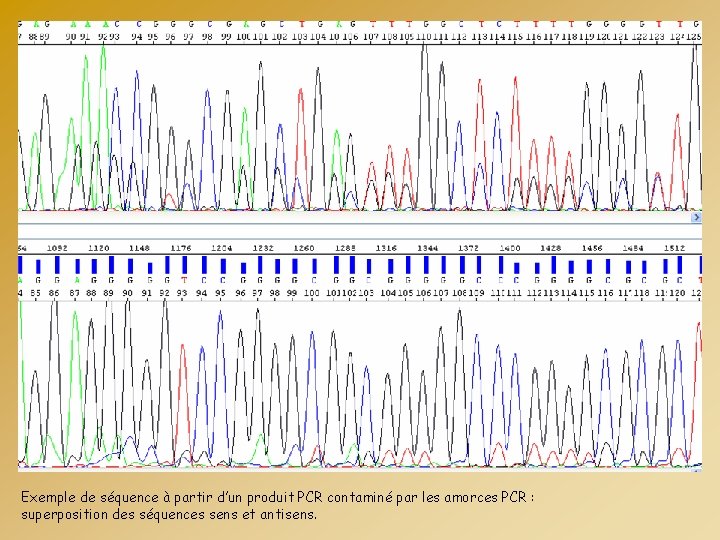
Exemple de séquence à partir d’un produit PCR contaminé par les amorces PCR : superposition des séquences sens et antisens.

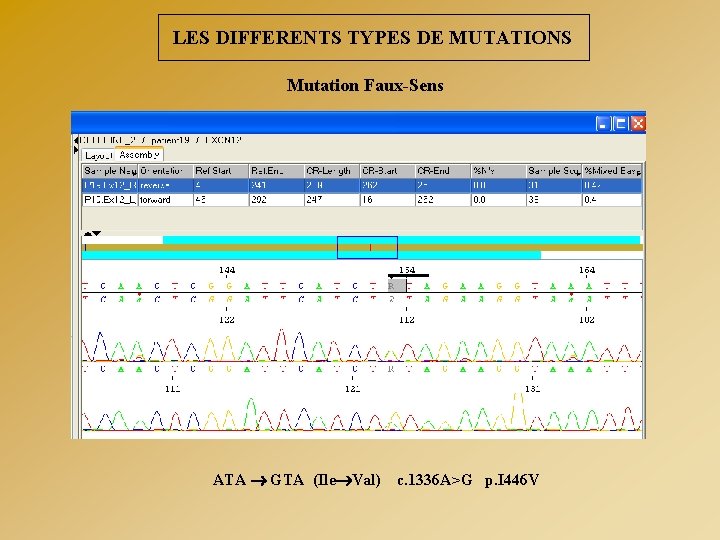
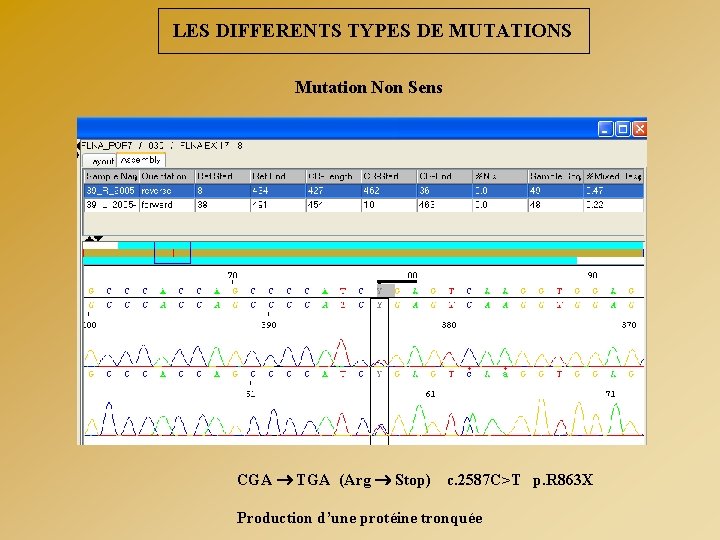
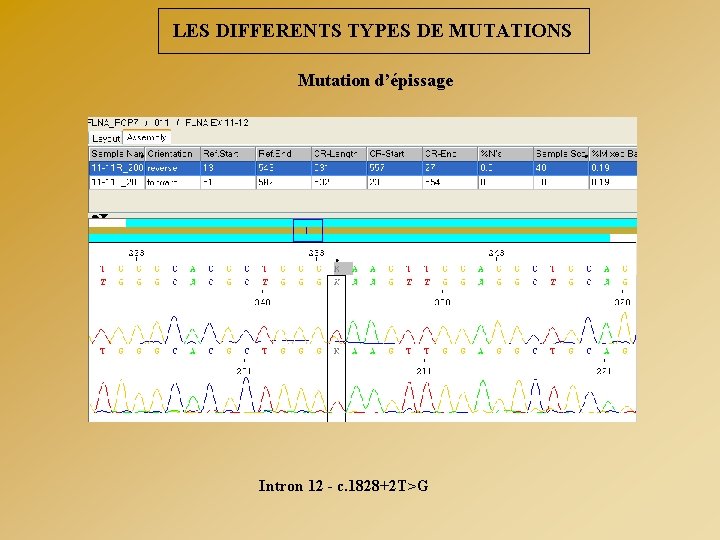
LES DIFFERENTS TYPES DE MUTATIONS - Mutation Faux-Sens Modifie l’acide aminé - Vérifier s’il s’agit d’un acide aminé conservé - Vérifier si la classe de l’acide aminé change - Mutation Non Sens L’acide aminé est remplacé par un Stop - Stop prématuré production d’une protéine tronquée - Mutation d’épissage - Au niveau du site donneur - Au niveau du site accepteur - Insertion/Délétion Décalage du cadre de lecture - Stop prématuré production d’une protéine tronquée - Mutation silencieuse Ne modifie pas l’acide aminé - Mutation intronique

LES DIFFERENTS TYPES DE MUTATIONS Mutation Faux-Sens ATA GTA (Ile Val) c. 1336 A>G p. I 446 V

LES DIFFERENTS TYPES DE MUTATIONS Mutation Non Sens CGA TGA (Arg Stop) c. 2587 C>T p. R 863 X Production d’une protéine tronquée

LES DIFFERENTS TYPES DE MUTATIONS Délétion/Insertion Séquence normale : TCA GAC TTT GTG GAG GCT Séquence mutée TCA GAC TGT GGA GGC TAT Séquence normale : TCA GAC TTT GTG GAG GCT ATC GGG GAC GTG GGC Séquence mutée : TCA GAC TGT GGA GGC TAT CGG GGA CGT GGG CAC GCT GGG CTT CTC GGT GGA AGG GCC ATC GCA GGC TAA Délétion de 2 pb : c. 1790_1 del. TT p. Phe 597 Cysfs. X 23

LES DIFFERENTS TYPES DE MUTATIONS Mutation d’épissage Intron 12 - c. 1828+2 T>G

SEQUENCE CONSENSUS DU SITE DONNEUR D’EPISSAGE %A 30 %T 20 %C 30 %G 19 EXON 40 64 9 7 13 12 43 12 6 9 12 73 A G INTRON 0 0 62 68 0 100 6 12 0 0 2 9 100 0 29 12 G T A A 9 17 39 5 63 22 2 12 21 84 9 18 G T 24 26 29 20

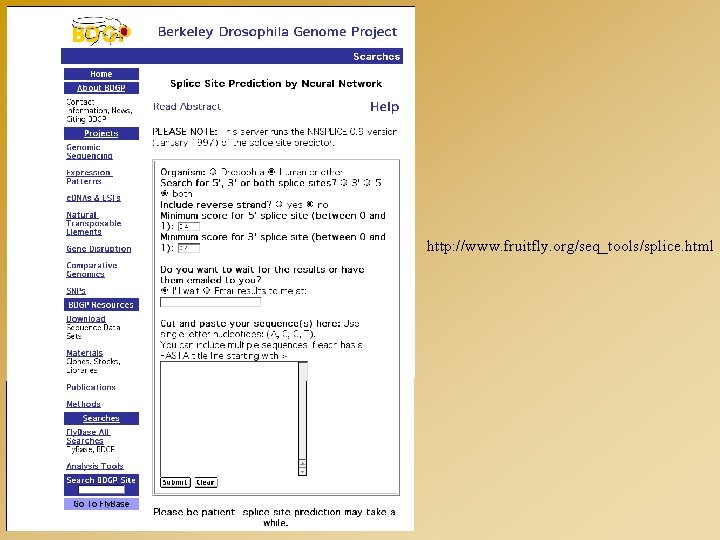
http: //www. fruitfly. org/seq_tools/splice. html



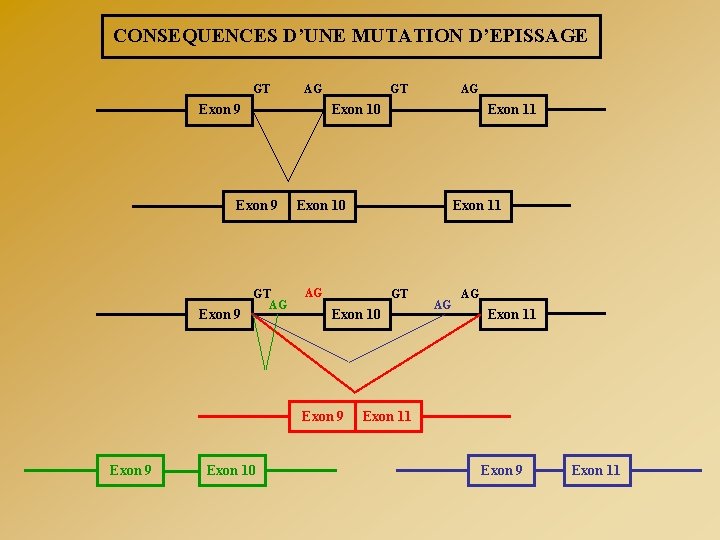
CONSEQUENCES D’UNE MUTATION D’EPISSAGE GT AG Exon 9 GT AG Exon 10 Exon 9 GT Exon 11 Exon 10 AG GT Exon 9 Exon 10 AG Exon 11

LOGICIELS D’ANALYSE : SEQUENCE NAVIGATOR - Mise en évidence de mutations A G Modification du site accepteur d’épissage

SEQUENCE CONSENSUS DU SITE ACCEPTEUR D’EPISSAGE %A 15 10 10 15 %T 51 44 50 53 %C 19 25 31 21 %G 15 21 10 10 Y Y 6 60 24 10 Y 15 49 30 6 Y 11 49 33 7 Y 19 45 28 9 Y 12 45 36 7 Y 3 57 36 7 Y INTRON 10 25 4 100 58 29 31 0 28 22 65 0 5 24 1 0 Y N Y A 0 0 0 100 G EXON 22 17 8 37 18 22 52 25 G Polypyrimidine track (Y = T ou C; N = n’importe quelle base)



CONSEQUENCES D’UNE MUTATION D’EPISSAGE GT AG Exon 9 GT AG Exon 10 Exon 11 Exon 10 AG GT Exon 10 Exon 9 AG Exon 10 Exon 9 GT AG AG Exon 11 Exon 9 Exon 11

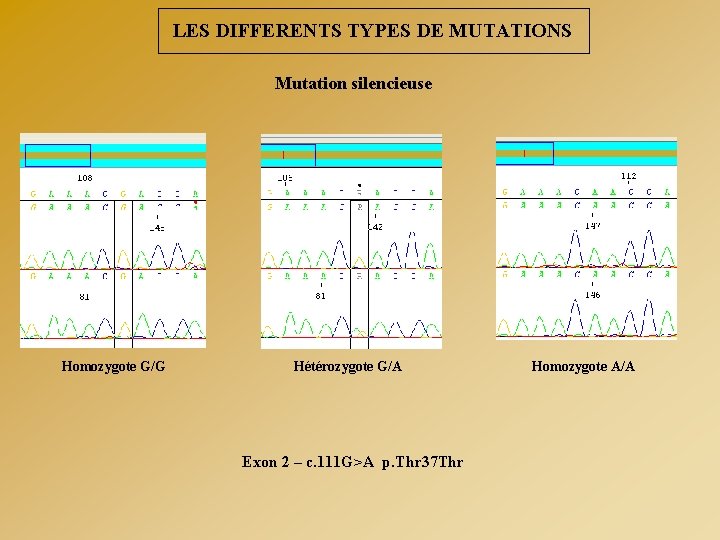
LES DIFFERENTS TYPES DE MUTATIONS Mutation silencieuse Homozygote G/G Hétérozygote G/A Exon 2 – c. 111 G>A p. Thr 37 Thr Homozygote A/A

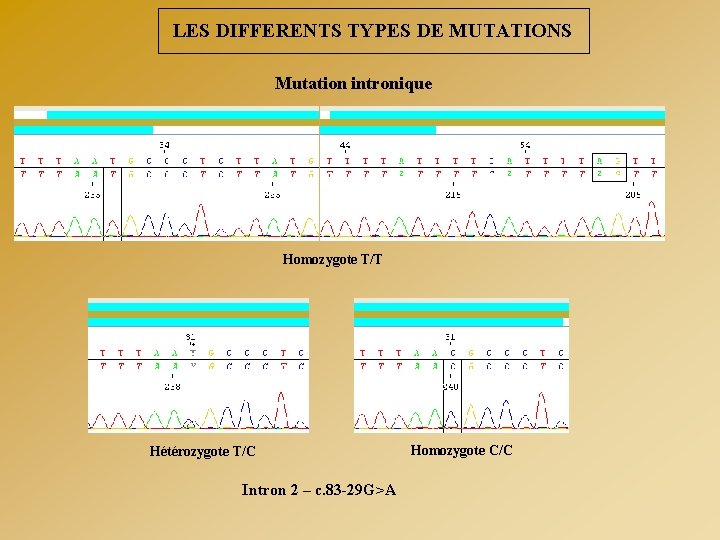
LES DIFFERENTS TYPES DE MUTATIONS Mutation intronique Homozygote T/T Hétérozygote T/C Intron 2 – c. 83 -29 G>A Homozygote C/C

LES DIFFERENTS TYPES DE MUTATIONS -Vérifier la présence de la mutation chez les parents du malade et d’autres membres de la famille - Mutation Faux-Sens - Vérifier s’il s’agit d’un acide aminé conservé - Vérifier si la classe de l’acide aminé change - Mutation Non Sens - Stop prématuré production d’une protéine tronquée - Vérification par Western Blot - Mutation d’épissage - Vérification par RT-PCR - Insertion/Délétion Décalage du cadre de lecture - Stop prématuré production d’une protéine tronquée - Vérification par Western Blot - Mutation silencieuse ou intronique - Vérifier s’il s’agit d’un polymorphisme - Répertorié dans les banques de données - Tester 50 à 100 individus contrôle


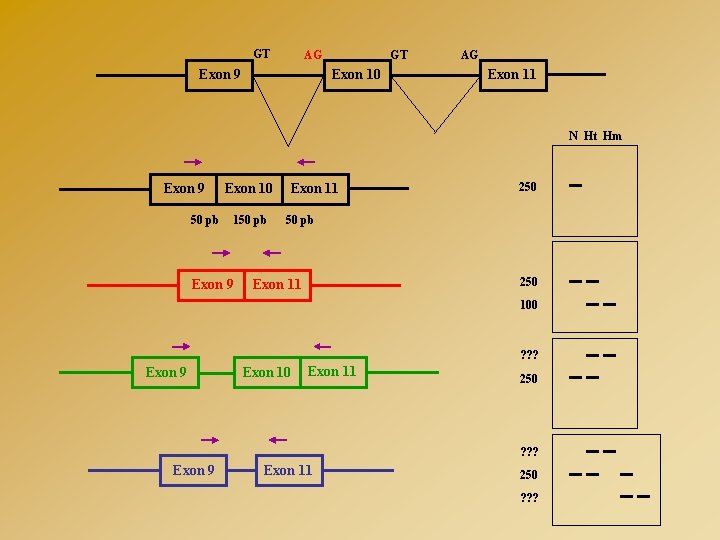
GT GT AG Exon 9 Exon 10 AG Exon 11 N Ht Hm Exon 9 50 pb Exon 10 150 pb Exon 9 Exon 11 250 50 pb 250 Exon 11 100 ? ? ? Exon 9 Exon 10 Exon 11 250 ? ? ? Exon 9 Exon 11 250 ? ? ?

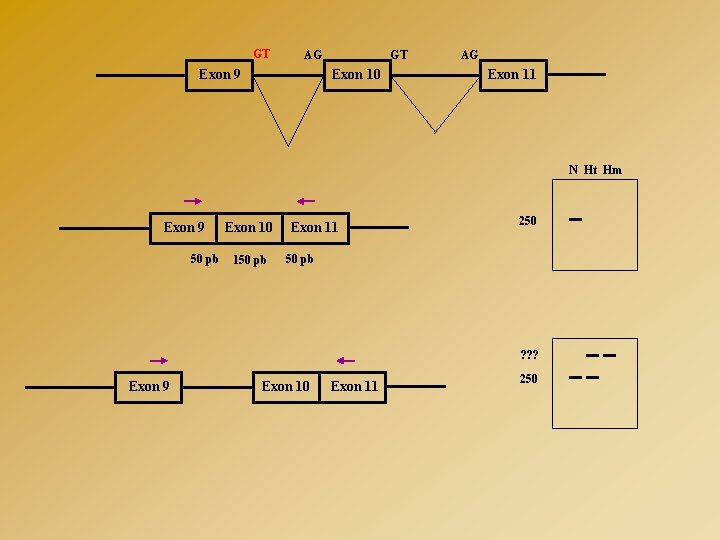
GT GT AG Exon 9 Exon 10 AG Exon 11 N Ht Hm Exon 9 50 pb Exon 10 150 pb Exon 11 250 50 pb ? ? ? Exon 9 Exon 10 Exon 11 250

ANALYSE DE MARQUEURS MICROSATELLITES LOGICIEL GENEMAPPER

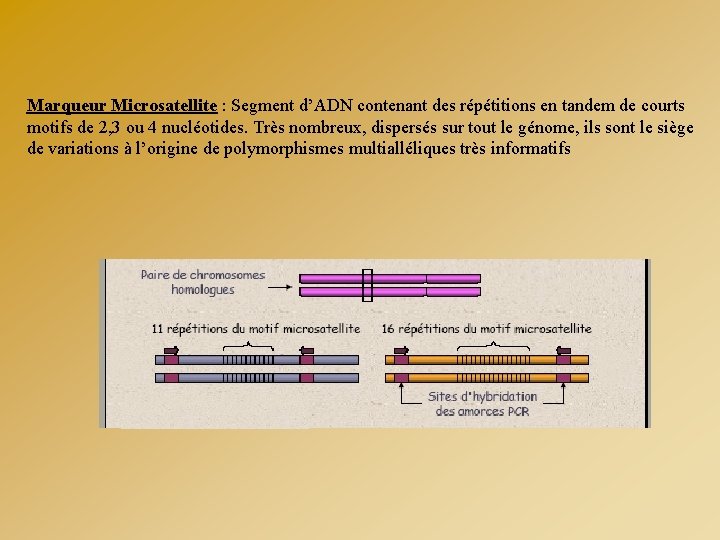
Marqueur Microsatellite : Segment d’ADN contenant des répétitions en tandem de courts motifs de 2, 3 ou 4 nucléotides. Très nombreux, dispersés sur tout le génome, ils sont le siège de variations à l’origine de polymorphismes multialléliques très informatifs

LOGICIELS D’ANALYSE : GENE MAPPER Analyse de marqueurs microsatellites - PCR : une des amorces marquée par un fluorochrome (6 FAM) - Produit de PCR fluorescent - Mélange du produit avec un marqueur de taille (35 à 350 pb) marqué par un autre fluorochrome - Séparation des fragments sur séquenceur capillaire - Détection des fragments fluorescents - Analyse des données

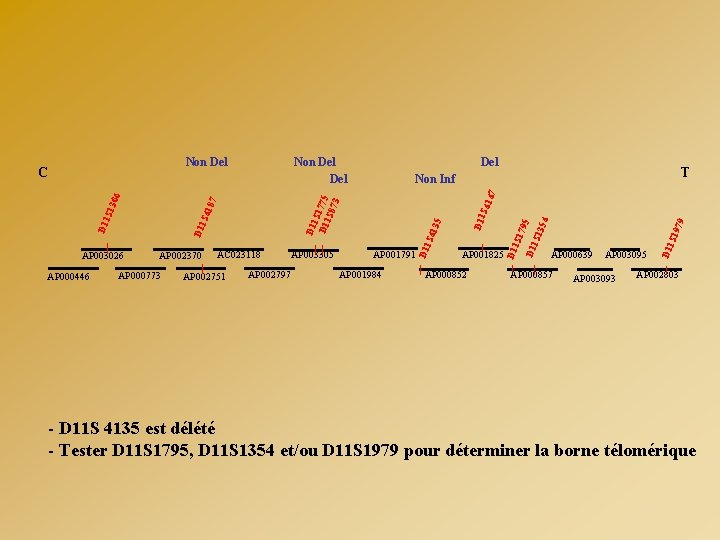
LOGICIELS D’ANALYSE : GENE MAPPER Exemple : - Mise en évidence d’une délétion sur le chromosome 11 chez un patient par FISH - Déterminer les bornes de la délétion par analyse des marqueurs microsatellites de la région - Choix de 5 marqueurs - Réalisation des PCR sur l’ADN du patient et de ses parents - Séparation des fragments sur séquenceur capillaire - Analyse par Gene. Mapper AP 000446 AP 000773 AP 002751 AP 003305 AP 002797 D 11 35 S 41 AP 001791 AP 001984 AP 001825 AP 000852 D 11 S 1 D 11 795 S 13 54 AC 023118 D 11 AP 002370 S 41 D 11 S 1775 S 87 3 87 S 41 S 13 D 11 AP 003026 47 T 06 C AP 000639 AP 000857 AP 003095 AP 003093 AP 002803

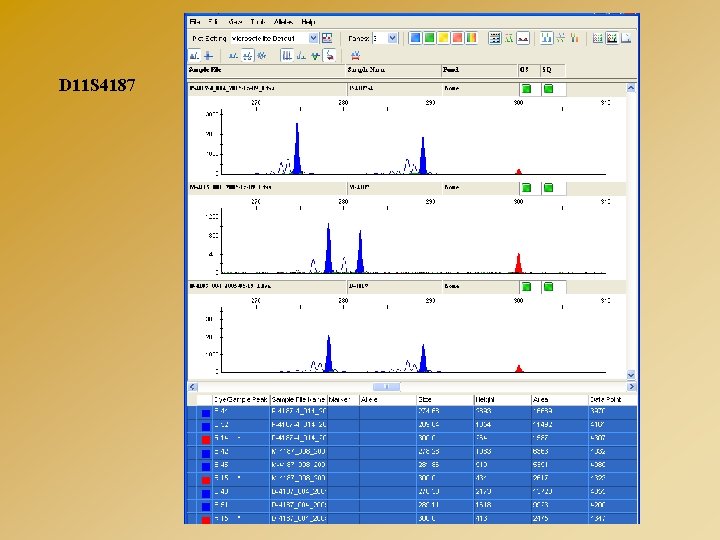
D 11 S 4187

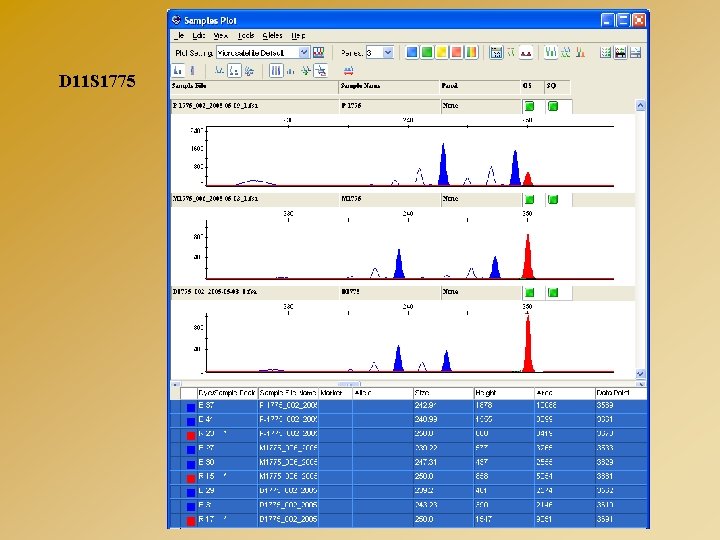
D 11 S 1775

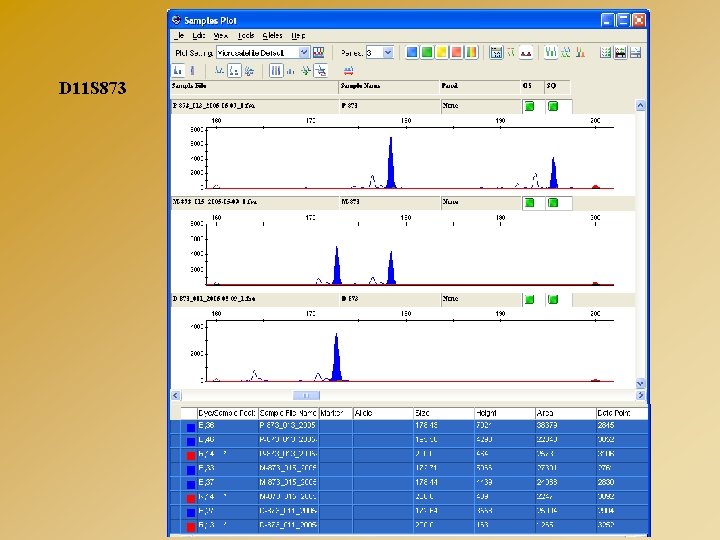
D 11 S 873

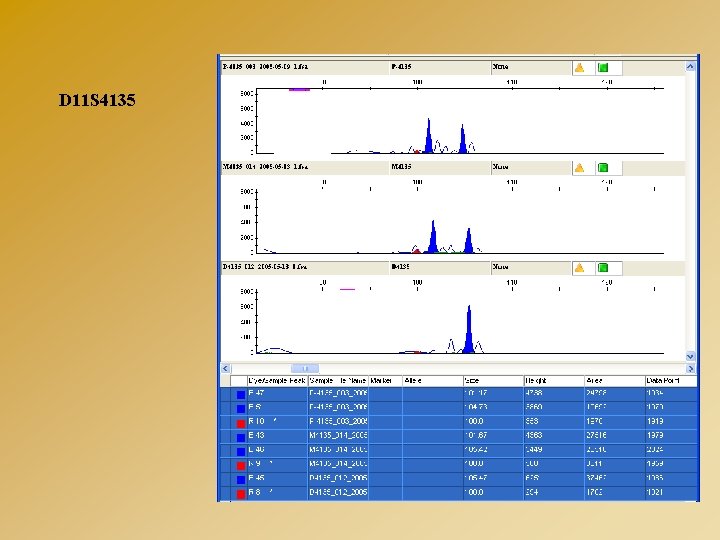
D 11 S 4135

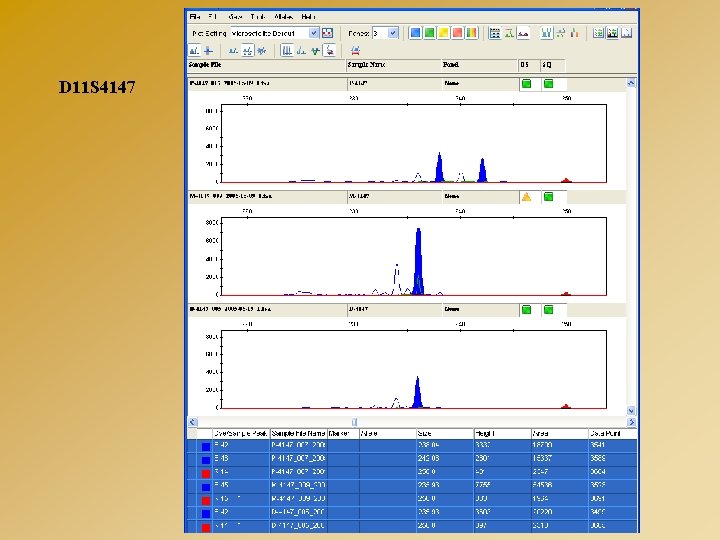
D 11 S 4147

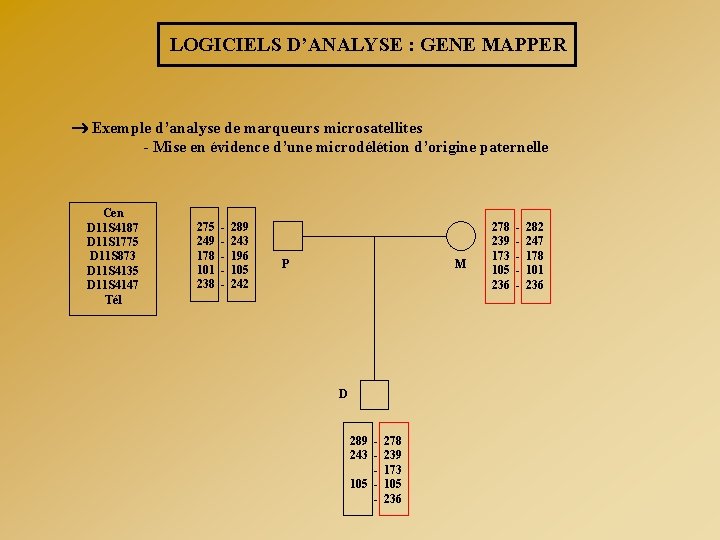
LOGICIELS D’ANALYSE : GENE MAPPER Exemple d’analyse de marqueurs microsatellites - Mise en évidence d’une microdélétion d’origine paternelle Cen D 11 S 4187 D 11 S 1775 D 11 S 873 D 11 S 4135 D 11 S 4147 Tél 275 249 178 101 238 - 289 243 196 105 242 P M D 289 - 278 243 - 239 - 173 105 - 236 278 239 173 105 236 - 282 247 178 101 236

AP 000446 AP 000773 AP 002751 AP 002797 AP 001791 AP 001984 AP 000852 9 197 D 11 S 41 47 AP 001825 AP 000639 AP 000857 AP 003095 AP 003093 D 11 S AP 003305 179 5 D 11 S 13 54 AC 023118 T Non Inf D 11 S AP 002370 Del D 11 S 41 35 D 11 S 41 87 D 11 S 13 06 AP 003026 Non Del D 11 S D 11 1775 S 87 3 Non Del C AP 002803 - D 11 S 4135 est délété - Tester D 11 S 1795, D 11 S 1354 et/ou D 11 S 1979 pour déterminer la borne télomérique