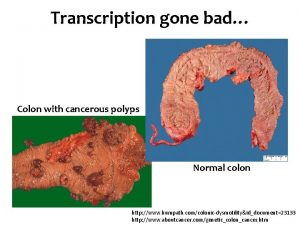
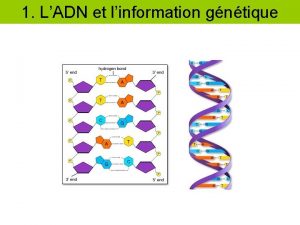
LA TRANSCRIPTION DE LADN CHEZ LES EUCARYOTES 1

































- Slides: 45

LA TRANSCRIPTION DE L’ADN CHEZ LES EUCARYOTES


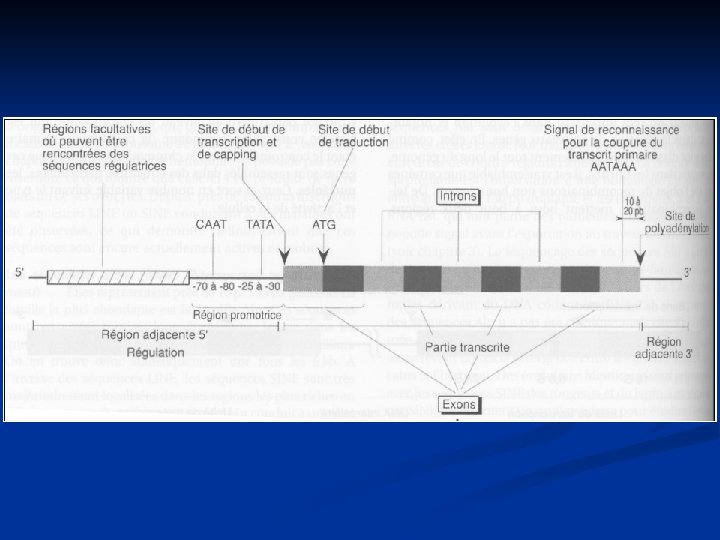
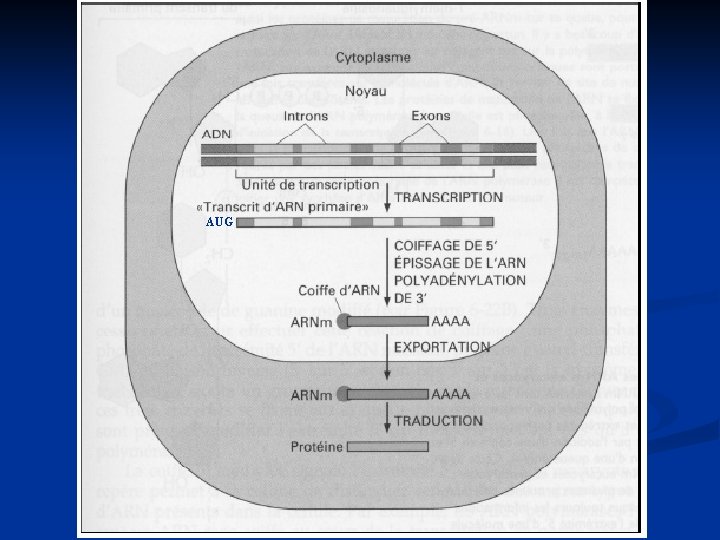
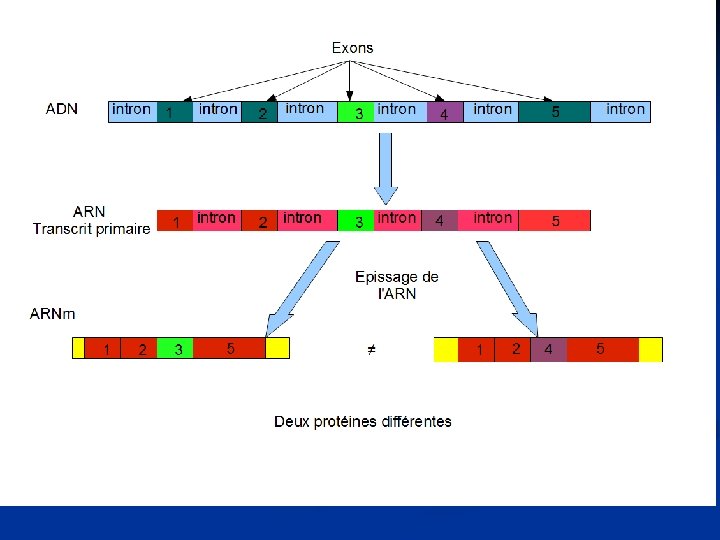
1. Les différentes phases de la transcription ØTranscription intégrale du gène (exons + introns) Ø Addition du « cap » en 5’ : GMP méthylé sur l’azote 7 Mise en place rapide (avant la fin de la transcription) Protection de l’ARNm des enzymes de dégradation Ø Addition de poly. A Après transcription, addition d’environ 250 A Aide passage vers cytoplasme Ø Maturation du pré ARNm Ø Épissage = coupure et élimination des introns

AUG

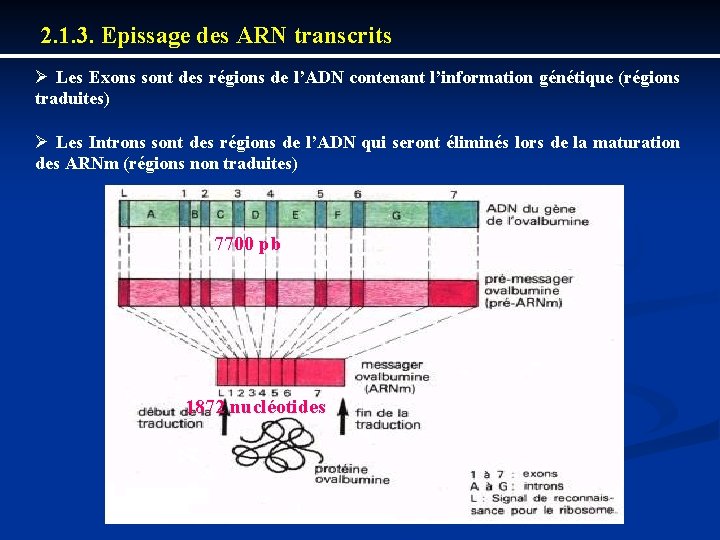
2. 1. 3. Epissage des ARN transcrits Ø Les Exons sont des régions de l’ADN contenant l’information génétique (régions traduites) Ø Les Introns sont des régions de l’ADN qui seront éliminés lors de la maturation des ARNm (régions non traduites) 7700 pb 1872 nucléotides

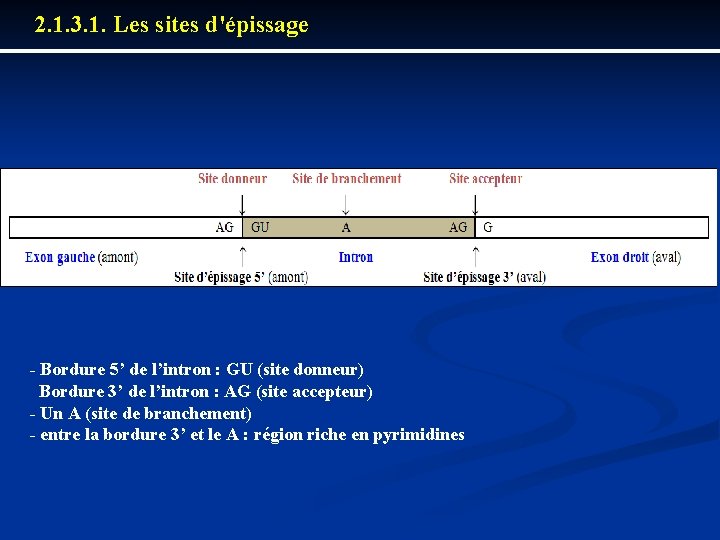
2. 1. 3. 1. Les sites d'épissage Bordure 5’ de l’intron : GU (site donneur) Bordure 3’ de l’intron : AG (site accepteur) Un A (site de branchement) entre la bordure 3’ et le A : région riche en pyrimidines

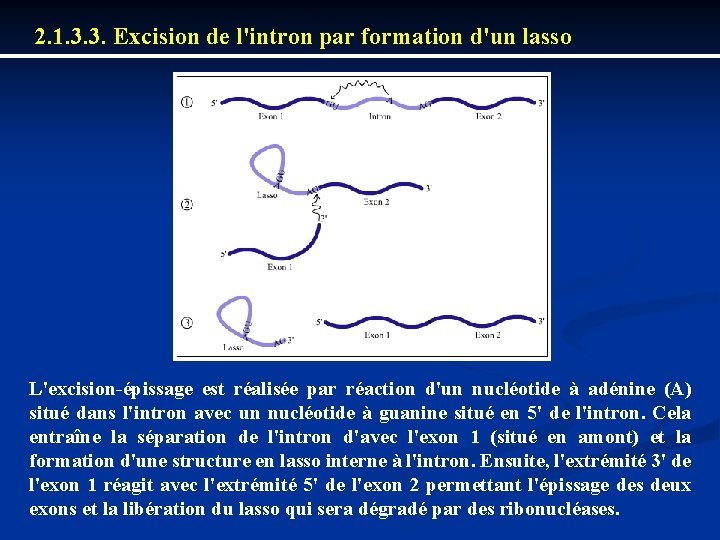
2. 1. 3. 3. Excision de l'intron par formation d'un lasso L'excision épissage est réalisée par réaction d'un nucléotide à adénine (A) situé dans l'intron avec un nucléotide à guanine situé en 5' de l'intron. Cela entraîne la séparation de l'intron d'avec l'exon 1 (situé en amont) et la formation d'une structure en lasso interne à l'intron. Ensuite, l'extrémité 3' de l'exon 1 réagit avec l'extrémité 5' de l'exon 2 permettant l'épissage des deux exons et la libération du lasso qui sera dégradé par des ribonucléases.

LA REGULATION DE L’EXPRESSION DES GENES

v la régulation de l’expression des gènes est cruciale pour la survie des organismes. v Chaque étape doit être régulé , depuis les gènes jusqu'à la formation des protéines. v Les mécanismes de régulation sont différents entre les procaryotes et les eucaryotes.

LA REGULATION DE L’EXPRESSION DES GENES CHEZ LES EUCARYOTES



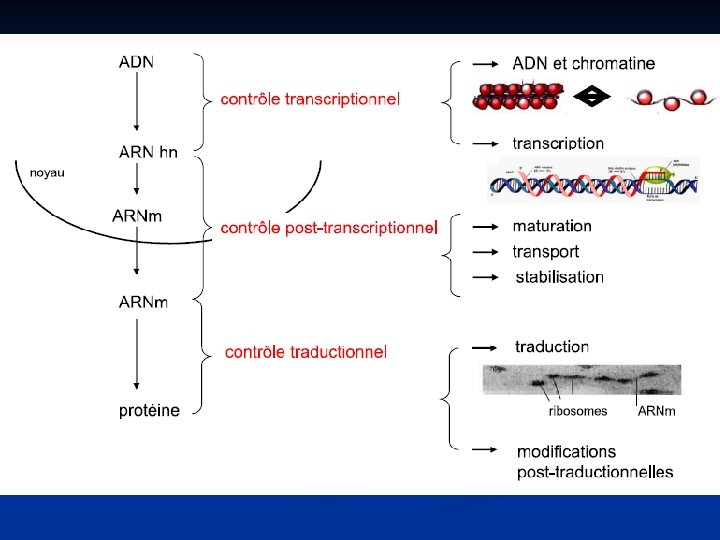
Chez les eucaryotes 5 niveaux de régulation: ■ Chromatinien. ■ Transcriptionnel. ■ Post transcriptionnel. ■ Traductionnel. ■ Post traductionnel.

Chez les eucaryotes ■ Niveau chromatinien: ■ Niveau post transcriptionnel: Acétylation des histones. Epissage alternatif. Méthylation des cytosines. Durée de vie de l’ARNm. ■ Niveau transcriptionnel: L’édition. ■ Niveau traductionnel: Choix du promoteur. Facteurs transcriptionnnnnels. Protéine activatrice ou répressive. ■ Niveau post traductionnel: Coupures. Modifications covalentes.

Niveau chromatinien


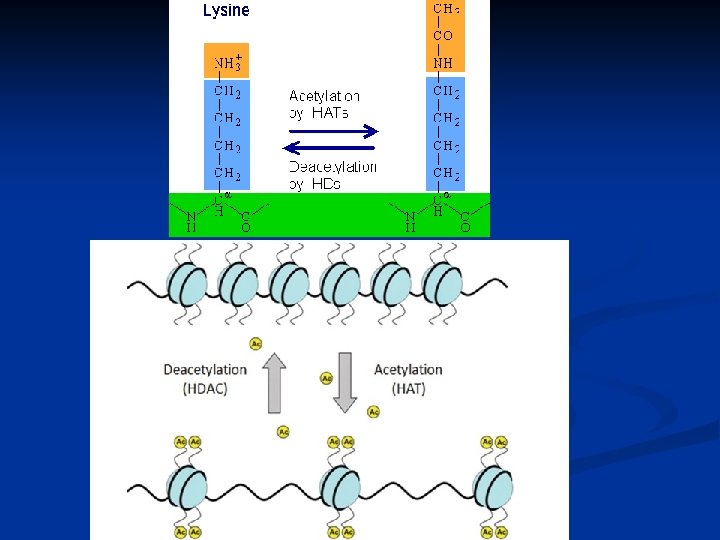
Acétylation des histones. L'acétylation de résidus de lysine à l'extrémité N-Terminale d'une protéine histone neutralise les charges positives et modifie la taille de la chaine latérale du résidu, ce qui induit un changement de conformation des protéines modifiées et le mode d'interaction avec leurs molécules cibles, en particulier l'ADN qui est chargé négativement. Autrement dit, l'acétylation des lysines réduit l'affinité "histone/ADN". Ceci favorise l'accès de la région promotrice du gène à l'ARN polymérase et aux facteurs de transcription. L'acétylation des histones est une modification réversible et favorise la transcription tandis que le clivage (désacétylation), catalysée par des déacétylases, de ces mêmes histones l'inhibe.

§l'hyper-acétylation des histones par les histones acétyltransferases (HAT) est associée à l'activation de la transcription. §la désacétylation par les histones désacétylases ("histone deacetylase" - HDAC) est associée à la répression de la transcription. L'acétylation des histones favorise la transcription : ven décompactant la chromatine. ven diminuant la force d'interaction histones-ADN. ven créant des sites de fixation pour les complexes d'activation de la transcription



Méthylation des cytosines. La méthylation de l'ADN correspond à l'ajout d'un groupement méthyl. CH 3 sur le carbone 5 des résidus cytosines de l'ADN. Cette modification non réversible est assurée par des enzymes que sont les ADN méthyltransférases ou DNMT à partir d'un donneur de méthyl, la Sadénosyl-méthionine (SAM) Chez les mammifères, ces événements de méthylation ont lieu majoritairement au niveau de dinucléotides Cp. G (cytosine-phosphateguanine). Soixante à 90 % des dinucléotides Cp. G sont méthylés et se localisent dans des régions de chromatine compactée ou hétérochromatine, inaccessibles à la machinerie transcriptionnelle La méthylation des régions du promoteur des gènes s'oppose à la transcription des gènes. Au contraire, les gènes hypométhylés ont une transcription active.


Niveau transcriptionnel

■ Niveau transcriptionnel: Choix du promoteur. Certains gènes possèdent plusieurs promoteurs de force inégale et plusieurs sites d'initiation de la transcription. Le choix du promoteur dépend de facteurs de transcriptions qui peuvent n'être présents que dans certains tissus. Les ARNm produits sont alors différents, qualitativement et quantativement, selon les tissus. Exemple : gène de l'α-amylase (enzyme : hydrolyse de l'amidon et du glycogène). Le gène de l'α-amylase comprend 2 promoteurs alternatifs P 1 et P 2. P 1 est fonctionnel dans les glandes salivaires (très actif), P 2 est fonctionnel dans le foie (peu actif).

Facteurs transcriptionnnnnels. Toute protéine nécessaire à l'initiation de la transcription est définie comme un facteur de transcription. Plusieurs facteurs de transcription agissent en reconnaissant directement des séquences cis-régulatrices au niveau des régions promotrices des gènes. Cependant, la liaison à l'ADN n'est pas toujours requise pour l'action d'un facteur de transcription. Un facteur de transcription peut ainsi reconnatre un autre facteur de transcription qui lie l'ADN directement, ou bien reconnatre directement l'ARN polymérase. Les facteurs de transcription reconnaissent généralement des petites séquences d'ADN conservées contenues au niveau des promoteurs de leurs gènes cibles. Certains de ces facteurs et de ces séquences sont communs à plusieurs gènes et utilisés de manière constitutive, et d'autres sont spécifiques de gènes et leur activité est régulée.

Les facteurs de transcription qui coopèrent avec l'ARN polymérase peuvent êtres divisés en trois groupes : Les facteurs généraux de transcription, qui sont nécessaires à l'initiation de la synthèse de l'ARN au niveau des gènes de classe II (gènes codants pour des protéines). Ils forment avec l'ARN pol II un complexe au niveau du site d'initiation de la transcription et déterminent ainsi le site de démarrage de la transcription ; ce complexe est appelé complexe basal de transcription. Les facteurs de transcription spécifiques, qui reconnaissent des petites séquences d'ADN localisées en amont du site de démarrage de la transcription augmentent l'efficacité de la transcription des gènes contenant les botes GC dans leurs promoteurs. Les facteurs de transcription inductibles, qui sont synthétisés ou activés d'une manière spécifique de tissu. Les séquences qu'ils reconnaissent sont appelées les éléments de réponse.

Les facteurs de transcription dits trans-régulateurs sont des protéines particulières produites par d'autres gènes. Ces protéines présentent des caractéristiques structurales ; les domaines Plusieurs familles de facteurs de transcription ont été aussi définies selon les domaines de liaison à l'ADN utilisés.


Les régions cis régulatrices Les séquences amplificatrices de type enhancers Les enhancers fixent des protéines qui vont permettre l’amplification de l’expression des gènes de 10 à 100 fois. Ils sont actifs dans les deux directions. Ils sont généralement situés en amont du site d’initiation mais peuvent également se situer en aval au milieu de l’unité de transcription. Les séquences extinctrices de type silencers Les silencers sont des séquences fixant des protéines qui inhibent l’expression des gènes. Les séquences isolantes de type insulators Les insulators sont des séquences isolantes qui permettent d’isoler certaines régions du génome.

Niveau post transcriptionnel

Niveau post transcriptionnel: Epissage alternatif. Le génome humain compte environ 30 000 gènes. Le protéome correspond à l’ensemble des protéines codées par l’ensemble des gènes. Le protéome humain estimé entre 100 000 et 1 000 de protéines.

A partir d’un transcrit primaire on peut avoir deux ou plus ARNm matures qui seront à l’origine de la formation des protéines-isoformes. Ceci est possible grâce à l’épissage alternatif qui consiste en l’élimination de certains exons. En effet certains exons sont constants au niveau des différents ARNm matures et d’autres sont variables et spécifiques du tissu dans lequel se trouve la protéine isoforme. Un même ARN peut être différement épissé selon le type cellulaire ou selon stade de développement. Ces épissages sont "tissus-spécifiques". Chez l'Homme, on estime que 60% des gènes font l'objet d'un épissage alternatif.


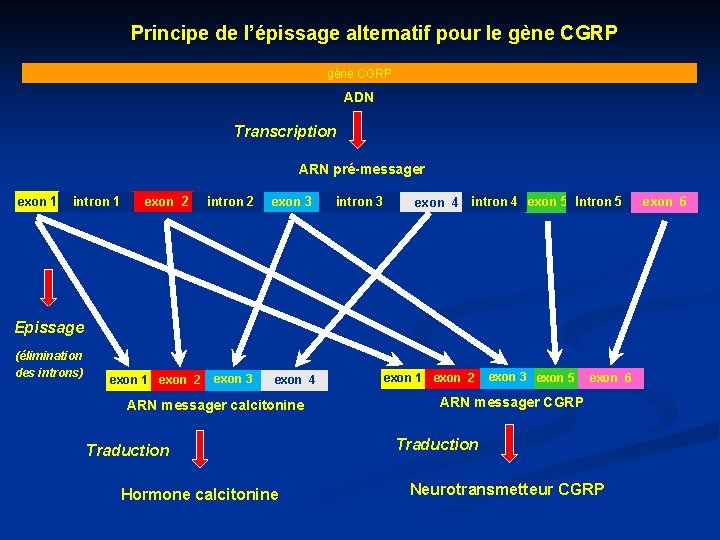
Principe de l’épissage alternatif pour le gène CGRP ADN Transcription ARN pré-messager exon 1 intron 1 exon 2 intron 2 exon 3 intron 3 exon 4 intron 4 exon 5 Intron 5 Epissage (élimination des introns) exon 1 exon 2 exon 3 exon 4 ARN messager calcitonine Traduction Hormone calcitonine exon 1 exon 2 exon 3 exon 5 exon 6 ARN messager CGRP Traduction Neurotransmetteur CGRP exon 6


Durée de vie de l’ARNm Régulation de la durée de vie des ARNm : v des séquences particulières présentes dans la région 3'UTR (5' Untranslated Transcribed Region) (Les régions non traduites) des ARNm sont responsables de leur stabilité. ≈ 30 min à 24 H. v augmentation des degrés de stabilité = augmentation degrés de quantité d'ARNm = augmentation degrés de quantité de protéines produites v diminution des degrés de stabilité = diminution degrés de quantité d'ARNm = diminution degrés de quantité de protéines produites

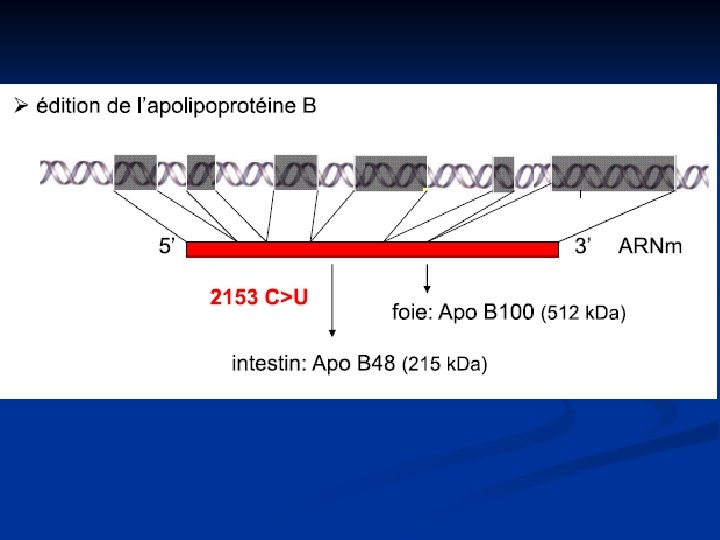
L’édition Addition, suppression ou substitution d'un ou plusieurs nucléotide aboutissant à un ARNm dont la séquence nucléotidique différe de celle du brin sens du géne. Elle a lieu dans le noyau de la cellule L'enchaînement des acides aminés dans la protéine n'est plus exactement celui de l'enchaînement des nucléotides dans les exons du gène. Un même gène peut donner naissance à des protéines différentes. Exemple le plus connu : l'apolipoprotéine B (protéine de transport des lipides dans le sang). Dans le foie, l'ARNm donne une protéine de 4536 acides aminés (Apo. B 100). Dans les cellules intestinales, un C est désaminé en U → un codon CAA (Gln) est modifié en un codon UAA (codon stop). L'ARNm transcrit dans l'intestin donne une protéine de 2152 aa (B 48). L'enzyme qui intervient est un éditosome : son activité est modulée par des facteurs spécifiques du tissu.


Niveau traductionnel

Protéine activatrice ou répressive Chez les eucaryotes, il y a des mécanismes généraux qui passent par des modifications de l'activité des facteurs de traduction, qui sont régulés via des modifications comme des phosphorylations. C'est le cas en particulier de la voie m. TOR, qui adapte l'efficacité de traduction à la présence de nutriments ou de facteurs de croissance, en agissant sur l'activité du facteur d'initiation e. IF 4 E. Une structure secondaire de l'ARNm située entre la coiffe et le codon de démarrage peut bloquer le balayage par la sous-unité 40 S du ribosome (eucaryotes). Ce mécanisme est en particulier rencontré pour la régulation du gène de la ferritine chez l'homme

La ferritine est une protéine permettant le stockage du fer. Elle a une fonction de réserve. Son dosage permet d'évaluer les réserves en fer et ainsi de dépister précocement une carence en fer ou à l'opposé Les protéines régulatrices du fer (IRP, iron regulatory protein), sont des protéines capables de se fixer sur une partie spécifique de l'ARN messager nommé élément de réponse au fer ou IRE (iron responsive element).

Niveau post traductionnel

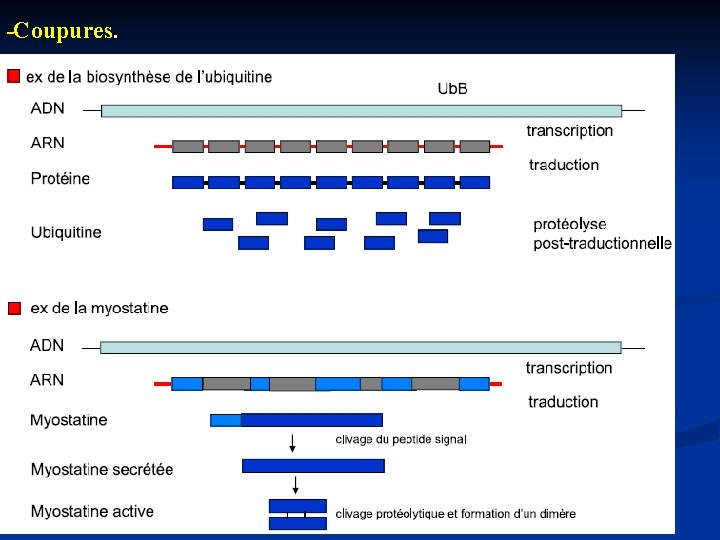
Coupures.

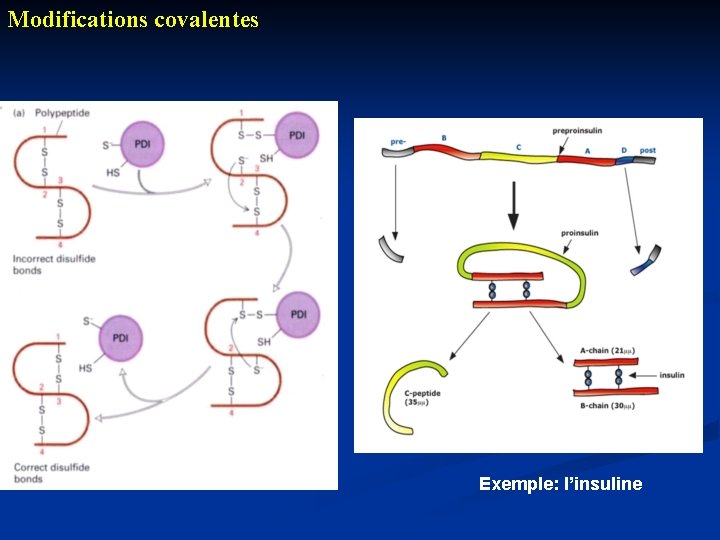
Modifications covalentes Exemple: l’insuline

FIN
 Rentrer chez dieu
Rentrer chez dieu Rentrer chez dieu comme on rentre chez soi robert lebel
Rentrer chez dieu comme on rentre chez soi robert lebel Domaine des eucaryotes
Domaine des eucaryotes Cellule eucaryote animale et végétale
Cellule eucaryote animale et végétale Mécanisme intervenant dans les échanges cellulaires
Mécanisme intervenant dans les échanges cellulaires Paurométaboles
Paurométaboles Digestion microbienne chez les ruminants
Digestion microbienne chez les ruminants Vol chez le commissaire kivala
Vol chez le commissaire kivala Chez mylo menu
Chez mylo menu L elephant kamasutra
L elephant kamasutra Gène sry chez la femme
Gène sry chez la femme Aishwarya ne
Aishwarya ne Test de coombs direct positif chez le nouveau-né
Test de coombs direct positif chez le nouveau-né Pps chez lise
Pps chez lise Travailler chez ibm
Travailler chez ibm Osteome brachial anterieur
Osteome brachial anterieur Entrez dans ma demeure
Entrez dans ma demeure Nous sommes chez nous
Nous sommes chez nous Conclusion d'un rapport de stage
Conclusion d'un rapport de stage Qu'est ce que l'absurde selon camus
Qu'est ce que l'absurde selon camus La langue de chez nous lyrics
La langue de chez nous lyrics Dna replication transcription and translation
Dna replication transcription and translation Transcription or translation
Transcription or translation Dna to rna transcription
Dna to rna transcription Phonetic transcription
Phonetic transcription Transcription
Transcription Speech phonetic transcription
Speech phonetic transcription Basal transcription apparatus
Basal transcription apparatus Transcription
Transcription Termination of transcription in prokaryotes
Termination of transcription in prokaryotes Rho-independent terminator
Rho-independent terminator What kind of mutation
What kind of mutation Idea transcription
Idea transcription Dna transcription and translation
Dna transcription and translation Rna transcription
Rna transcription 2 phonetics
2 phonetics Transcription translation replication
Transcription translation replication Differential transcription
Differential transcription Dna transcription
Dna transcription Ocr transcription
Ocr transcription Transcription
Transcription Transcription
Transcription Translation transcription
Translation transcription Translation transcription
Translation transcription Biology transcription and translation
Biology transcription and translation Read the transcriptions and write the words
Read the transcriptions and write the words