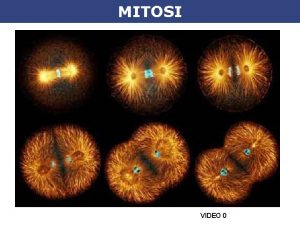
La meccanica della mitosi Cell cycle control from




































- Slides: 61

La meccanica della mitosi


Cell cycle control: from models to molecules Inhibitory kinase CLB (cdc 13) wee 1 CLB cdc 2 Inactive (weakly active) Active MPF (CDK 1) P CLB (cdc 13) cdc 2 P cdc 2 CDK 1 Inactive P (cdc 13) Remove inhibitory phosphate cdc 25 P Activating kinase Positive feedback ECB 18 -11 and 18 -12 “MPF” contains two components: cdc 25 (inactive) cdc 2 gene product = catalytic subunit of protein kinase cyclin B (CLB = cdc 13): regulatory subunit activates kinase MPF = “Cyclin-dependent kinase (CDK 1)” MPF (CDK 1) activity is also regulated by phosphorylation wee 1 is inhibitory kinase cdc 25 is activating phosphatase “Switching on” CDK 1 (MPF) drives cell into M-phase Phosphorylate M-phase substrates Histones Lamins MAPs etc

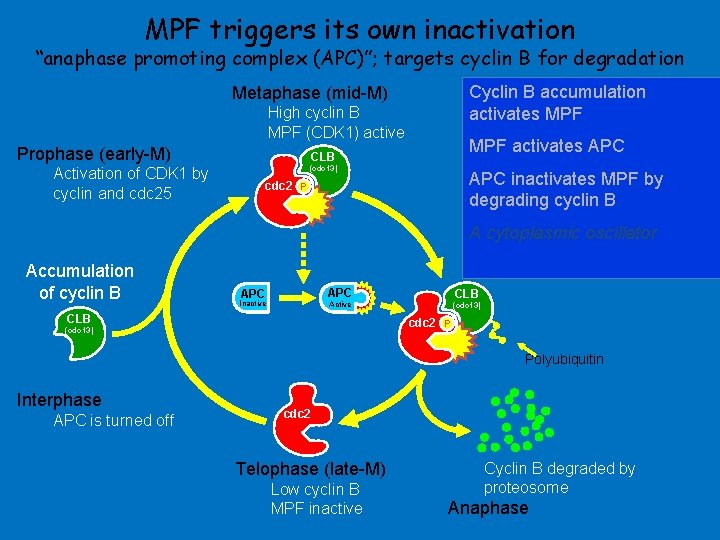
MPF triggers its own inactivation “anaphase promoting complex (APC)”; targets cyclin B for degradation Cyclin B accumulation activates MPF Metaphase (mid-M) High cyclin B MPF (CDK 1) active Prophase (early-M) Activation of CDK 1 by cyclin and cdc 25 MPF activates APC CLB (cdc 13) APC inactivates MPF by degrading cyclin B cdc 2 P A cytoplasmic oscillator Accumulation of cyclin B APC Inactive CLB Active CLB (cdc 13) cdc 2 P (cdc 13) Polyubiquitin Interphase APC is turned off cdc 2 Telophase (late-M) Low cyclin B MPF inactive Cyclin B degraded by proteosome Anaphase

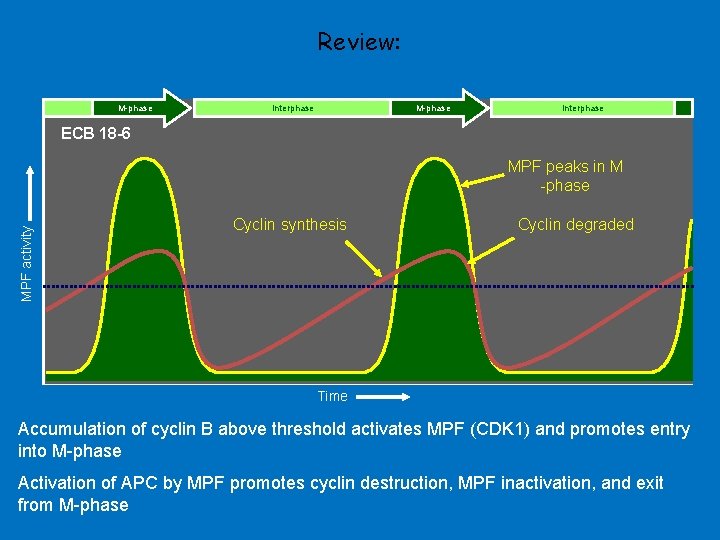
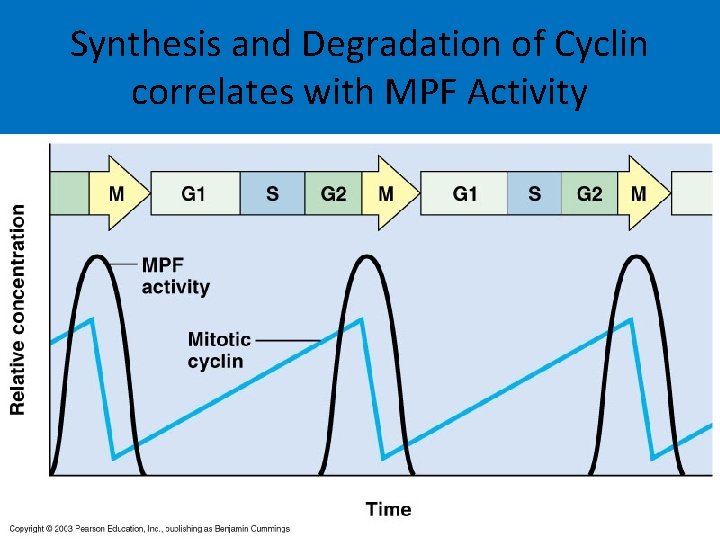
Review: M-phase Interphase ECB 18 -6 MPF activity MPF peaks in M -phase Cyclin synthesis Cyclin degraded Time Accumulation of cyclin B above threshold activates MPF (CDK 1) and promotes entry into M-phase Activation of APC by MPF promotes cyclin destruction, MPF inactivation, and exit from M-phase

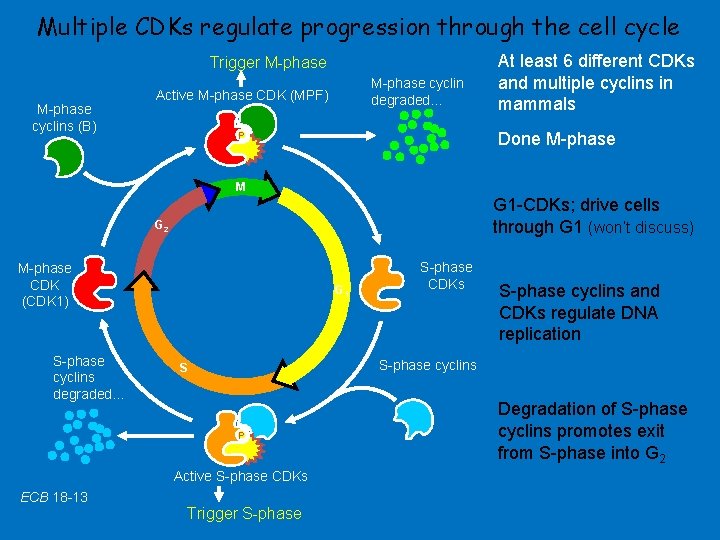
Multiple CDKs regulate progression through the cell cycle Trigger M-phase cyclins (B) M-phase cyclin degraded… Active M-phase CDK (MPF) At least 6 different CDKs and multiple cyclins in mammals Done M-phase P M G 1 -CDKs; drive cells through G 1 (won’t discuss) G 2 M-phase CDK (CDK 1) S-phase cyclins degraded… G 1 S-phase cyclins and CDKs regulate DNA replication S-phase cyclins S P Active S-phase CDKs ECB 18 -13 S-phase CDKs Trigger S-phase Degradation of S-phase cyclins promotes exit from S-phase into G 2

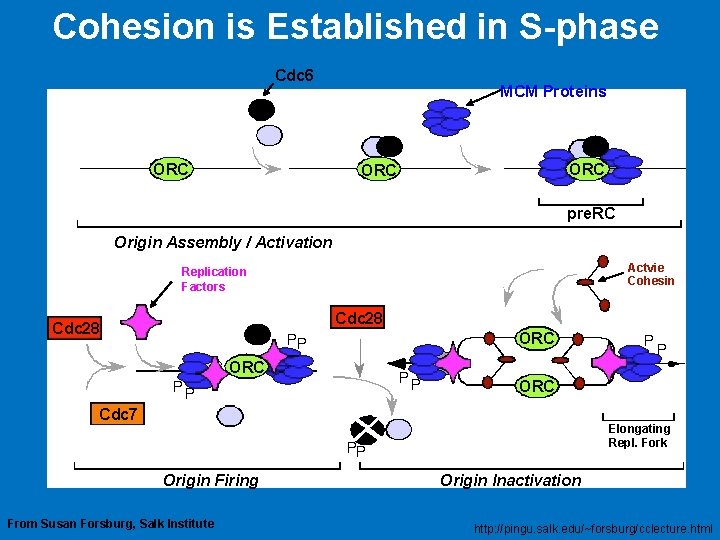
S-Cdk regulates DNA replication Origin recognition complex protein scaffolding for assembly of other proteins Cdc 6 increases in G 1; binds ORC and induces binding of other proteins forming prereplicative complex Origin is ready to fire ECB 18 -14 Active S-Cdk 1 - phosphorylates ORC causing origin to fire = replication 2 -phosphorylates Cdc 6 leading to ubiquitination and degradation Cdc 6 not made until next G 1 - prevents origin from double firing

Completion of critical cellular processes is monitored at cell cycle “check points” ECB 18 -17 Is the cell big enough? Is the environment favorable? Is DNA undamaged? Yes? Enter S phase Is DNA undamaged? Is DNA replicated? Is cell big enough? Yes? Enter M phase Have all chromosomes attached to spindle? Yes? Proceed to anaphase Of these, the G 1/S checkpoint for damaged DNA is best understood

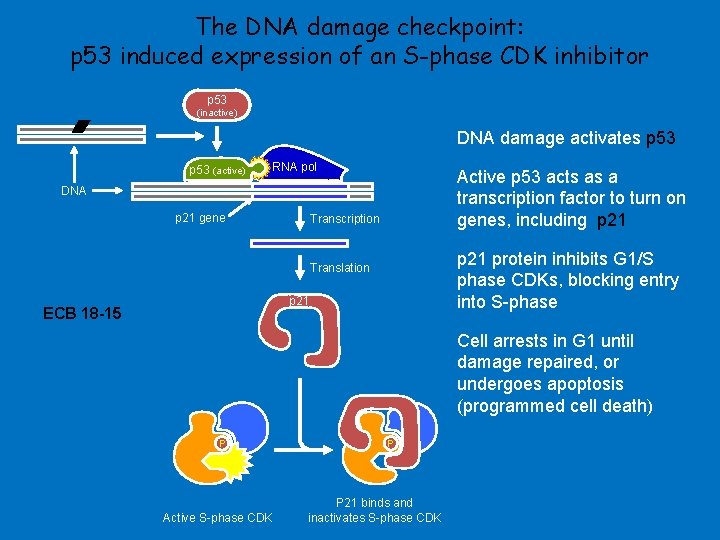
The DNA damage checkpoint: p 53 induced expression of an S-phase CDK inhibitor p 53 (inactive) DNA damage activates p 53 (active) RNA pol Active p 53 acts as a transcription factor to turn on genes, including p 21 DNA p 21 gene Transcription p 21 protein inhibits G 1/S phase CDKs, blocking entry into S-phase Translation p 21 ECB 18 -15 Cell arrests in G 1 until damage repaired, or undergoes apoptosis (programmed cell death) P Active S-phase CDK P P 21 binds and inactivates S-phase CDK

• The mechanism of regulation of the entry into mitosis at the G 2/M checkpoint involves the active form of the protein MPF (cyclin B/Cdk 1 complex phosphorylated at one amino acid, dephosphorylated at two other amino acids). (MPF stands for "maturation promoting factor. ") • This is related to the continual synthesis of cyclin, its degradation, a kinase that phosphorylates Cdk 1, and a phosphatase that dephosphorylates Cdk 1. DNA damage activates a separate kinase that results in deactivation of MPF. • Examples of Cdk 1/Cyclin B's Action: Condensins (protein complexes that are responsible for chromosome condensation during mitosis and meiosis) are activated by Cdk 1/cyclin B phosphorylation. Also, the breakdown of the nuclear envelope that occurs at the outset of prophase (or prophase I). The membrane breakdown involves the phosphorylation of lamins by Cdk 1/cyclin B, causing their depolymerization.



Synthesis and Degradation of Cyclin correlates with MPF Activity

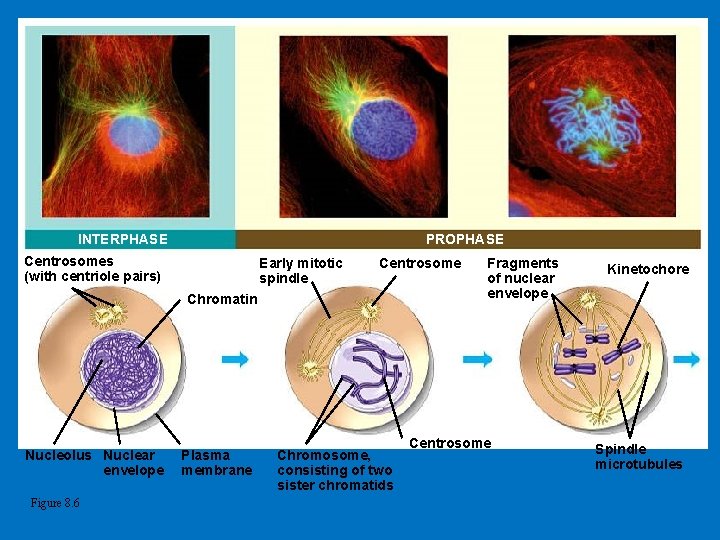
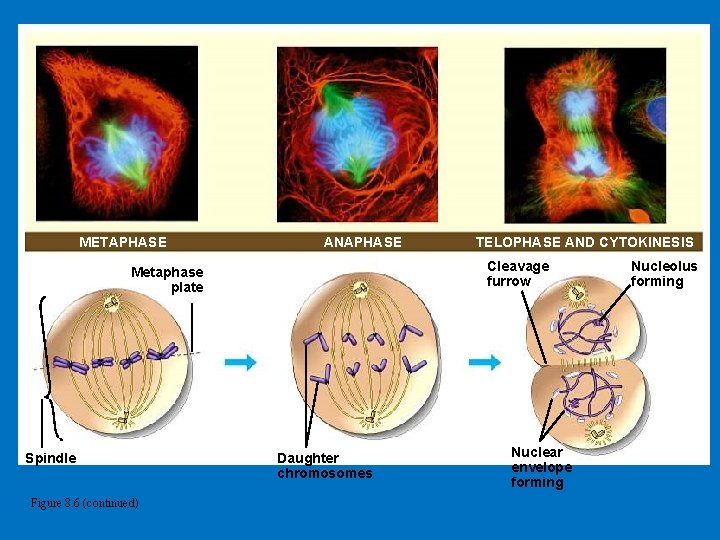
INTERPHASE PROPHASE Centrosomes (with centriole pairs) Early mitotic spindle Centrosome Chromatin Nucleolus Nuclear envelope Figure 8. 6 Plasma membrane Chromosome, consisting of two sister chromatids Fragments of nuclear envelope Centrosome Kinetochore Spindle microtubules


METAPHASE ANAPHASE Cleavage furrow Metaphase plate Spindle Figure 8. 6 (continued) TELOPHASE AND CYTOKINESIS Daughter chromosomes Nuclear envelope forming Nucleolus forming




Gli Attori: Una cellula Cromosomi Il fuso mitotico: Microtubuli… ma non solo Motori proteici e altro ancora

Anatomia di un cromosoma


Chromosome maintenance • Origins of replication • Telomeres • Centromeres


Problema n° 1 replicare e compattare ma non separare

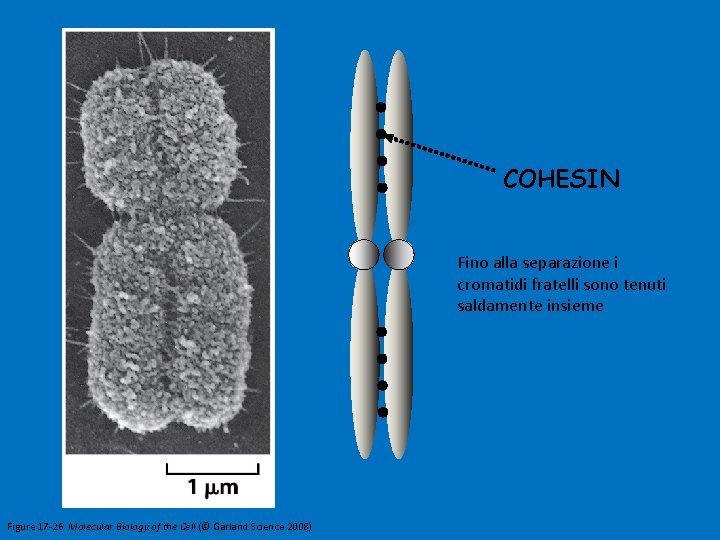
COHESIN Fino alla separazione i cromatidi fratelli sono tenuti saldamente insieme Figure 17 -26 Molecular Biology of the Cell (© Garland Science 2008)

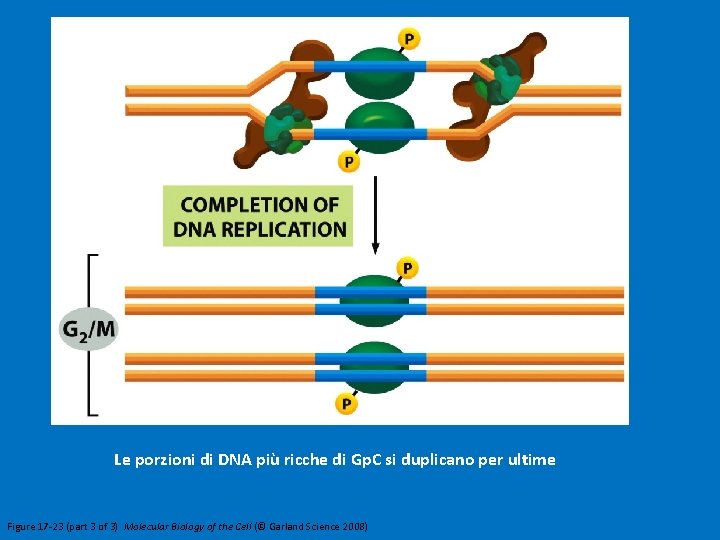
Le porzioni di DNA più ricche di Gp. C si duplicano per ultime Figure 17 -23 (part 3 of 3) Molecular Biology of the Cell (© Garland Science 2008)

Soluzione:

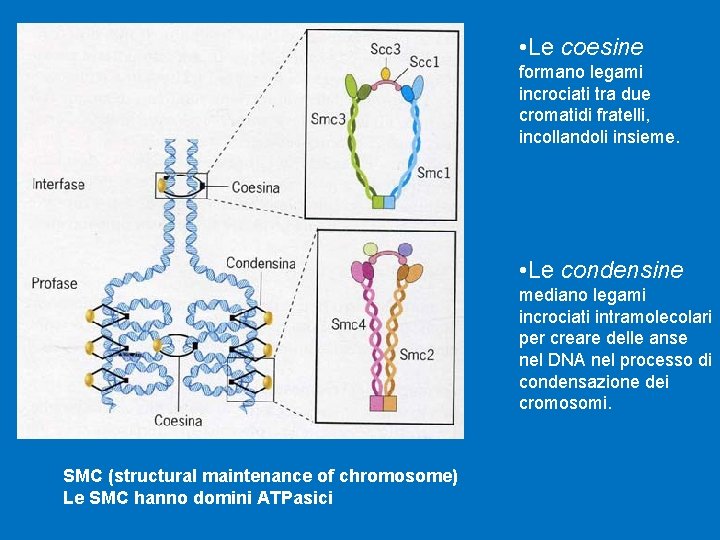
soluzione Le condensine e le coesine hanno struttura e funzione correlate: • Entrambe le proteine hanno domini di legame all’DNA e all’ATP identici ad un’estremità e una regione di cerniera nell’altra, collegate da due regioni lunghe e a “coiled-coil”. Questa struttura flessibile è ben adatta per il loro ruolo come molecole che formano legami incrociati con il DNA.

• Le coesine formano legami incrociati tra due cromatidi fratelli, incollandoli insieme. • Le condensine mediano legami incrociati intramolecolari per creare delle anse nel DNA nel processo di condensazione dei cromosomi. SMC (structural maintenance of chromosome) Le SMC hanno domini ATPasici


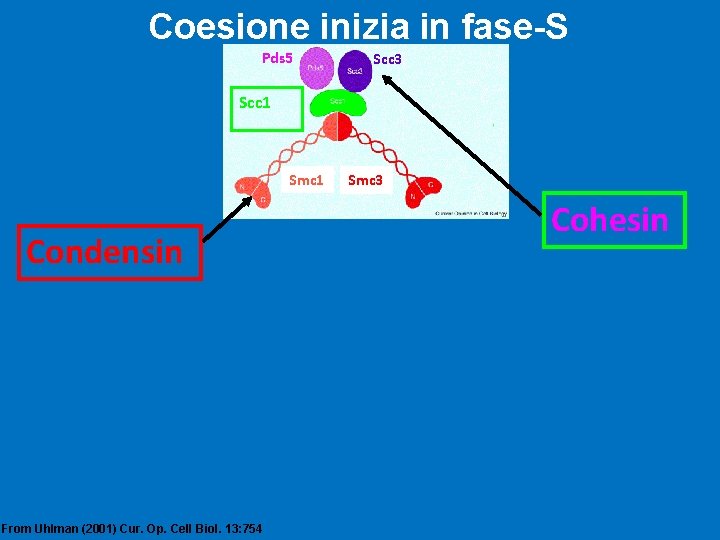
Coesione inizia in fase-S Pds 5 Scc 3 Scc 1 Smc 1 Condensin From Uhlman (2001) Cur. Op. Cell Biol. 13: 754 Smc 3 Cohesin

Coesione inizia in fase-S Condensin Cohesin Chromosome Cohesion Chromosome Compaction Scissile Subunit (Scc 1) Cleaved at M/A From Uhlman (2001) Cur. Op. Cell Biol. 13: 754

Cohesion is Established in S-phase Cdc 6 ORC MCM Proteins ORC pre. RC Origin Assembly / Activation Actvie Cohesin Replication Factors Cdc 28 ORC ORC Cdc 7 Elongating Repl. Fork Origin Firing From Susan Forsburg, Salk Institute Origin Inactivation http: //pingu. salk. edu/~forsburg/cclecture. html

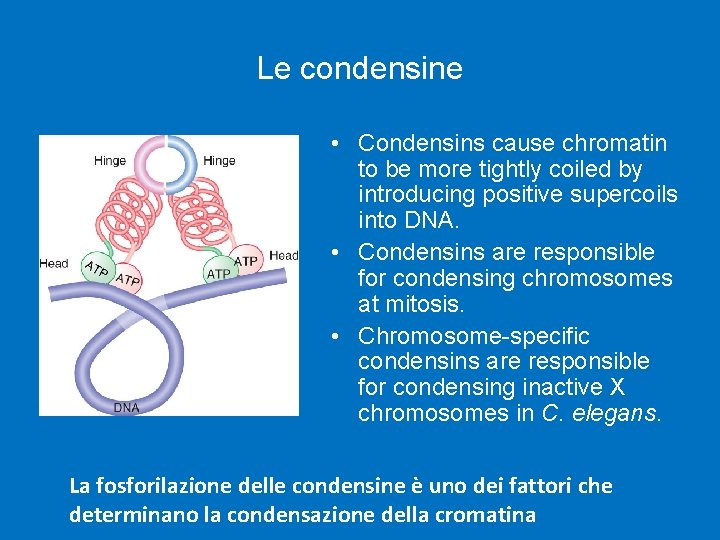
Le condensine • Condensins cause chromatin to be more tightly coiled by introducing positive supercoils into DNA. • Condensins are responsible for condensing chromosomes at mitosis. • Chromosome-specific condensins are responsible for condensing inactive X chromosomes in C. elegans. La fosforilazione delle condensine è uno dei fattori che determinano la condensazione della cromatina

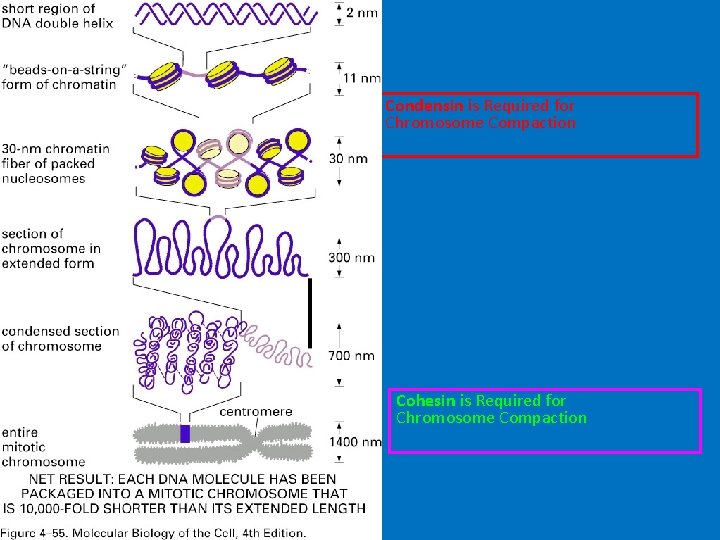
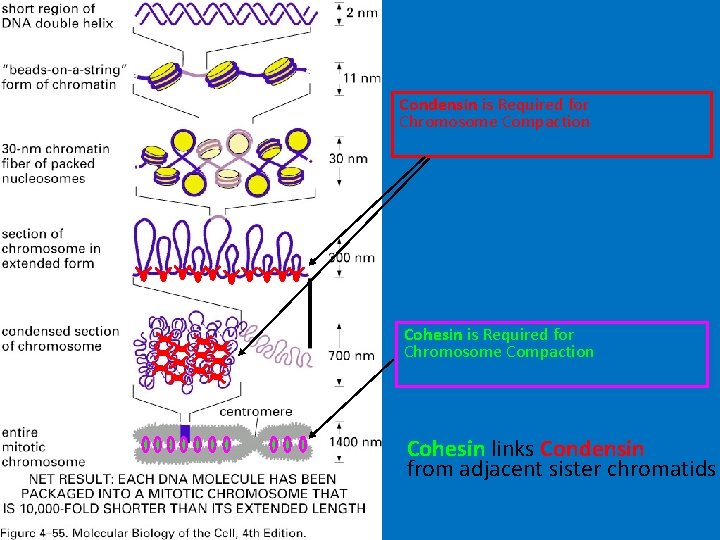
Condensin is Required for Chromosome Compaction Cohesin is Required for Chromosome Compaction

Condensin is Required for Chromosome Compaction Cohesin links Condensin from adjacent sister chromatids

Le code amino terminali degli istoni del core nucleosomale sono essenziali per la condensazione cromosomica • H 1 e’ substrato di ciclina B/ Cdk 1, tuttavia la condensazione cromosomica in estratti mitotici di Xenopus avviene anche senza H 1 • La coda N-terminale dell’istone H 3 e’ essenziale per la condensazione mitotica

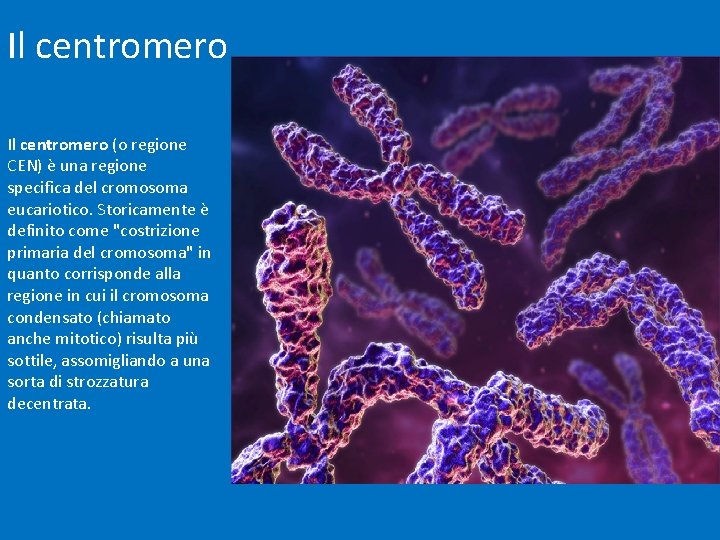
Il centromero (o regione CEN) è una regione specifica del cromosoma eucariotico. Storicamente è definito come "costrizione primaria del cromosoma" in quanto corrisponde alla regione in cui il cromosoma condensato (chiamato anche mitotico) risulta più sottile, assomigliando a una sorta di strozzatura decentrata.

Il centromero rappresenta una struttura altamente differenziata del cromosoma costituito da DNA e proteine. E’ una struttura indispensabile del cromosoma eucariotico che consente di tenere uniti i due cromatidi fratelli di ciascun cromosoma fino alla metafase e consente altresì una corretta separazione dei due cromatidi fratelli all’anafase. Per potere funzionare in modo corretto, ogni cromosoma deve possedere un solo centromero: cromosomi senza centromero o con due centromeri (dicentrici) si ripartiscono in modo anomalo durante la mitosi determinando la formazione di cellule figlie con corredo genico non bilanciato.

Modello di organizzazione del centromero La formazione del centromero è un processo complesso che consiste nell’organizzazione del DNA in cromatina centromerica tipica la quale si associa con proteine specifiche. Tre osservazioni suggeriscono che per la formazione dei centromeri non sono indispensabili sequenze specifiche ma che il meccanismo implicato è strettamente correlato a modificazioni epigenetiche: • la sequenza ripetuta centromerica presente negli eucarioti superiori non è conservata in insetti, funghi, piante e neanche nei mammiferi; • cromosomi isodicentrici, sebbene hanno due blocchi di sequenze alfa satelliti hanno un solo centromero attivo; la scoperta di neocentromeri analfoidi in Drosophila e nell’uomo ha confermato che cinetocori funzionali possono essere assemblati in una grande varietà di sequenze genomiche.

Sebbene, verosimilmente, qualsiasi sequenza di DNA può formare un centromero, è poco chiaro in che modo una sequenza può determinare la centromerizzazione. E’ possibile, comunque, che alcune strutture primarie possano più di altre determinare strutture secondarie e terziarie che costituirebbero lo scaffold del centromero. Numerosi meccanismi epigenetici noti sembrano essere coinvolti direttamente nella formazione del centromero modificando la struttura della cromatina. Tra tali meccanismi epigenetici ricordiamo: • la metilazione dell’eterocromatina pericentromerica ricca di Cp. G; • la deacetilazione degli istoni H 3 e H 4 nelle regione centromeriche e pericentromeriche; • fosforilazione degli istoni H 1 e H 3 cionvolta nella compattazione della cromatina; • i cambiamenti conformazionali di livello superiore della cromatina determinati dal legame tra il DNA centromerico e proteine centromeriche specifiche quali per esempio. CENP-A, CENP-C.

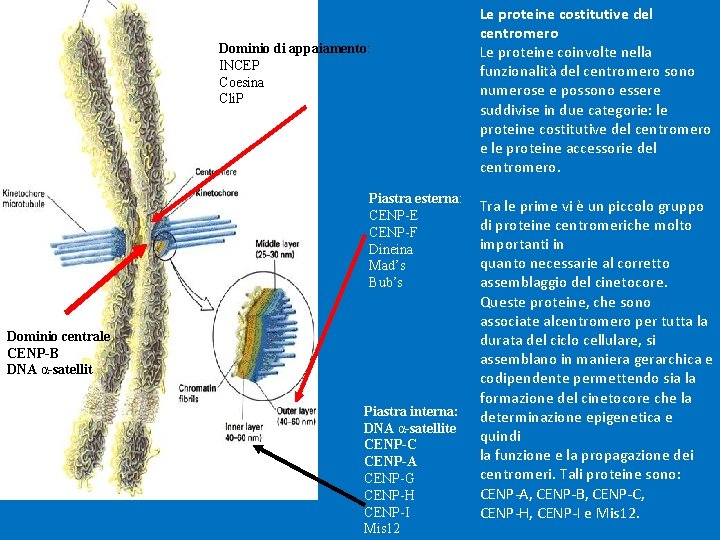
Dominio di appaiamento: INCEP Coesina Cli. P Piastra esterna: CENP-E CENP-F Dineina Mad’s Bub’s Dominio centrale CENP-B DNA α-satellit Piastra interna: DNA α-satellite CENP-C CENP-A CENP-G CENP-H CENP-I Mis 12 Le proteine costitutive del centromero Le proteine coinvolte nella funzionalità del centromero sono numerose e possono essere suddivise in due categorie: le proteine costitutive del centromero e le proteine accessorie del centromero. Tra le prime vi è un piccolo gruppo di proteine centromeriche molto importanti in quanto necessarie al corretto assemblaggio del cinetocore. Queste proteine, che sono associate alcentromero per tutta la durata del ciclo cellulare, si assemblano in maniera gerarchica e codipendente permettendo sia la formazione del cinetocore che la determinazione epigenetica e quindi la funzione e la propagazione dei centromeri. Tali proteine sono: CENP-A, CENP-B, CENP-C, CENP-H, CENP-I e Mis 12.

Il dominio centrale La cromatina centromerica può essere suddivisa in due parti: la “cromatina centrica” e la “cromatina pericentrica”. La distinzione è basata sulla presenza/assenza della proteina centromerica CENP-A. Tale proteina, che è una variante dell’istone H 3, determina la formazione di nucleosomi con caratteristiche peculiari. Infatti, la cromatina centrica, è presente nel sito in cui si assembla il cinetocore, ed è costituita da nucleosomi contenenti CENP-A alternati ad altri che presentano l’istone H 3, mentre la cromatina pericentrica ha solo nucleosomi normali, cioé contenenti l’istone H 3, e forma la cosiddetta eterocromatina, importante per la stabilizzazione e l’integrità del centromero. Il DNA centromerico è costituito da ripetizioni in tandem di piccole sequenze organizzate testa-coda. Nell’uomo il monomero ripetuto di DNA centromerico è lungo 171 bp ed è conosciuto come alfa-satellite. L’alfa-satellite è il tipo di sequenza maggiormente rappresentato nelle regioni centromeriche dei cromosomi umani e mostra dimensioni variabili (da circa 200 Kb a più di 4 Mb). L’alfa-satellite umano presenta una specifica sequenza di 17 bp, detta CENP-B box, una sequenza che è riconosciuta dalla proteina centromerica CENP-B

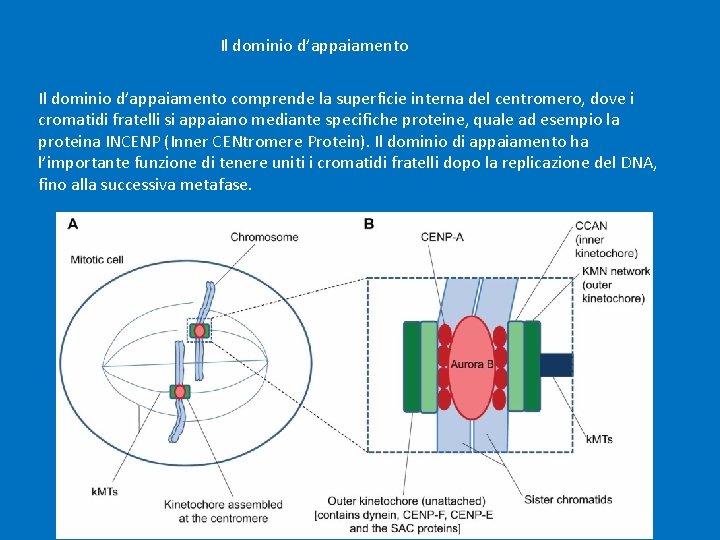
Il dominio d’appaiamento comprende la superficie interna del centromero, dove i cromatidi fratelli si appaiano mediante specifiche proteine, quale ad esempio la proteina INCENP (Inner CENtromere Protein). Il dominio di appaiamento ha l’importante funzione di tenere uniti i cromatidi fratelli dopo la replicazione del DNA, fino alla successiva metafase.

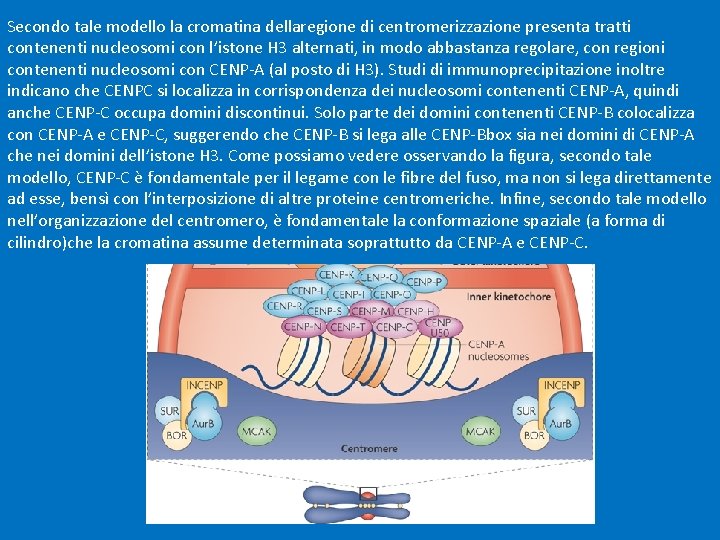
Secondo tale modello la cromatina dellaregione di centromerizzazione presenta tratti contenenti nucleosomi con l’istone H 3 alternati, in modo abbastanza regolare, con regioni contenenti nucleosomi con CENP-A (al posto di H 3). Studi di immunoprecipitazione inoltre indicano che CENPC si localizza in corrispondenza dei nucleosomi contenenti CENP-A, quindi anche CENP-C occupa domini discontinui. Solo parte dei domini contenenti CENP-B colocalizza con CENP-A e CENP-C, suggerendo che CENP-B si lega alle CENP-Bbox sia nei domini di CENP-A che nei domini dell’istone H 3. Come possiamo vedere osservando la figura, secondo tale modello, CENP-C è fondamentale per il legame con le fibre del fuso, ma non si lega direttamente ad esse, bensì con l’interposizione di altre proteine centromeriche. Infine, secondo tale modello nell’organizzazione del centromero, è fondamentale la conformazione spaziale (a forma di cilindro)che la cromatina assume determinata soprattutto da CENP-A e CENP-C.

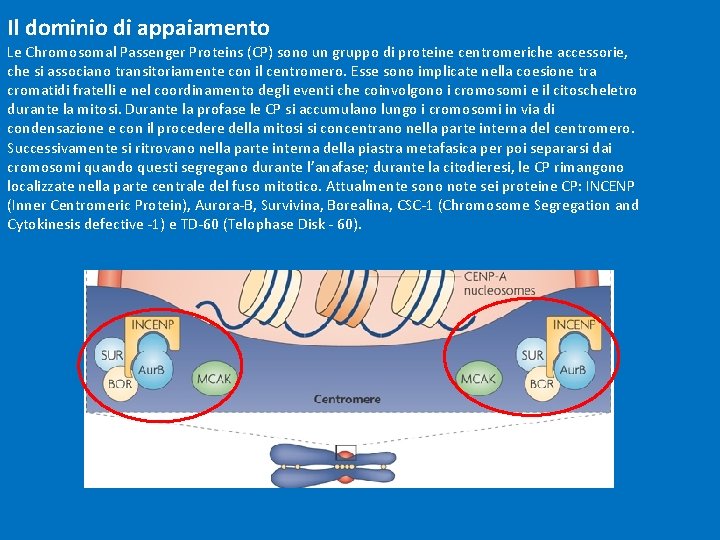
Il dominio di appaiamento Le Chromosomal Passenger Proteins (CP) sono un gruppo di proteine centromeriche accessorie, che si associano transitoriamente con il centromero. Esse sono implicate nella coesione tra cromatidi fratelli e nel coordinamento degli eventi che coinvolgono i cromosomi e il citoscheletro durante la mitosi. Durante la profase le CP si accumulano lungo i cromosomi in via di condensazione e con il procedere della mitosi si concentrano nella parte interna del centromero. Successivamente si ritrovano nella parte interna della piastra metafasica per poi separarsi dai cromosomi quando questi segregano durante l’anafase; durante la citodieresi, le CP rimangono localizzate nella parte centrale del fuso mitotico. Attualmente sono note sei proteine CP: INCENP (Inner Centromeric Protein), Aurora-B, Survivina, Borealina, CSC-1 (Chromosome Segregation and Cytokinesis defective -1) e TD-60 (Telophase Disk - 60).

Il cinetocore: il sito d'attacco per i microtubuli del fuso mitotico

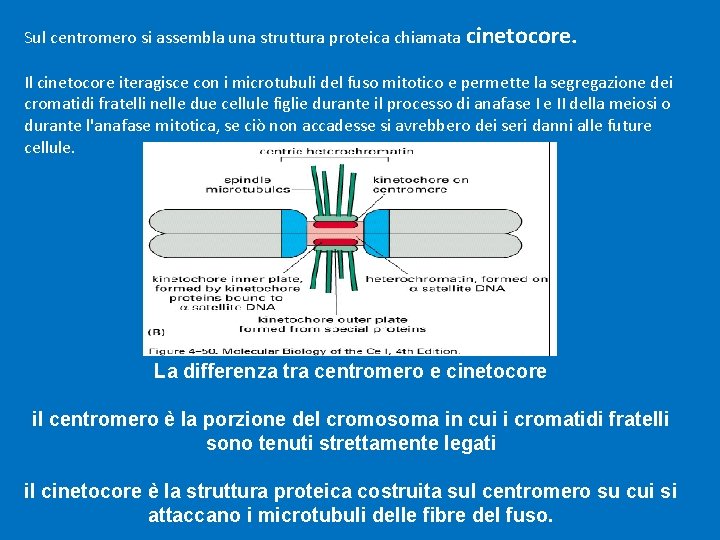
Sul centromero si assembla una struttura proteica chiamata cinetocore. Il cinetocore iteragisce con i microtubuli del fuso mitotico e permette la segregazione dei cromatidi fratelli nelle due cellule figlie durante il processo di anafase I e II della meiosi o durante l'anafase mitotica, se ciò non accadesse si avrebbero dei seri danni alle future cellule. La differenza tra centromero e cinetocore il centromero è la porzione del cromosoma in cui i cromatidi fratelli sono tenuti strettamente legati il cinetocore è la struttura proteica costruita sul centromero su cui si attaccano i microtubuli delle fibre del fuso.


COME E’ FATTO UN CINETOCORE? I cinetocori sono posizionati ai lati opposti della costrizione primaria, sulla faccia esterna del cromosoma. Al microscopio elettronico presentano una struttura a forma di disco trilaminare: due parti, la piastra esterna e quella interna, sono elettrondense, tra di loro si trova la terza parte, più chiara, di 15 -35 nm di spessore. La piastra interna è associata con la superfice dell'eterocromatina centromerica, mentre qualla esterna prende contatto con le fibre del fuso mitotico. Il cinetocore contiene proteine con funzione diversa. Alcune sono coinvolte nell’attività motoria e fondamentali per l’organizzazione strutturale come le proteine CENP (CENtromere Protein), mentre altre sono implicate nella regolazione della transizione metafaseanafase



Il complesso centromero-cinetocore è capace di svolgere funzioni essenziali in differenti aspetti della mitosi e della meiosi: -è responsabile dell’appaiamento dei cromatidi fratelli, -rappresenta il sito d'attacco per i microtubuli del fuso mitotico, -controlla la transizione metafase-anafase nel ciclo cellulare regolando il movimento dei cromosomi.

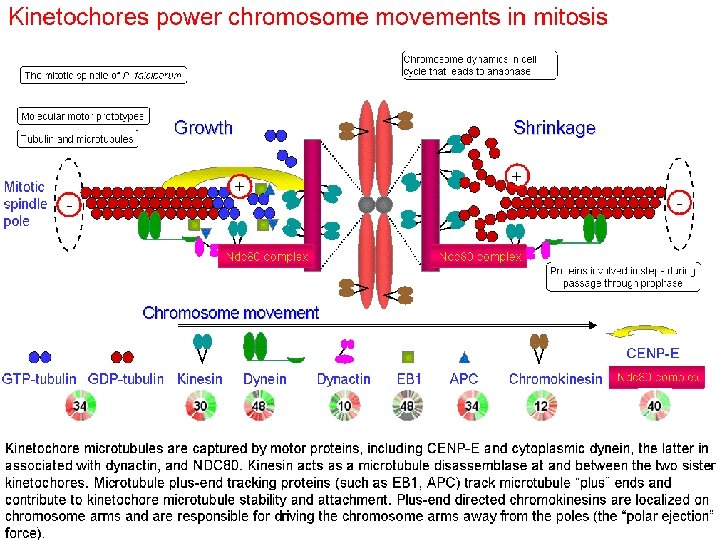
• Il corretto orientamento dei cromatidi fratelli e la segregazione degli stessi durante l'anafase sono determinati dal legame dell’estremità positiva (plus-end) dei microtubuli del fuso mitotico, provenienti dai poli opposti della cellula, alla piastra esterna del cinetocore (che contiene proteine importanti per il movimento dei cromosomi quali CENP-E e la dineina). In genere i cinetocori sono in grado di legare dai 10 ai 45 microtubuli.

A CHE SERVE IL FUSO MITOTICO? COME SI FORMA? COME FUNZIONA? COME SI ALLINEANO I CENTROSOMI? COME SI SEPARANO I CENTROSOMI? COME I MICROTUBULI SI LEGANO AI CROMOSOMI? COME VENGONO ‘AGGANCIATI’ I CROMOSOMI? COME SI MUOVONO I CROMOSOMI? COME SI SEPARANO I CROMATIDI?


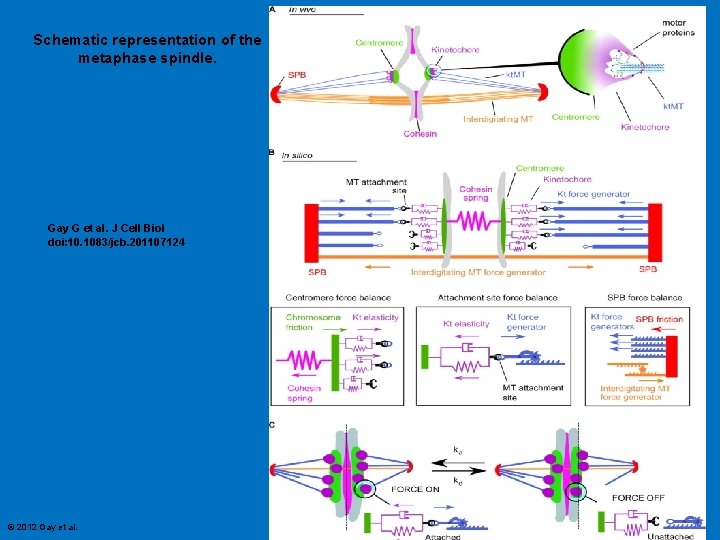
Schematic representation of the metaphase spindle. Gay G et al. J Cell Biol doi: 10. 1083/jcb. 201107124 © 2012 Gay et al.

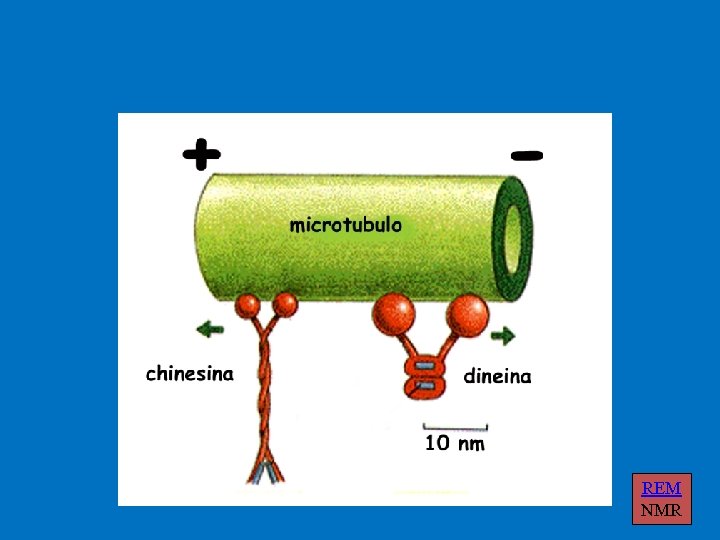
REM NMR

I motori proteici chinesine del fuso Bim. C CENP-E, ncd,

 Meiosi schema
Meiosi schema Cromosomi dicromatidici
Cromosomi dicromatidici Mitosi video
Mitosi video Aploide e diploide
Aploide e diploide Mitosi e meiosi
Mitosi e meiosi Profase mitosi zanichelli
Profase mitosi zanichelli Section 10-2 cell division
Section 10-2 cell division Cell cycle and cell division
Cell cycle and cell division Biology.arizona.edu/cell bio/activities/cell cycle/01.html
Biology.arizona.edu/cell bio/activities/cell cycle/01.html Phases of cell cycle
Phases of cell cycle Ventilazione meccanica controllata decentrata
Ventilazione meccanica controllata decentrata Impedenza meccanica
Impedenza meccanica Meccanica classica
Meccanica classica Sifone idraulico meccanica dei fluidi
Sifone idraulico meccanica dei fluidi Cqps
Cqps Meccanica del continuo
Meccanica del continuo Soffiatura meccanica del vetro
Soffiatura meccanica del vetro Sezioni con piani concorrenti
Sezioni con piani concorrenti Uno nessuno centomila schema
Uno nessuno centomila schema Durkheim solidarietà
Durkheim solidarietà Punture esplorative
Punture esplorative Lavoro ed energia potenziale
Lavoro ed energia potenziale Lavorazione meccanica
Lavorazione meccanica Energia potenziale gravitazionale
Energia potenziale gravitazionale Fatto
Fatto Energia potenziale scuola primaria
Energia potenziale scuola primaria Meccanica del continuo
Meccanica del continuo Energia potenziale negativa
Energia potenziale negativa Principio di conservazione dell'energia
Principio di conservazione dell'energia Calcolatrice meccanica funzionamento
Calcolatrice meccanica funzionamento Istituto comprensivo lorenzo lotto
Istituto comprensivo lorenzo lotto Energia meccanica
Energia meccanica Tecnologia meccanica unina
Tecnologia meccanica unina Mazzoldi nigro voci meccanica e termodinamica
Mazzoldi nigro voci meccanica e termodinamica Forma funzionale burocratica
Forma funzionale burocratica Prove della sfericità della terra zanichelli
Prove della sfericità della terra zanichelli Soluzioni il racconto della chimica e della terra
Soluzioni il racconto della chimica e della terra Il racconto della chimica e della terra soluzioni
Il racconto della chimica e della terra soluzioni Soluzioni il racconto delle scienze naturali
Soluzioni il racconto delle scienze naturali F92 disturbi misti della condotta e della sfera emozionale
F92 disturbi misti della condotta e della sfera emozionale Favola la coccinella in cerca della felicità
Favola la coccinella in cerca della felicità Prove della sfericità della terra
Prove della sfericità della terra Fichte 3 principi
Fichte 3 principi Culture e formati della televisione e della radio
Culture e formati della televisione e della radio Elena bettinelli
Elena bettinelli Soluzioni il racconto della chimica e della terra
Soluzioni il racconto della chimica e della terra Ritopei
Ritopei Fasi lavorazione seta
Fasi lavorazione seta Simonetta klein il racconto della chimica
Simonetta klein il racconto della chimica Moti millenari della terra zanichelli
Moti millenari della terra zanichelli Il racconto della chimica e della terra
Il racconto della chimica e della terra Ritratto della mia bambina
Ritratto della mia bambina Golgi body school analogy
Golgi body school analogy Advantages and disadvantages of diaphragm cell process
Advantages and disadvantages of diaphragm cell process Prokaryotic cell vs eukaryotic cell
Prokaryotic cell vs eukaryotic cell Prokaryotic reproduction
Prokaryotic reproduction Venn diagram of plant and animal cell
Venn diagram of plant and animal cell Spontaneity electrochemistry
Spontaneity electrochemistry Dry cell vs wet cell
Dry cell vs wet cell Plant cell animal cell venn diagram
Plant cell animal cell venn diagram What is the function of cell wall in plant cell
What is the function of cell wall in plant cell Plant cell structure
Plant cell structure