Initiation Factors prokaryotes Activity eukaryotes IF 3 e











- Slides: 52

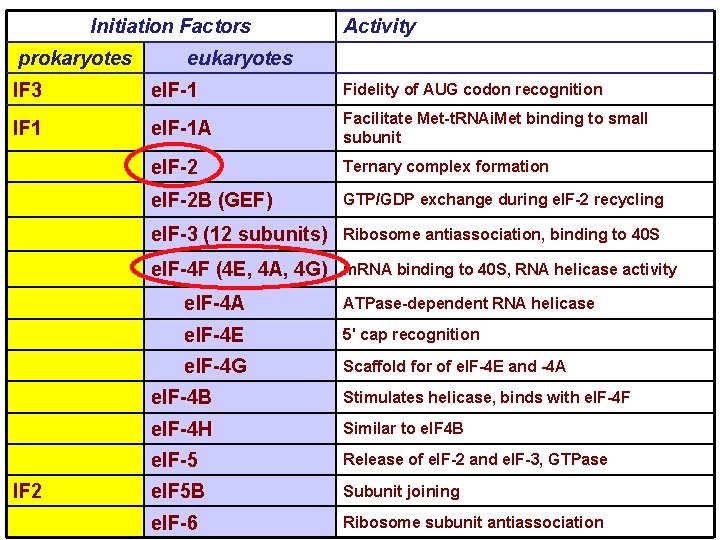
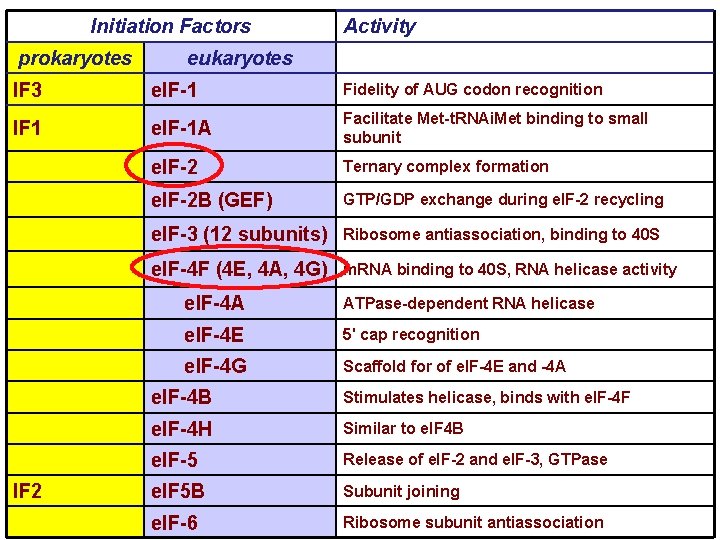
Initiation Factors prokaryotes Activity eukaryotes IF 3 e. IF-1 Fidelity of AUG codon recognition IF 1 e. IF-1 A Facilitate Met-t. RNAi. Met binding to small subunit e. IF-2 Ternary complex formation e. IF-2 B (GEF) GTP/GDP exchange during e. IF-2 recycling e. IF-3 (12 subunits) Ribosome antiassociation, binding to 40 S e. IF-4 F (4 E, 4 A, 4 G) m. RNA binding to 40 S, RNA helicase activity IF 2 e. IF-4 A ATPase-dependent RNA helicase e. IF-4 E 5' cap recognition e. IF-4 G Scaffold for of e. IF-4 E and -4 A e. IF-4 B Stimulates helicase, binds with e. IF-4 F e. IF-4 H Similar to e. IF 4 B e. IF-5 Release of e. IF-2 and e. IF-3, GTPase e. IF 5 B Subunit joining e. IF-6 Ribosome subunit antiassociation

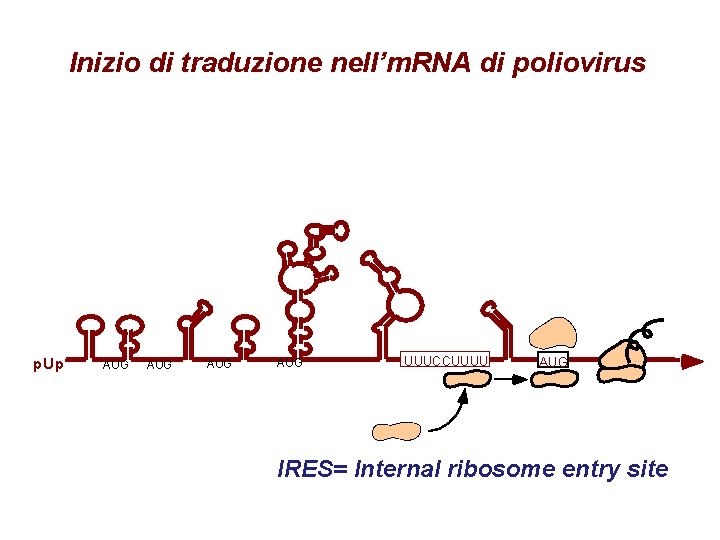
Inizio di traduzione nell’m. RNA di poliovirus p. Up AUG AUG UUUCCUUUU AUG IRES= Internal ribosome entry site

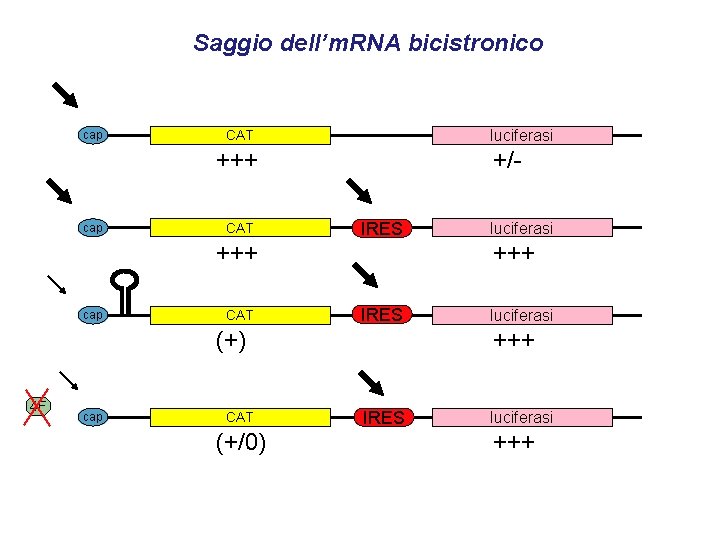
Saggio dell’m. RNA bicistronico cap luciferasi CAT +/- +++ cap CAT IRES cap CAT (+/0) luciferasi +++ (+) 4 F luciferasi IRES luciferasi +++


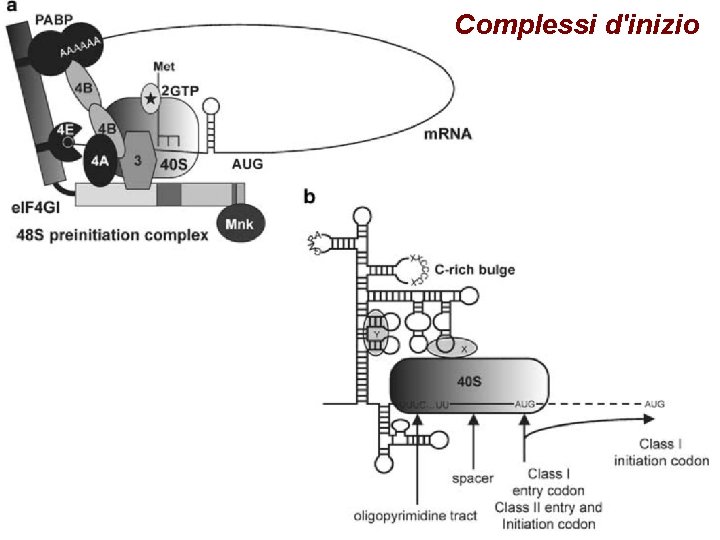
Complessi d'inizio

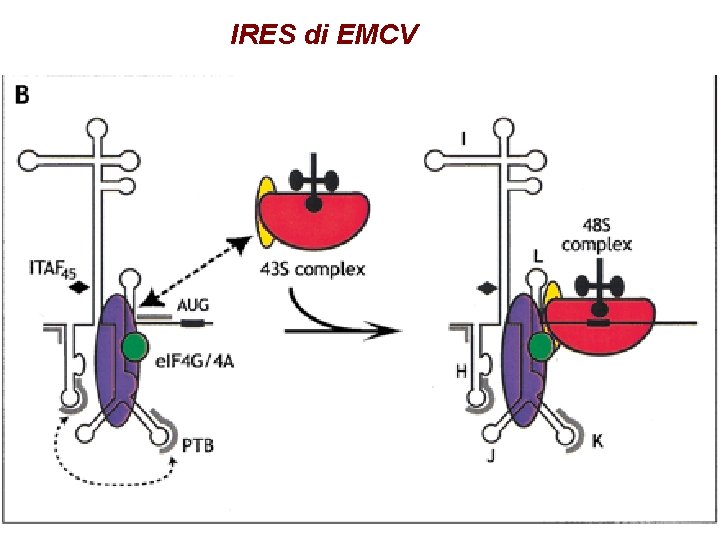
IRES di EMCV

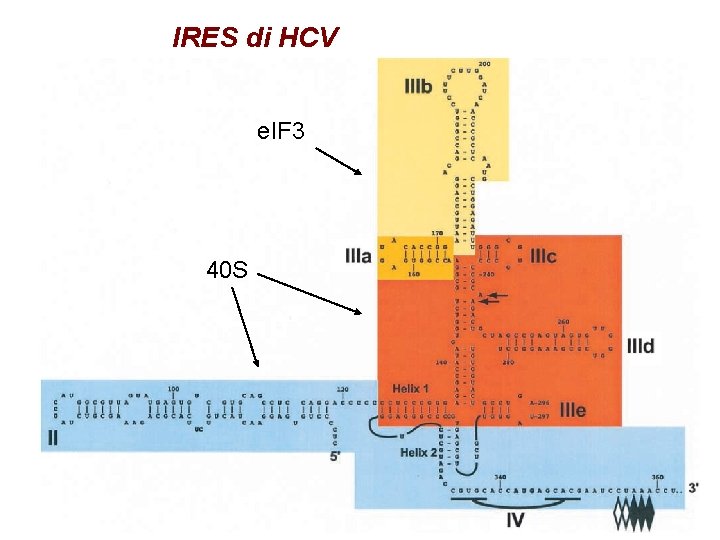
IRES di HCV e. IF 3 40 S

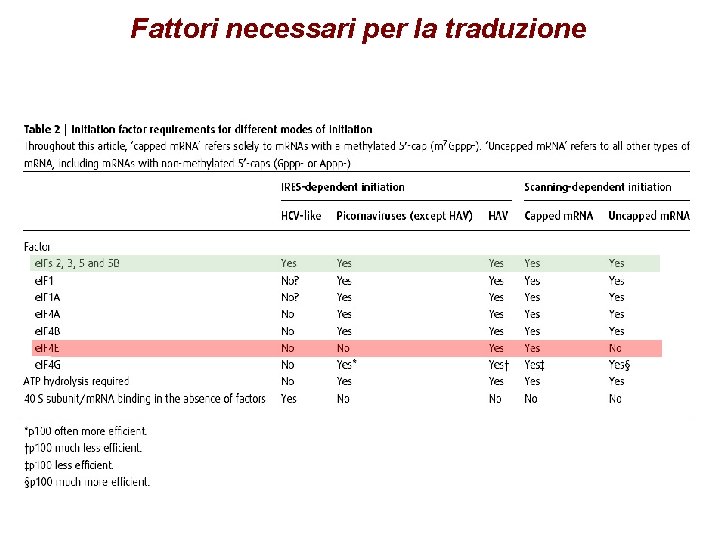
Fattori necessari per la traduzione

IRES di Cr. PV

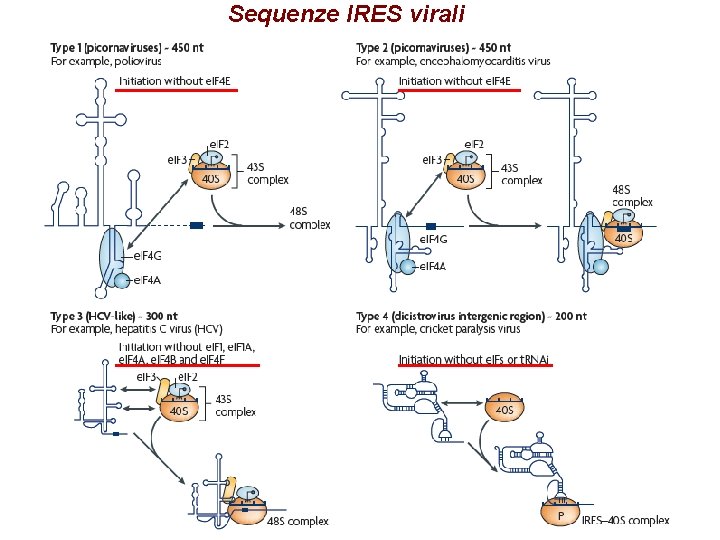
Sequenze IRES virali

IRES negli m. RNA cellulari

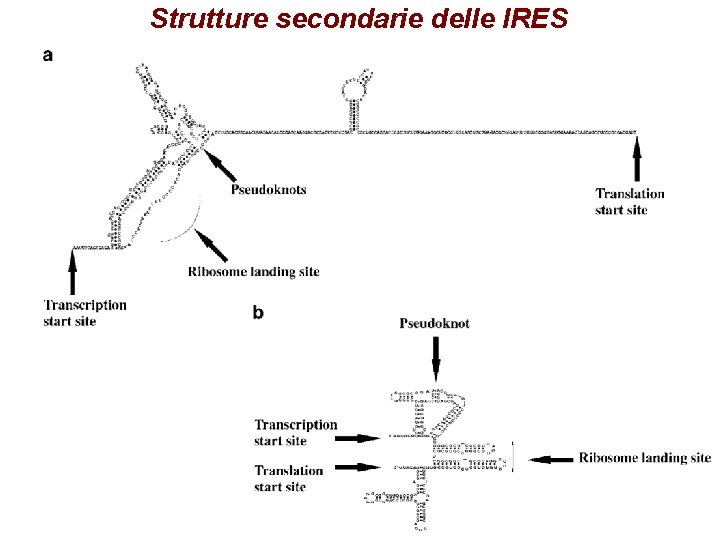
Strutture secondarie delle IRES

Fattori trans-agenti delle IRES (ITAF)

Ruolo delle IRES

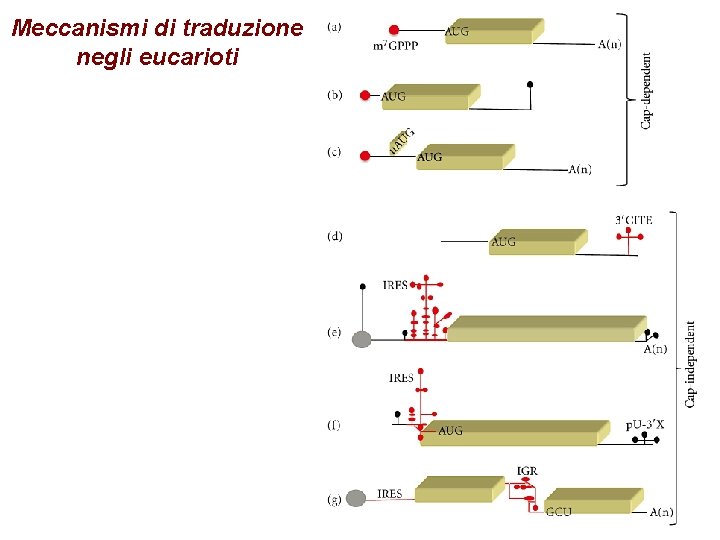
Meccanismi di traduzione negli eucarioti

Regolazione della traduzione • generale • specifica

Initiation Factors prokaryotes Activity eukaryotes IF 3 e. IF-1 Fidelity of AUG codon recognition IF 1 e. IF-1 A Facilitate Met-t. RNAi. Met binding to small subunit e. IF-2 Ternary complex formation e. IF-2 B (GEF) GTP/GDP exchange during e. IF-2 recycling e. IF-3 (12 subunits) Ribosome antiassociation, binding to 40 S e. IF-4 F (4 E, 4 A, 4 G) m. RNA binding to 40 S, RNA helicase activity IF 2 e. IF-4 A ATPase-dependent RNA helicase e. IF-4 E 5' cap recognition e. IF-4 G Scaffold for of e. IF-4 E and -4 A e. IF-4 B Stimulates helicase, binds with e. IF-4 F e. IF-4 H Similar to e. IF 4 B e. IF-5 Release of e. IF-2 and e. IF-3, GTPase e. IF 5 B Subunit joining e. IF-6 Ribosome subunit antiassociation

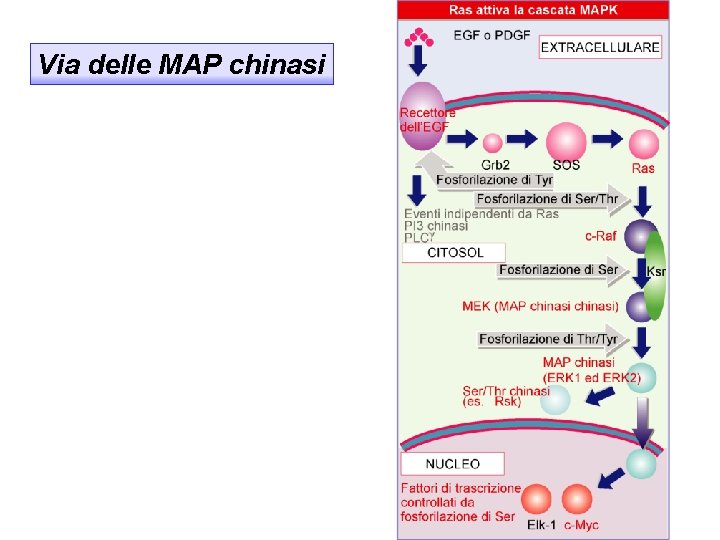
Via delle MAP chinasi

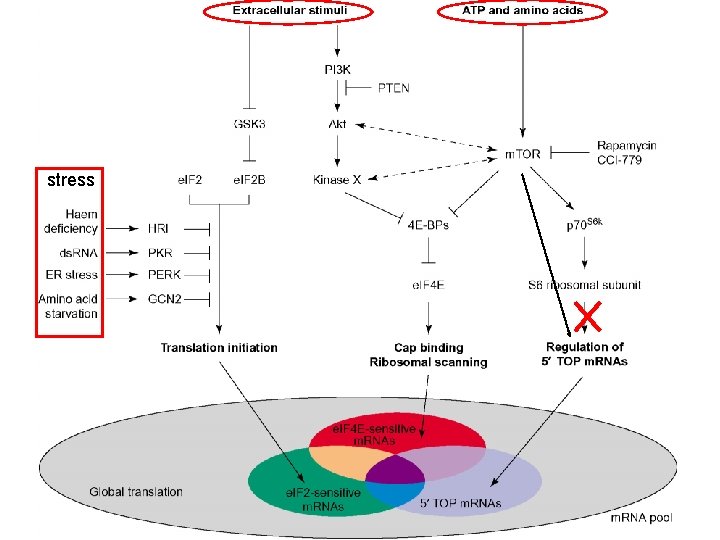
stress

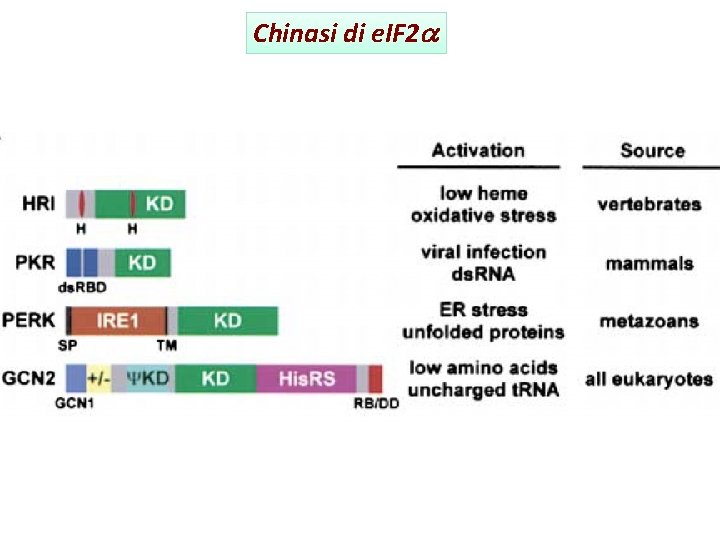
Chinasi di e. IF 2 a

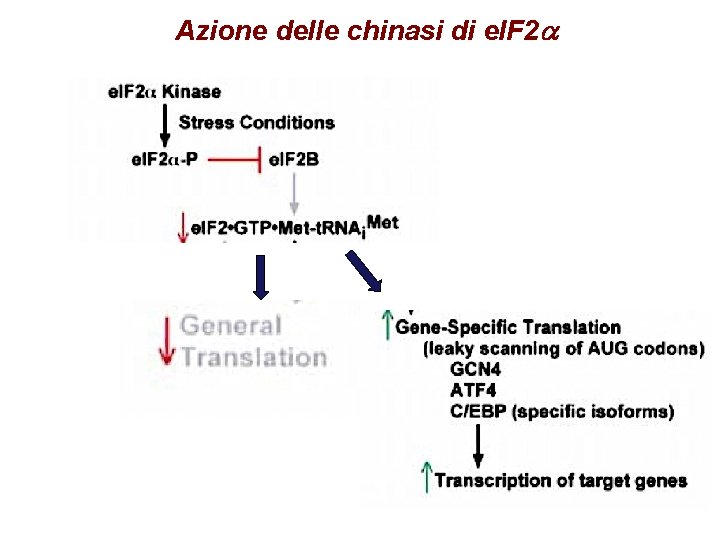
Azione delle chinasi di e. IF 2 a

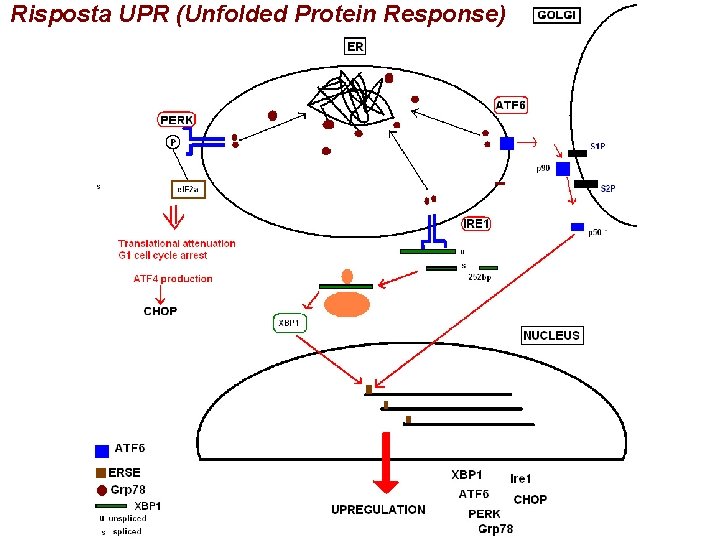
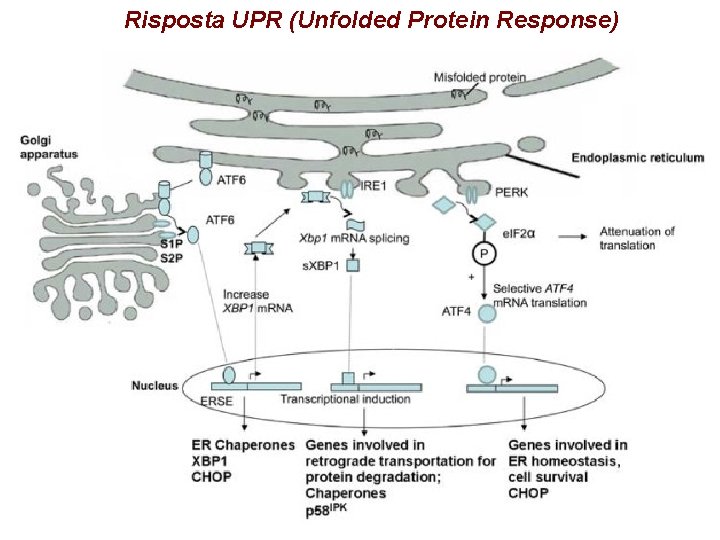
Risposta UPR (Unfolded Protein Response)

Risposta UPR (Unfolded Protein Response)

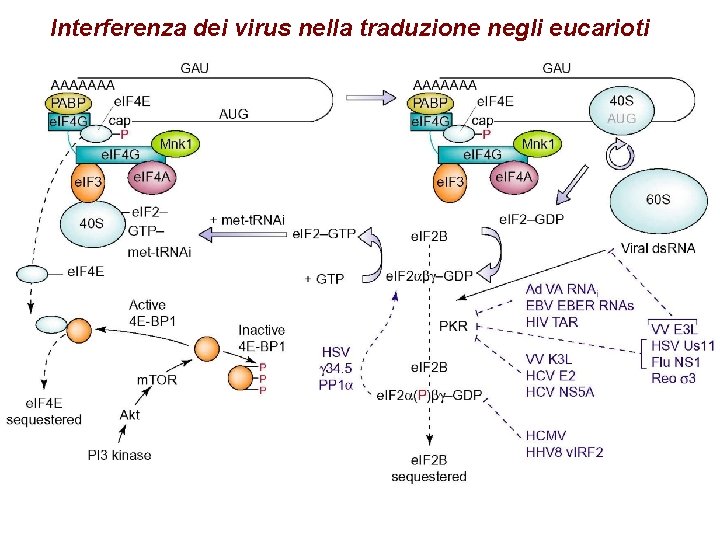
Interferenza dei virus nella traduzione negli eucarioti

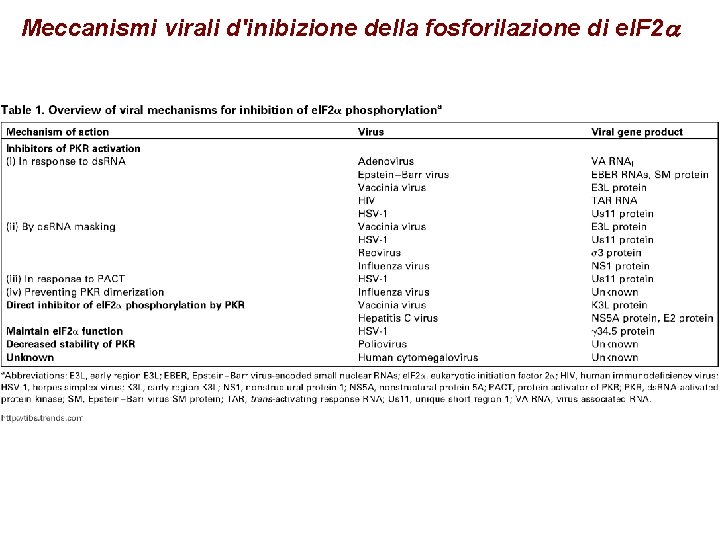
Meccanismi virali d'inibizione della fosforilazione di e. IF 2 a

Regolazione di e. IF 2 a § Fosforilazione su ser 51 in risposta a stress da parte di 4 chinasi: PKR, GCN 2, PERK, HRI § La fosforilazione impedisce il riciclo da parte di e. IF 2 B § La traduzione di alcuni m. RNA è stimolata da bassi livelli di e. IF 2 a attivo § Molti virus hanno sistemi per impedire la fosforilazione di e. IF 2 a

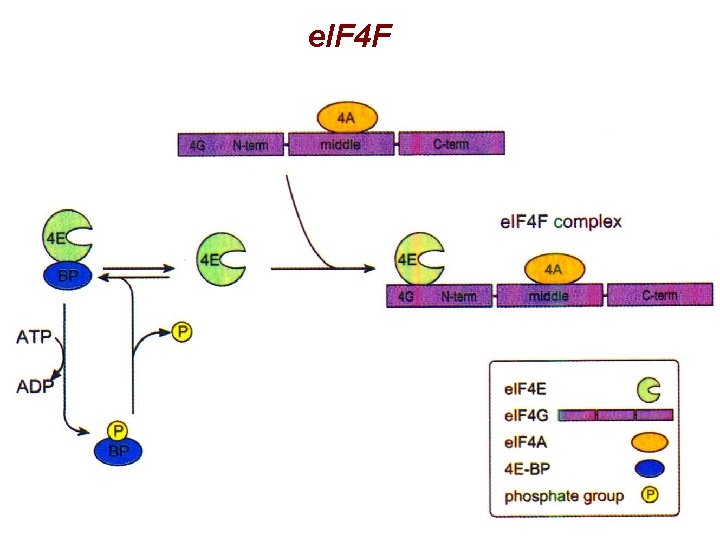
e. IF 4 F

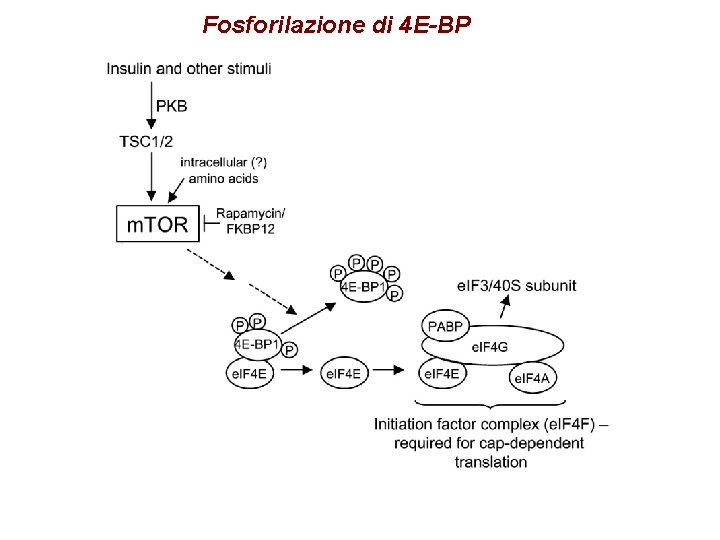
Fosforilazione di 4 E-BP

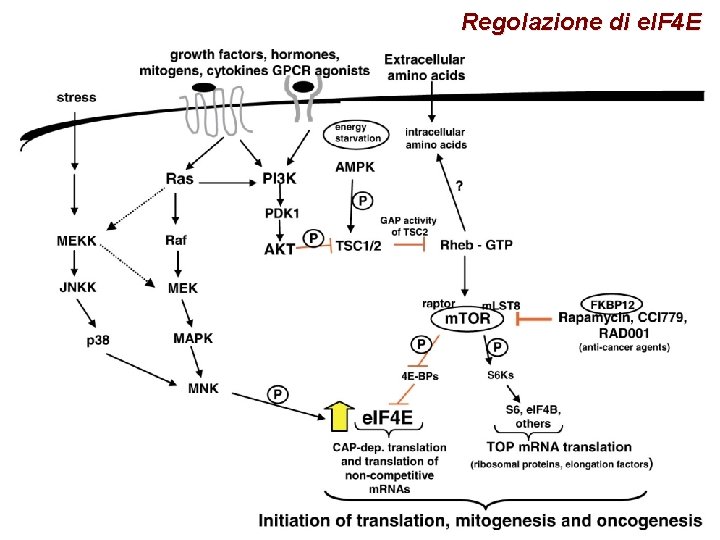
Regolazione di e. IF 4 E

Regolazione e. IF 4 E § Fosforilazione su ser 209 da parte di Mnk 1 (e Mnk 2) correlata con attivazione § Mnk interagiscono con e. IF 4 G e sono attivate dalla via delle MAP chinasi § Doppio KO per Mnk 1 e 2 è normale § e. IF 4 E è inibito dall’interazione con 4 E-BP (1, 2 e 3) § 4 E-BP sono inibite da fosforilazione dipendente dalla via di m. TOR

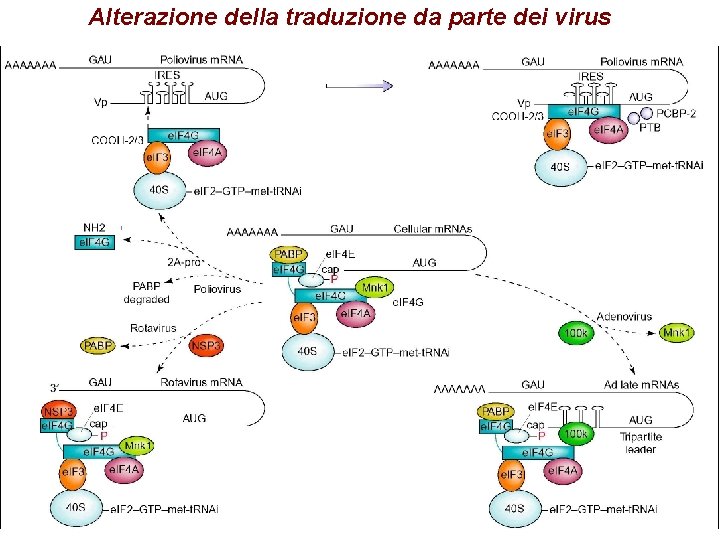
Alterazione della traduzione da parte dei virus

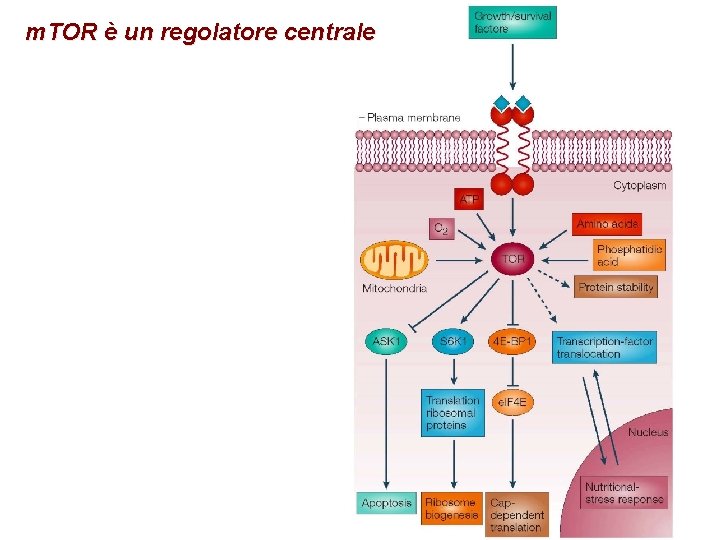
m. TOR è un regolatore centrale

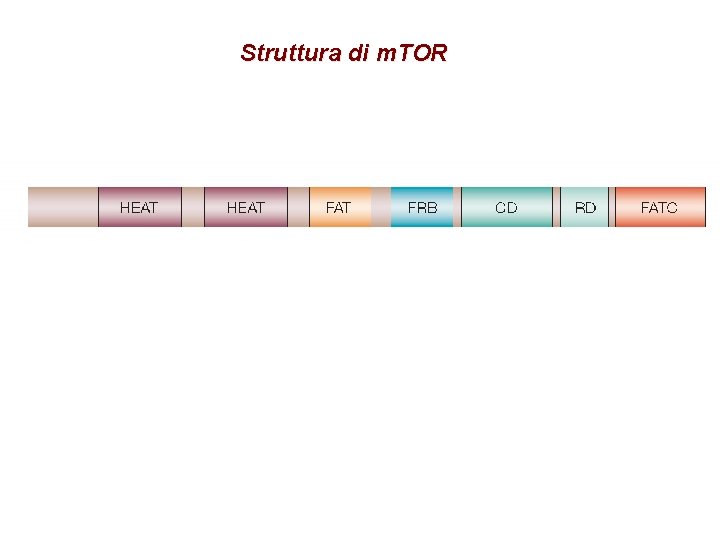
Struttura di m. TOR

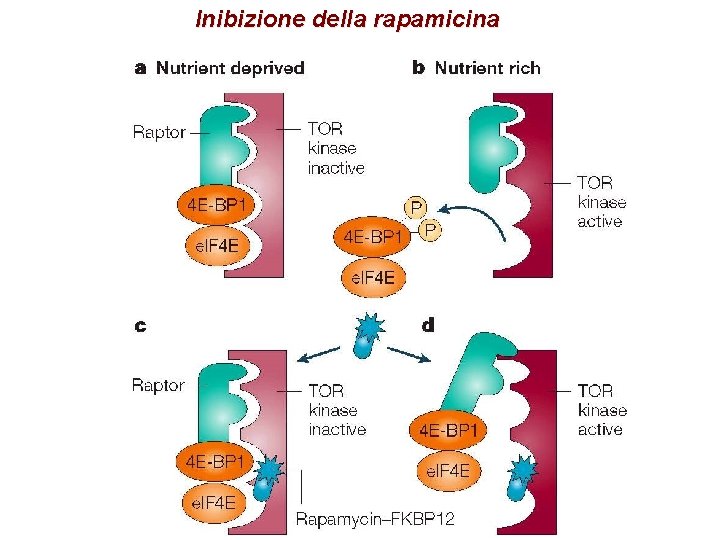
Inibizione della rapamicina

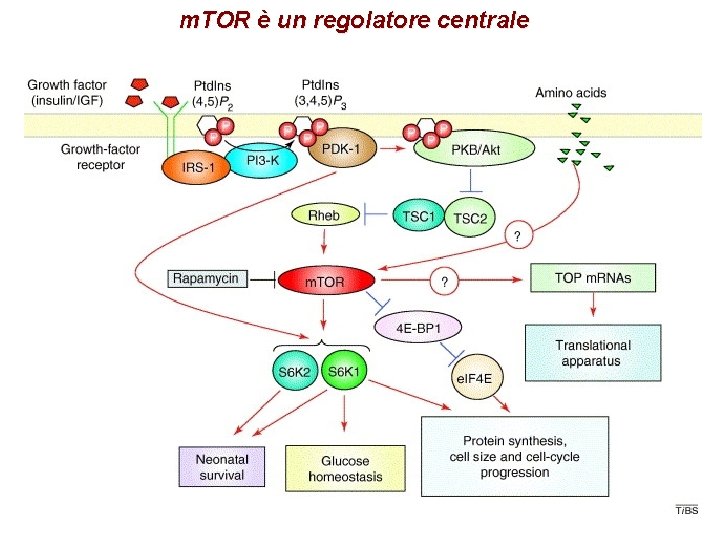
m. TOR è un regolatore centrale

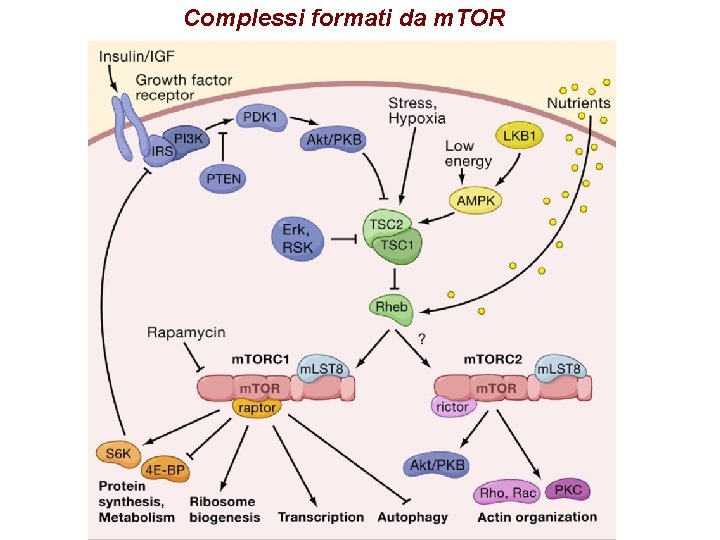
Complessi formati da m. TOR

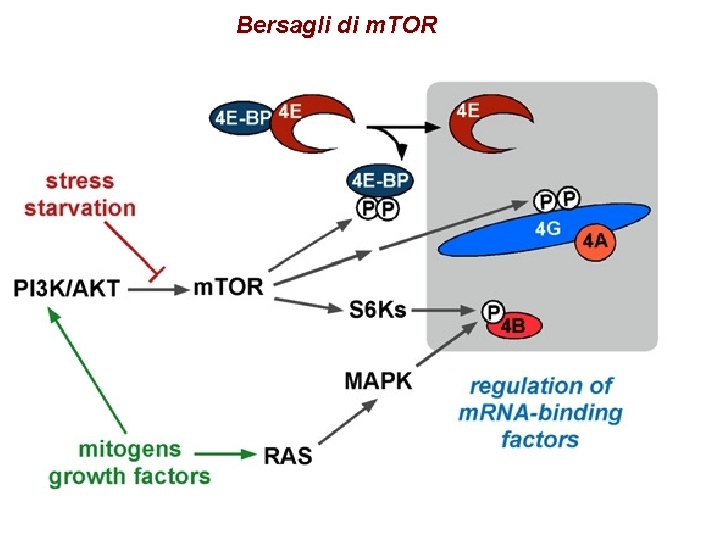
Bersagli di m. TOR

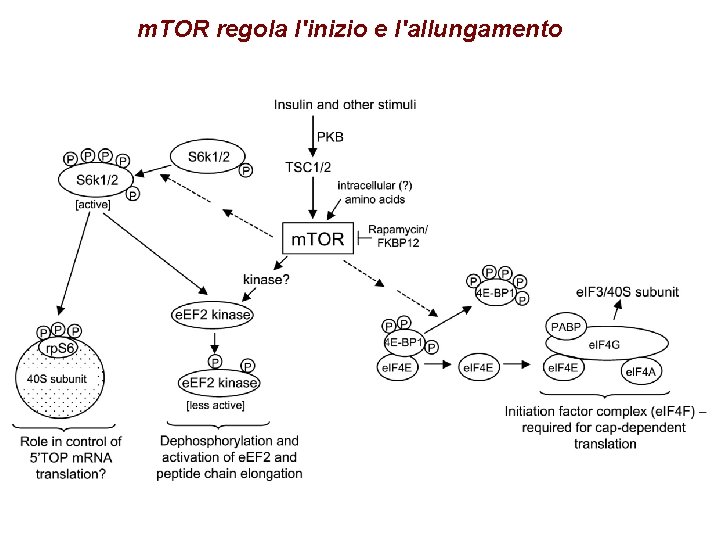
m. TOR regola l'inizio e l'allungamento

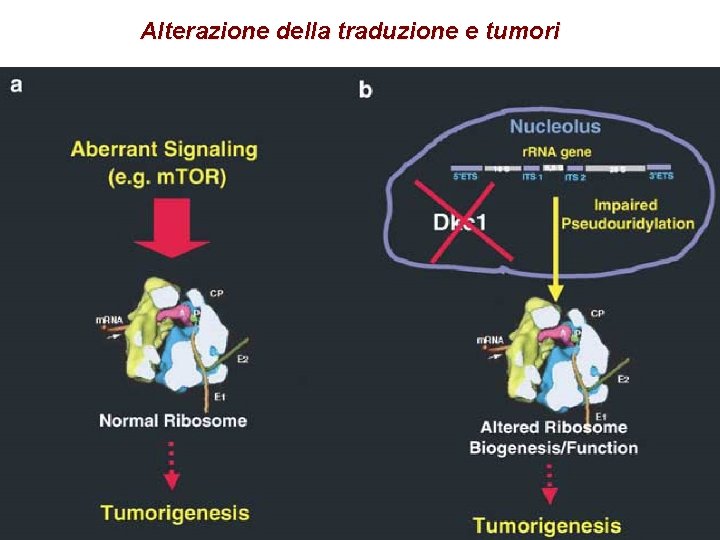
Translation and oncogenesis • Hypertrophic nucleoli were a prominent feature of malignant cell • Ribosome alterations are associated with malignancies • Common cancer-related mutations are found in pathways feeding into the translation machinery • Translation initiation factors are frequently amplified or dysregulated in tumours

Alterazione della traduzione e tumori

Role of ribosome in tumorigenesis defective ribosome QUANTITATIVE/QUALITATIVE ALTERATIONOF TRANSLATION tumorigenesis

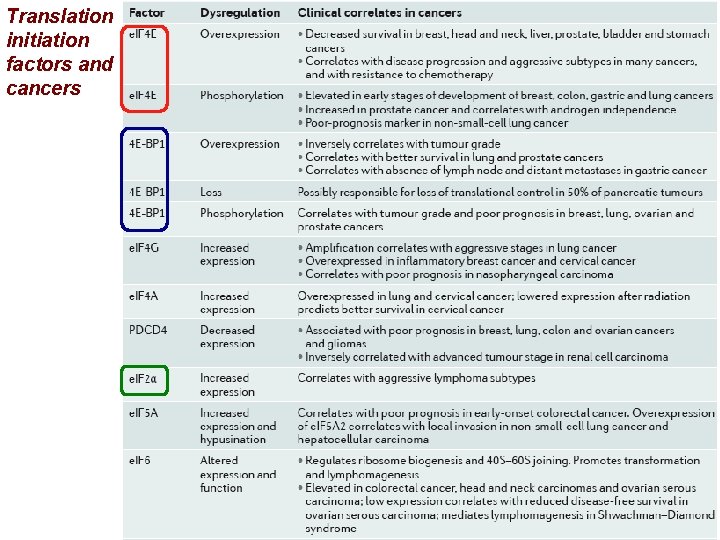
Translation initiation factors and cancers

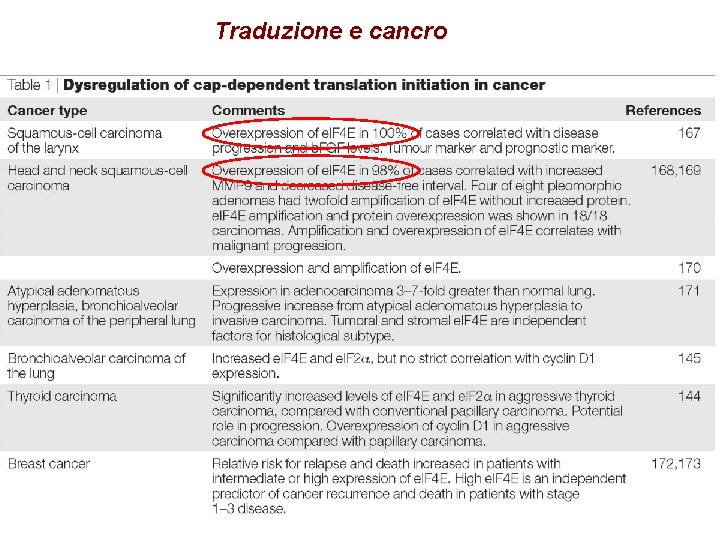
Traduzione e cancro

Regolazione della traduzione • generale • specifica

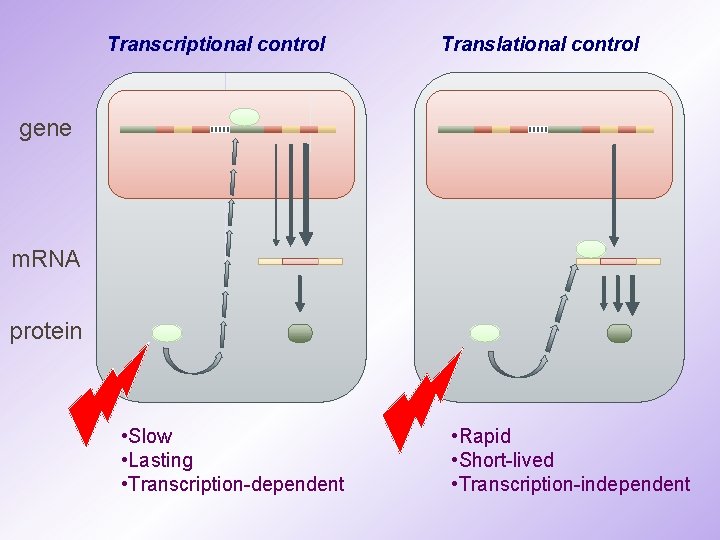
Transcriptional control Translational control gene m. RNA protein • Slow • Lasting • Transcription-dependent • Rapid • Short-lived • Transcription-independent

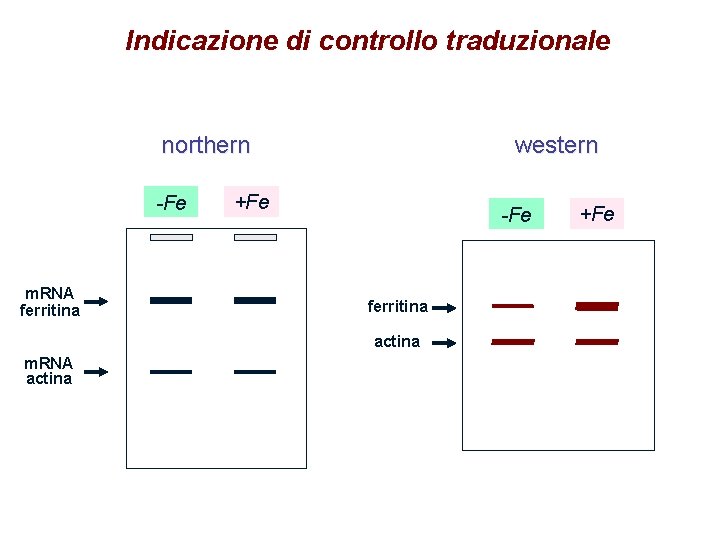
Indicazione di controllo traduzionale northern -Fe m. RNA ferritina western +Fe -Fe ferritina actina m. RNA actina +Fe

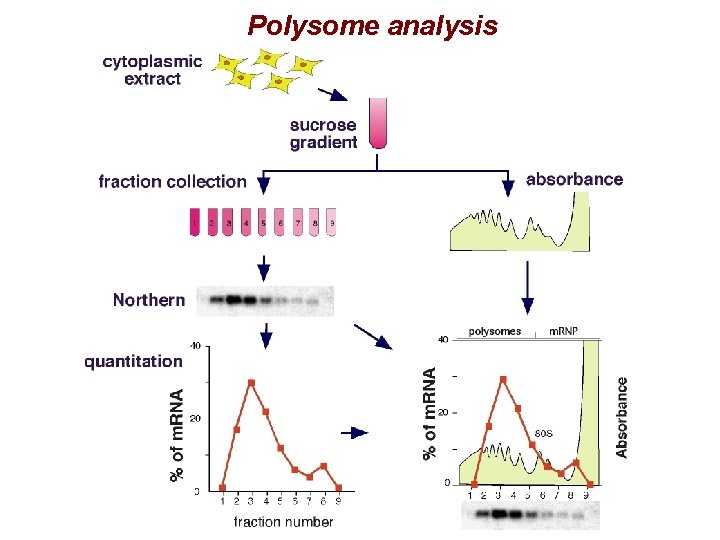
METODI PER STUDIARE IL CONTROLLO TRADUZIONALE • Confronto tra la variazione della quantità di m. RNA e quella del prodotto proteico Øgel poliacrilammide 2 D (DIGE) Øwestern blot • Analisi della stabilità delle proteine mediante l'uso di inibitori della traduzione • Analisi della quantità di m. RNA sui polisomi (distribuzione)

Polysome analysis

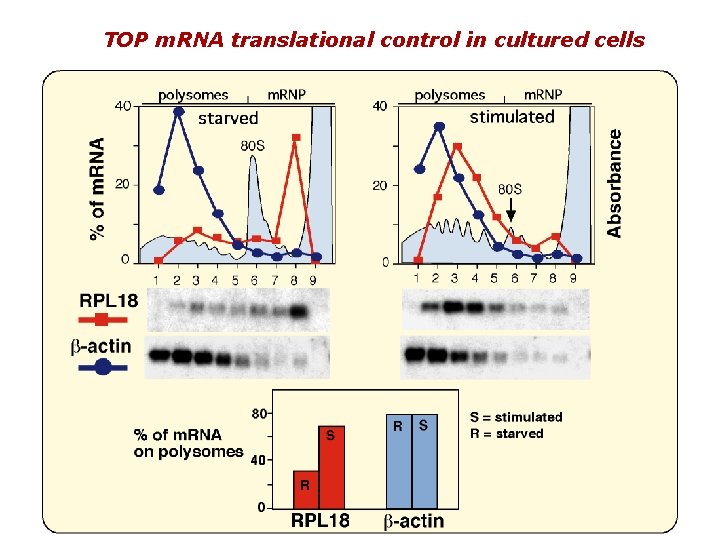
TOP m. RNA translational control in cultured cells

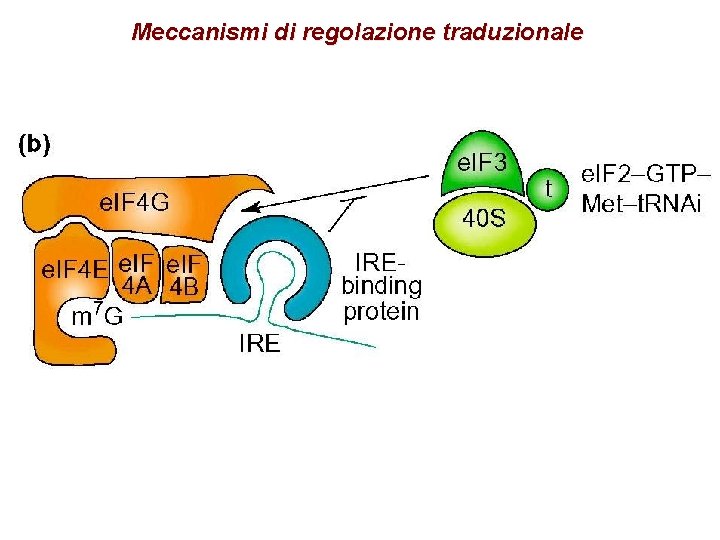
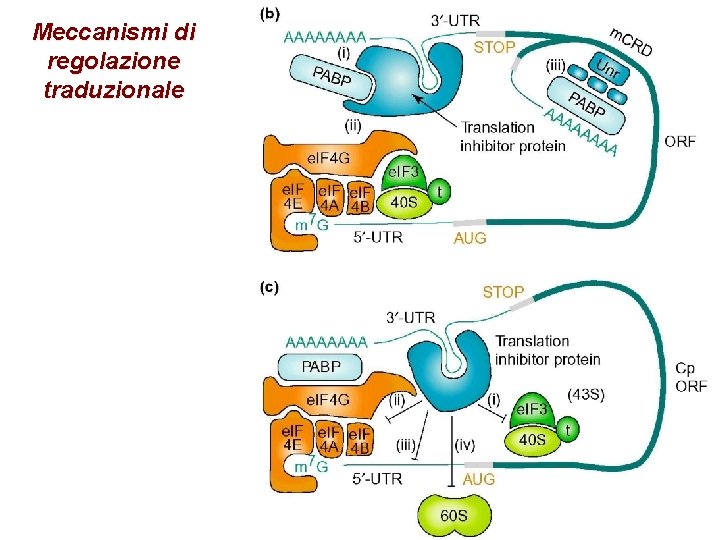
Meccanismi di regolazione traduzionale

Meccanismi di regolazione traduzionale
