Human Genome Center Genome Net n ACCESSION ENTRY














![NQS投入の例 gray{iisaka}19: blastp 1. 4. 9 MP [26 -March-1996] [Build 19: 24: 28 Dec NQS投入の例 gray{iisaka}19: blastp 1. 4. 9 MP [26 -March-1996] [Build 19: 24: 28 Dec](https://slidetodoc.com/presentation_image_h/2c852b836d88455b7dfb83da374e3338/image-31.jpg)
![NQS投入の例(つづき) more options? (? to see manual) [none]: output file? [test. seq. bla]: program: NQS投入の例(つづき) more options? (? to see manual) [none]: output file? [test. seq. bla]: program:](https://slidetodoc.com/presentation_image_h/2c852b836d88455b7dfb83da374e3338/image-32.jpg)





- Slides: 37






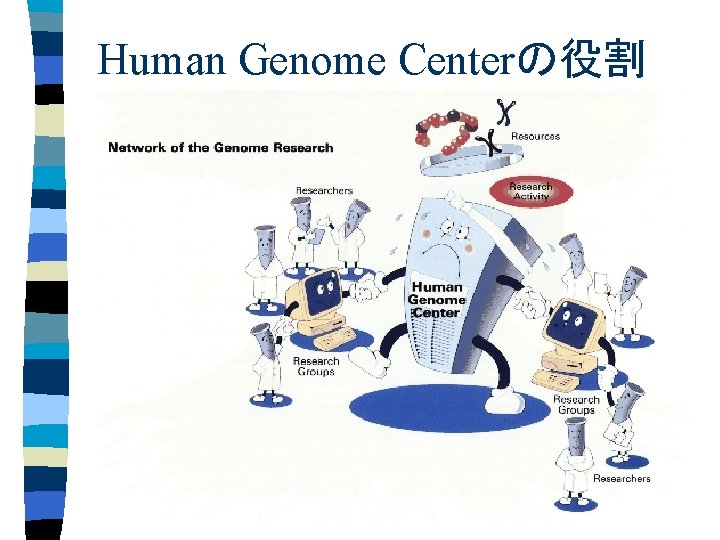
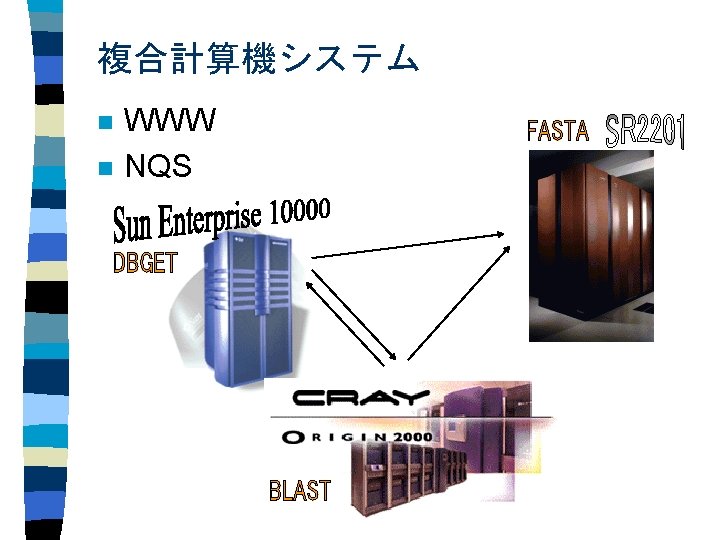
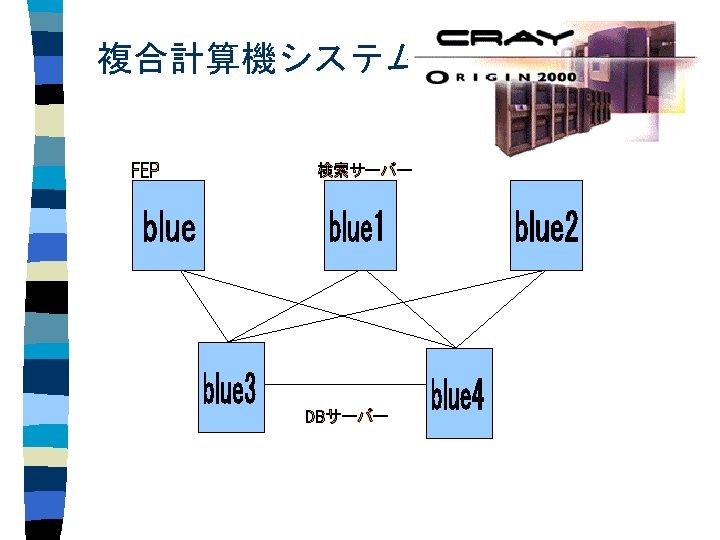
Human Genome Centerの役割

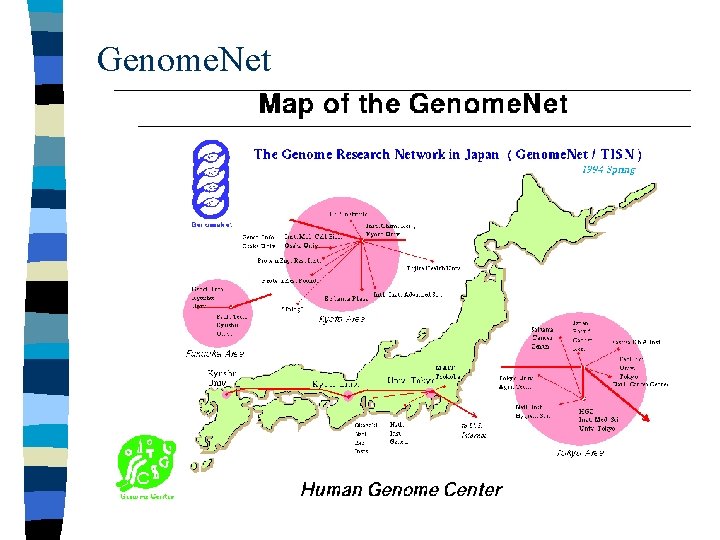
Genome. Net





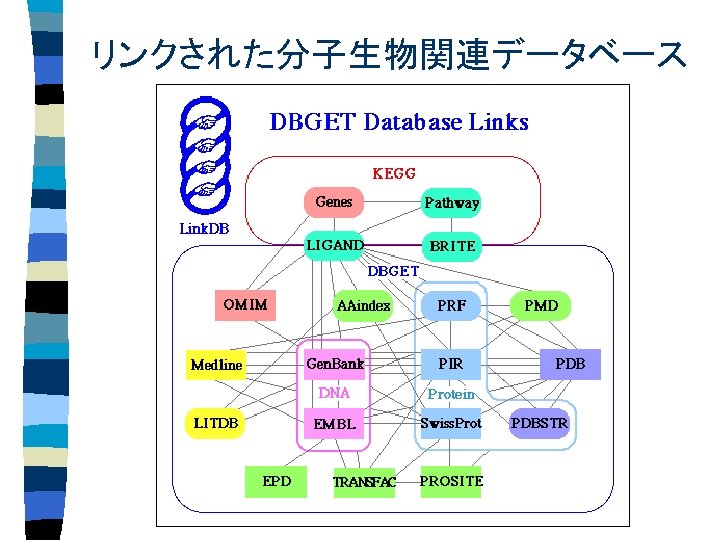
データベースサービス n エントリ名による検索 (ACCESSION, ENTRY など) n キーワードによる検索 – タイトル (DEFINITION, TITLE など) – 参考文献 (REFERENCE 内) – 種・組織 (SOURCE, ORGANISM 内) n n 核酸・アミノ酸類似度による検索 (ORIGIN, SEQUENCE など) リンク先の検索 (データベース内外) LOCUS HSU 88725 2093 bp DNA PRI 19 -MAR-1998 DEFINITION Human type 1 inositol 1, 4, 5 -trisphosphate receptor gene, promoter region and exon 1. ACCESSION U 88725 NID g 1857947 KEYWORDS. SOURCE human. ORGANISM Homo sapiens Eukaryota; Metazoa; Chordata; Vertebrata; Mammalia; Eutheria; Primates; Catarrhini; Hominidae; Homo. REFERENCE 1 (bases 1 to 2093) AUTHORS Deelman, L. E. , Jonk, L. J. C. and Henning, R. H. TITLE The isolation and characterization of the promoter of the human type 1 inositol 1, 4, 5 -trisphosphate receptor JOURNAL Gene 207 (2), 219 -225 (1998) REFERENCE 2 (bases 1 to 2093) AUTHORS Deelman, L. E. , Jonk, L. J. and Henning, R. H. TITLE Direct Submission JOURNAL Submitted (06 -FEB-1997) Clinical Pharmacology, University of Groningen, A. Deusinglaan 1, Groningen 9713 AV, The Netherlands FEATURES Location/Qualifiers source 1. . 2093 /organism="Homo sapiens" /db_xref="taxon: 9606" promoter 1. . 1948 TATA_signal 1922. . 1927 exon 1949. . 2093 /number=1 /product="type 1 inositol 1, 4, 5 -trisphosphate receptor" BASE COUNT 504 a 517 c 554 g 512 t 6 others ORIGIN 1 atcgtgaaca caaactttgt ctatttcaag agctttgccc cttcctggac tgacattaaa 61 tgcttatctg agcaggcaaa tacagtgcac atgtttacat tggggaggta gcggagacta 121 gtagtttagg accagggttc aagactcagc tatttgtgtg actttggagg aagtaattta 181 atcgctttgt gccccccctc agttttctca taagtaaaaa tggcttaata ggatttaatt 。。。 //





Genome. Net n WWW http: //www. genome. ad. jp

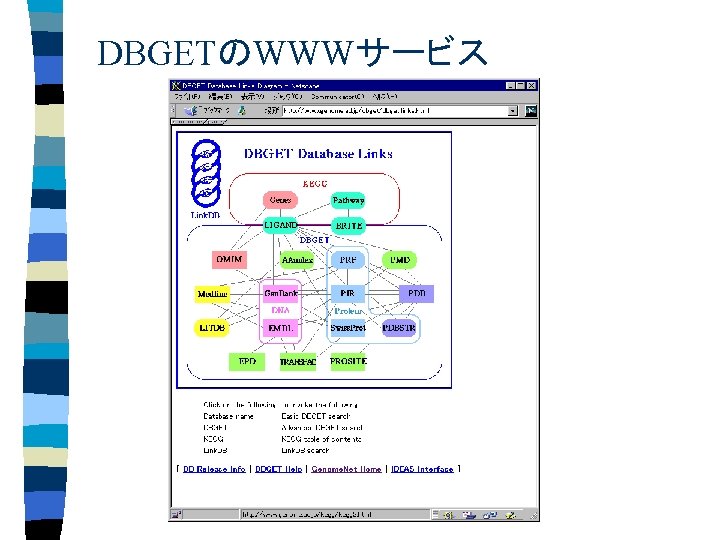
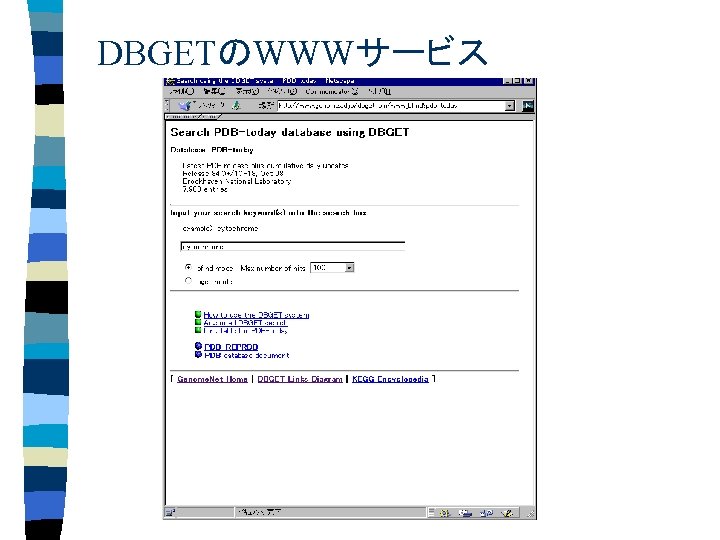
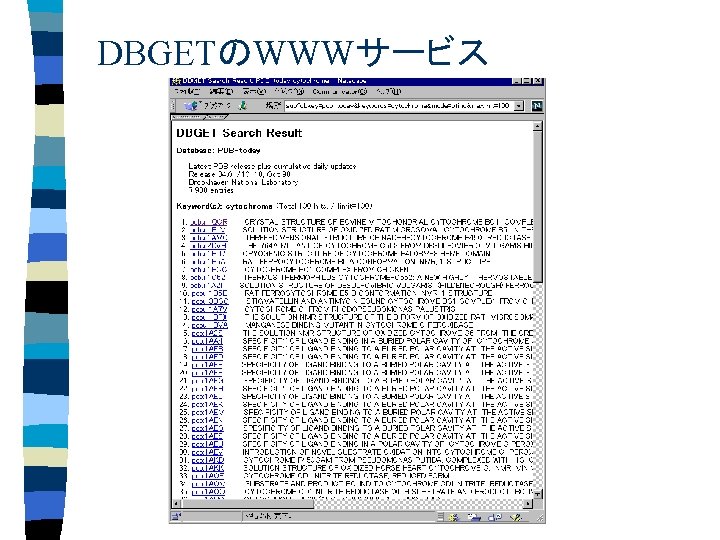
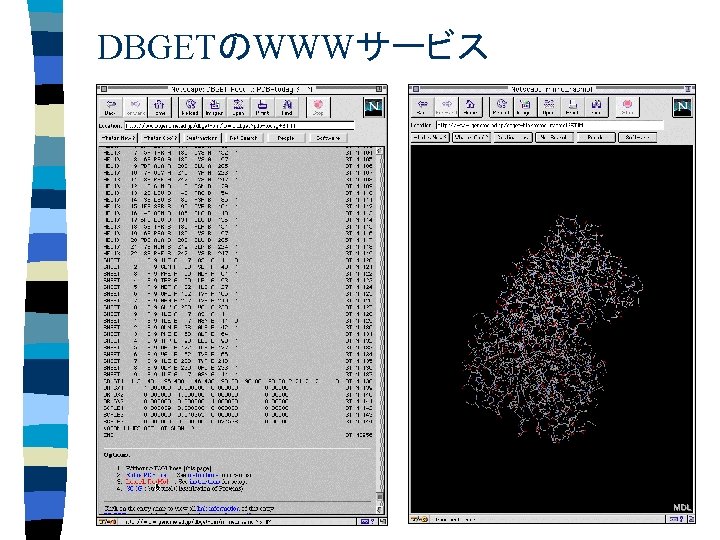
DBGETのWWWサービス

DBGETのWWWサービス

DBGETのWWWサービス

DBGETのWWWサービス


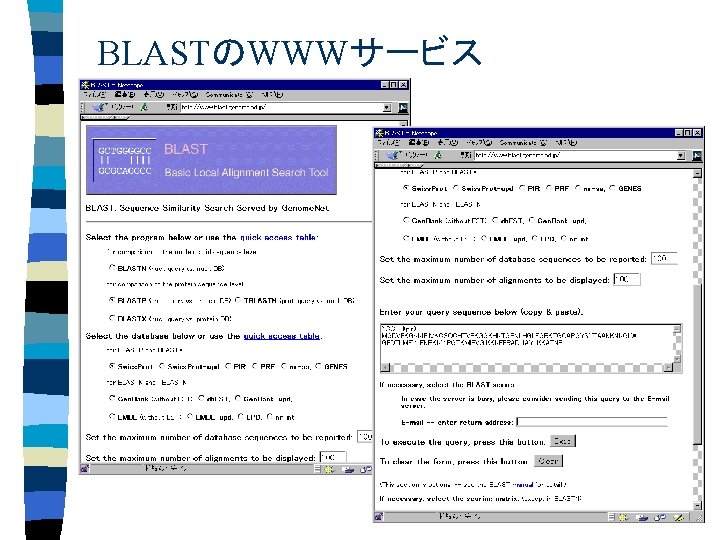
BLASTのWWWサービス

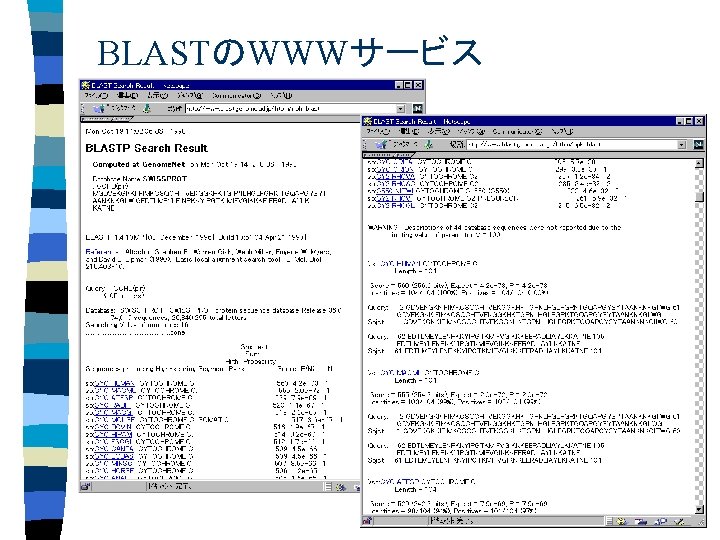
BLASTのWWWサービス


CGI: BLASTを呼び出す(部分) $CMD 1 = "$PROGRAM $dbname input. seq $options -p$NCPUS"; open( JOBF, ">$WORK/blast. job" )|| die "Cannot create Job file"; print JOBF "#!$CPULIMIT /bin/shn" ; print JOBF "$LIMITCMDn"; print JOBF "mkdir $TMPDIR/blast. job. $$n"; print JOBF "cd $TMPDIR/blast. job. $$n"; print JOBF "cat > input. seq <<__EOF__n" ; print JOBF `cat $WORK/blast. input` ; print JOBF "__EOF__n" ; print JOBF "$CMD 1 | $ANCHOR | $FILTER >output 2>/dev/nulln"; print JOBF "rcp output $WWW_HOST: $WORK/blast. resn" ; print JOBF "cd. . ; /sbin/rm -rf $TMPDIR/blast. job. $$ 2>/dev/nulln" ; close(JOBF); chmod( 0755, "$WORK/blast. job" ); system("rsh $BLAST_HOST $WORK/blast. job");




![NQS投入の例 grayiisaka19 blastp 1 4 9 MP 26 March1996 Build 19 24 28 Dec NQS投入の例 gray{iisaka}19: blastp 1. 4. 9 MP [26 -March-1996] [Build 19: 24: 28 Dec](https://slidetodoc.com/presentation_image_h/2c852b836d88455b7dfb83da374e3338/image-31.jpg)
NQS投入の例 gray{iisaka}19: blastp 1. 4. 9 MP [26 -March-1996] [Build 19: 24: 28 Dec 12 1997] Reference: Altschul, Stephen F. , Warren Gish, Webb Miller, Eugene W. Myers, and David J. Lipman (1990). Basic local alignment search tool. J. Mol. Biol. 215: 403 -10. test sequence file or directory: test. seq Choose sequence Library: 1. nr-aa (Rel. 98 -10 -18) 2. swissprot (Rel. 36. 0) 3. swissprot-upd (Rel. 36. 0+/10 -03) 4. pir (Rel. 57. 0) 5. prf (Rel. 98 -09) 6. genpept (Rel. 108. 0) 7. genpept-upd (Rel. 108. 0+/10 -18) 8. pdbstr-upd (Rel. 84. 0+/08 -02) Enter library filename (e. g. prot. lib), or number (e. g. 2): 4
![NQS投入の例つづき more options to see manual none output file test seq bla program NQS投入の例(つづき) more options? (? to see manual) [none]: output file? [test. seq. bla]: program:](https://slidetodoc.com/presentation_image_h/2c852b836d88455b7dfb83da374e3338/image-32.jpg)
NQS投入の例(つづき) more options? (? to see manual) [none]: output file? [test. seq. bla]: program: blastp datalib: pir options: -p 8 query type: protein input file: test. seq output file: test. seq. bla OK? [yes] nqs-181 qsub: INFO Request <22740. gray>: Submitted to queue <blast> by <iisaka(5015)>.

NQS投入のスクリプト(部分) sub qsub_blast { if ($input_ftype eq 'file') { open(QSUB, "| qsub -r $progname -eo -o $output -q $Qname{$queue}") || die; print QSUB "cd $CWD; $progpath/$progname $dbname $seqfile $optionsn"; close(QSUB); } elsif ($input_ftype eq 'directory') { if (! defined(@Files)) { opendir(DIR, $seqfile) || die "Can't access $seqfilen"; @Files = sort readdir(DIR); close(DIR); } foreach $f (@Files) { next if ($f =~ /^. /); print STDERR "$seqfile/$fn"; open(QSUB, "| qsub -r $progname -eo -o $output/$f -q $Qname{$queue}") || die; print QSUB "cd $CWD; $progpath/$progname $dbname $seqfile/$f $optionsn"; close(QSUB); print STDERR "Done. n"; } } }

NQS投入のスクリプト(ファイル時) if ($input_ftype eq 'file') { open(QSUB, "| qsub -r $progname -eo -o $output -q $Qname{$queue}") || die; print QSUB "cd $CWD; $progpath/$progname $dbname $seqfile $optionsn"; close(QSUB); }

NQS投入のスクリプト(ディレクトリ時) } elsif ($input_ftype eq 'directory') { if (! defined(@Files)) { opendir(DIR, $seqfile) || die "Can't access $seqfilen"; @Files = sort readdir(DIR); close(DIR); } foreach $f (@Files) { next if ($f =~ /^. /); print STDERR "$seqfile/$fn"; open(QSUB, "| qsub -r $progname -eo -o $output/$f -q $Qname{$queue}") || die; print QSUB "cd $CWD; $progpath/$progname $dbname $seqfile/$f $optionsn"; close(QSUB); print STDERR "Done. n"; } }

