Genomas Virales y Cromosomas CA Garca Seplveda MD
















- Slides: 34

Genomas Virales y Cromosomas CA García Sepúlveda MD Ph. D Laboratorio de Genómica Viral y Humana Facultad de Medicina, Universidad Autónoma de San Luis Potosí 1

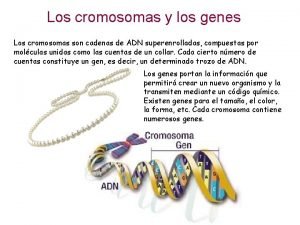
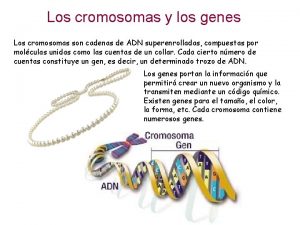
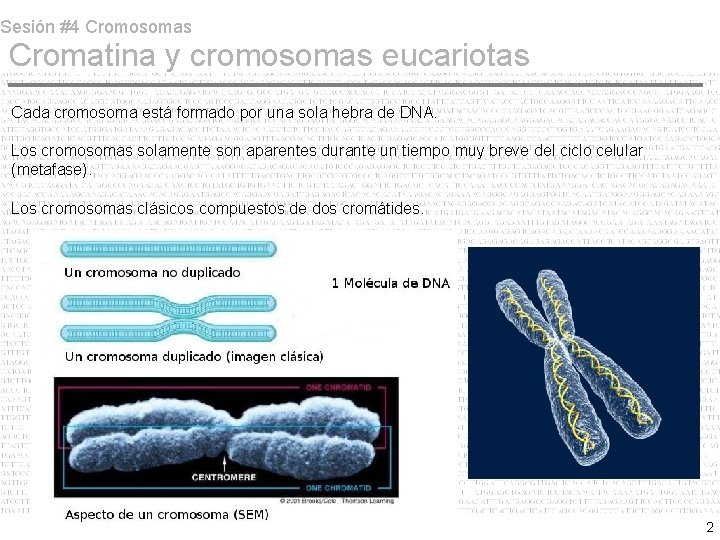
Sesión #4 Cromosomas Cromatina y cromosomas eucariotas Cada cromosoma está formado por una sola hebra de DNA. Los cromosomas solamente son aparentes durante un tiempo muy breve del ciclo celular (metafase). Los cromosomas clásicos compuestos de dos cromátides. 2

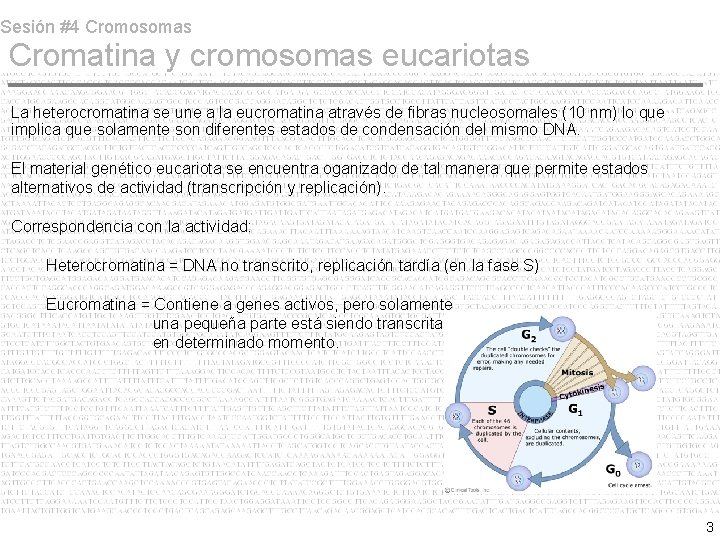
Sesión #4 Cromosomas Cromatina y cromosomas eucariotas La heterocromatina se une a la eucromatina através de fibras nucleosomales (10 nm) lo que implica que solamente son diferentes estados de condensación del mismo DNA. El material genético eucariota se encuentra oganizado de tal manera que permite estados alternativos de actividad (transcripción y replicación). Correspondencia con la actividad: Heterocromatina = DNA no transcrito, replicación tardía (en la fase S) Eucromatina = Contiene a genes activos, pero solamente una pequeña parte está siendo transcrita en determinado momento. 3

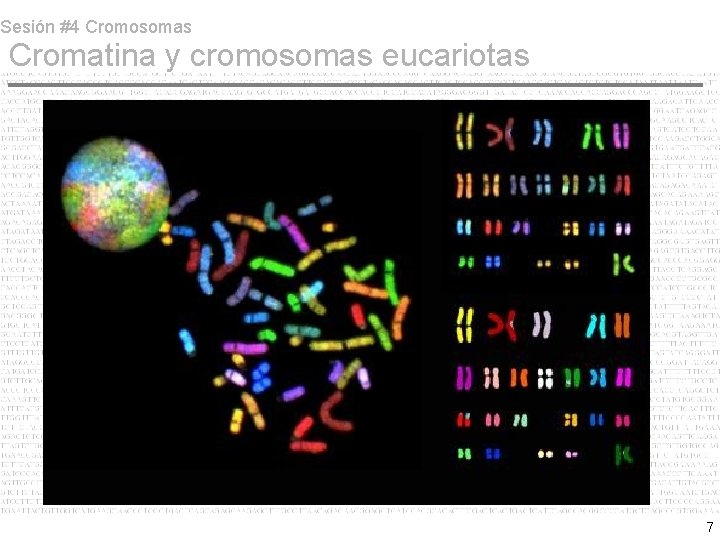
Sesión #4 Cromosomas Cromatina y cromosomas eucariotas A nivel individual, los cromosomas adoptan ultraestructuras reproducibles y predictibles. Los cromosomas originalmente definidos en base a su tamaño y localización del centrómero. Hoy en día los cromosomas son identificados en base al patrón de bandeo (bandas Giemsa o bandas G). . . o más recientemente por FISH. 4

Sesión #4 Cromosomas Cromatina y cromosomas eucariotas Las bandas son estructuras grandes (10 x 106 bp de DNA). Cientos de genes en cada banda. Técnica de enorme utilidad práctica, mecanismo sigue siendo un misterio. Tinte Giemsa pinta por igual al cromosoma no tratado (tripsina seguida de giemsa), pero de manera diferente al tratado. . . sugiriendo que el tratamiento expone (o elimina) ciertas propiedades fisicoquímicas. Bandas obscuras ricas en AT y pobres en genes (Z-DNA) 5

Sesión #4 Cromosomas Cromatina y cromosomas eucariotas 6

Sesión #4 Cromosomas Cromatina y cromosomas eucariotas 7

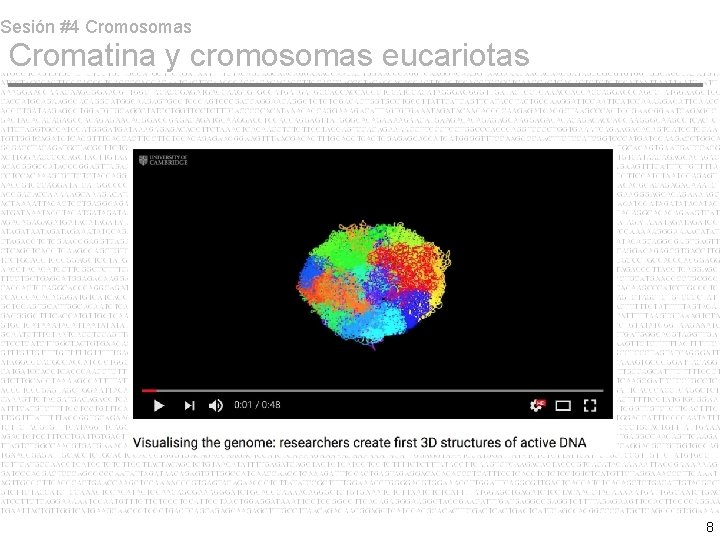
Sesión #4 Cromosomas Cromatina y cromosomas eucariotas 8

Sesión #4 Cromosomas escobillón La expresión génica puede ser visualizada en algunos raros casos durante la meiosis pero durante esta fase del ciclo suele haber muy poca expresión génica. En ciertos casos, algunos organismos como Notophthalmus viridescens (tritón) detienen el proceso meiótico durante varios meses. Durante esta fase, los cromosomas adoptan una forma elongada facilmente visible al MO. Estos cromosomas constituyen bivalentes meióticos consistentes en dos pares de cromátidas hermanas y se llaman cromosomas en escobillon o “lampbrush”. Los cromosomas en escobillón de N. viridescens miden entre 400 y 800 nm. 9

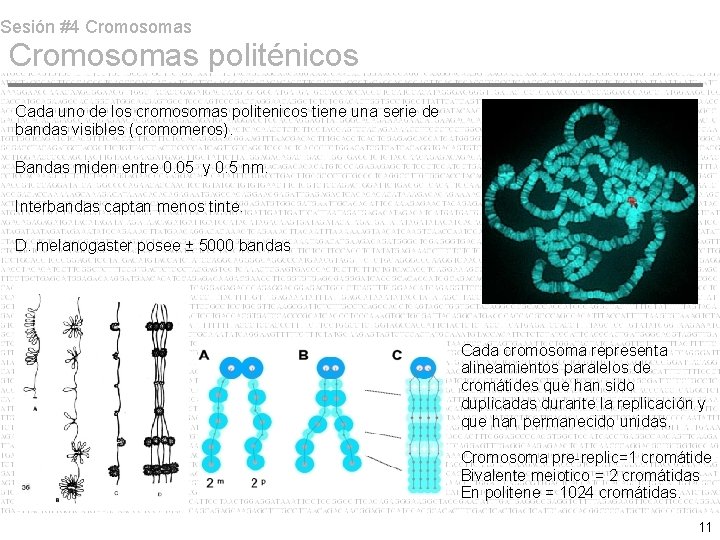
Sesión #4 Cromosomas politénicos Los núcleos de interfase (durante el politeno) de las larvas de algunos dípteros contienen cromosomas con diámetros superiores a los normales. Resultado de endomitosis (replicación sin división celular). Ejemplo clásico de los cromosomas politénicos de las glandulas salivales de D. melanogaster. Ventaja transcripcional enorme, necesario en la larva para producir cantidades industriales de pegamento antes de la pupación. D. melanogaster posee cuatro cromosomas agregados en un cromocentro de heterocromatina (contiene el cromosoma Y de machos). El complejo tiene una dimensión de ± 2000 nm. El DNA completamente extendido mediría ± 40, 000 nm. PR = ± 20. 10

Sesión #4 Cromosomas politénicos Cada uno de los cromosomas politenicos tiene una serie de bandas visibles (cromomeros). Bandas miden entre 0. 05 y 0. 5 nm. Interbandas captan menos tinte. D. melanogaster posee ± 5000 bandas Cada cromosoma representa alineamientos paralelos de cromátides que han sido duplicadas durante la replicación y que han permanecido unidas. Cromosoma pre-replic=1 cromátide Bivalente meiotico = 2 cromátidas En politene = 1024 cromátidas. 11

Sesión #4 Cromosomas politénicos Los sitios activos (de expresión génica) pueden ser fácilmente visualizados en los cromosomas politénicos. Estos sitios provocan ensanchamientos de las bandas conocidos como “Puffs”. Término correcto = Anillos de Balbiani. Representan la extrusión del material genético como consecuencia de su des-empaquetamiento para expresar los genes codficados en ellos = Síntesis de RNA. 12

Sesión #4 Cromosomas politénicos Los anillos de Balbiani pueden ser provocados por la expresión de un solo gen. Las características tanto de los Cromosomas en escobillón como de los cromosomas politénicos implican que el material genético debe ser descompactado para poder realizar su función (transcripción y replicación). 13

Sesión #4 Cromosomas eucariotas Los cromosomas de metafase poseen tres unidades estructurales funcionales: • Dos cromátides hermanas en cuyos brazos se codifica la información genética. • Un centrómero, ultraestructura encargada del tráfico de las cromátides. • Cuatro telómeros, ultraestructura encargada de sellar la información de las cromátides y permitir su replicación integra. 14

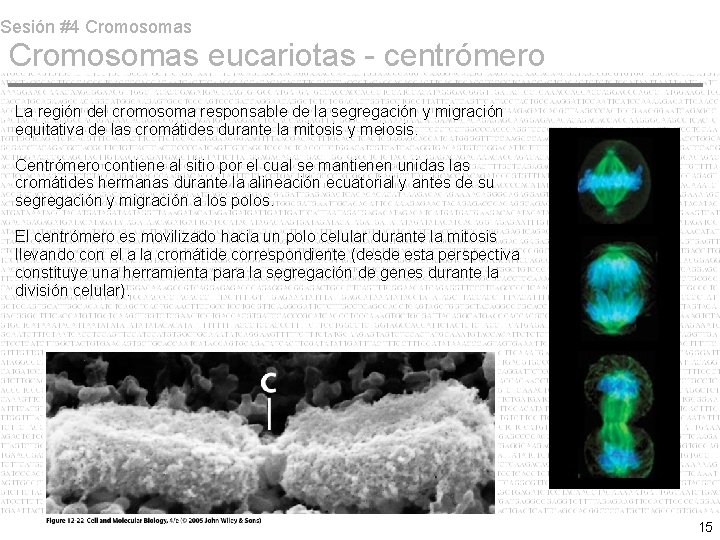
Sesión #4 Cromosomas eucariotas - centrómero La región del cromosoma responsable de la segregación y migración equitativa de las cromátides durante la mitosis y meiosis. Centrómero contiene al sitio por el cual se mantienen unidas las cromátides hermanas durante la alineación ecuatorial y antes de su segregación y migración a los polos. El centrómero es movilizado hacia un polo celular durante la mitosis llevando con el a la cromátide correspondiente (desde esta perspectiva constituye una herramienta para la segregación de genes durante la división celular). 15

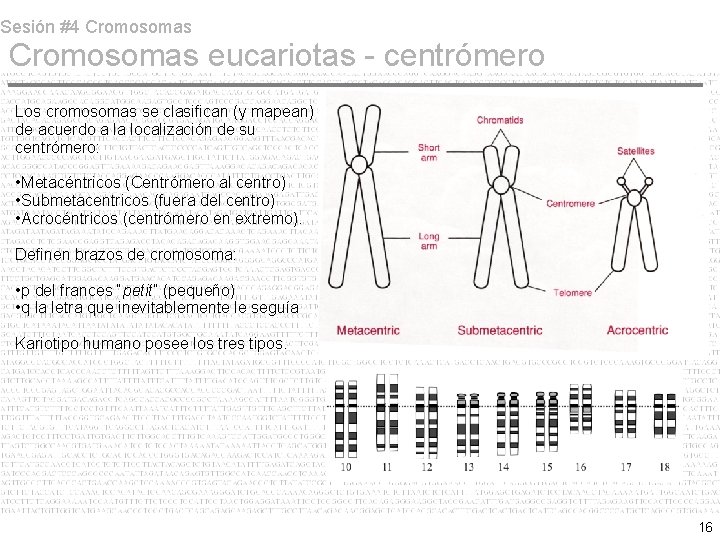
Sesión #4 Cromosomas eucariotas - centrómero Los cromosomas se clasifican (y mapean) de acuerdo a la localización de su centrómero: • Metacéntricos (Centrómero al centro) • Submetacentricos (fuera del centro) • Acrocéntricos (centrómero en extremo). Definen brazos de cromosoma: • p del frances “petit” (pequeño) • q la letra que inevitablemente le seguía Kariotipo humano posee los tres tipos. 16

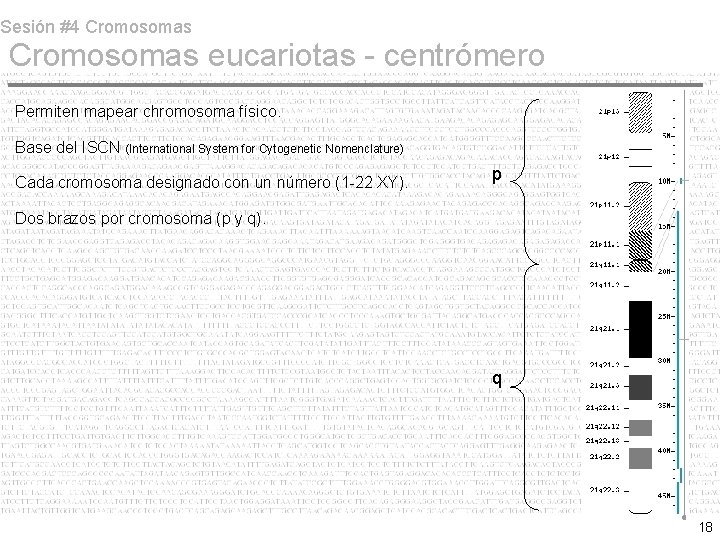
Sesión #4 Cromosomas eucariotas - centrómero Permiten mapear chromosoma físico. Base del ISCN (International System for Cytogenetic Nomenclature) Cada cromosoma designado con un número (1 -22 XY). 21 17

Sesión #4 Cromosomas eucariotas - centrómero Permiten mapear chromosoma físico. Base del ISCN (International System for Cytogenetic Nomenclature) Cada cromosoma designado con un número (1 -22 XY). p Dos brazos por cromosoma (p y q). q 18

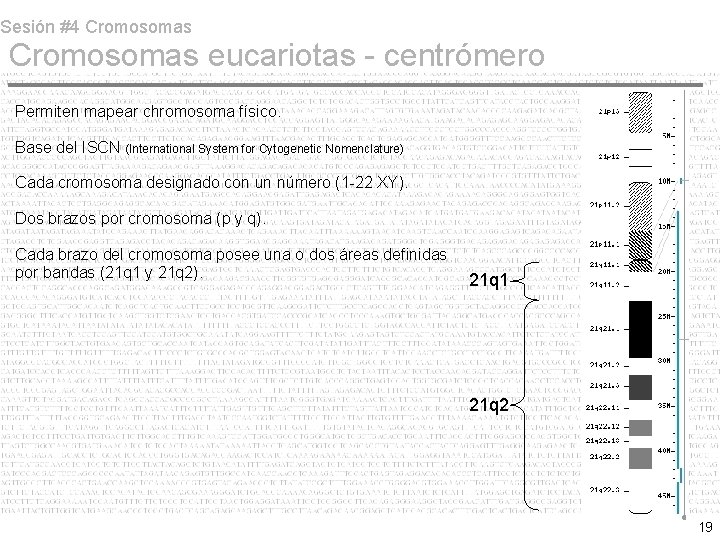
Sesión #4 Cromosomas eucariotas - centrómero Permiten mapear chromosoma físico. Base del ISCN (International System for Cytogenetic Nomenclature) Cada cromosoma designado con un número (1 -22 XY). Dos brazos por cromosoma (p y q). Cada brazo del cromosoma posee una o dos áreas definidas por bandas (21 q 1 y 21 q 2). 21 q 1 21 q 2 19

Sesión #4 Cromosomas eucariotas - centrómero Permiten mapear chromosoma físico. Base del ISCN (International System for Cytogenetic Nomenclature) Cada cromosoma designado con un número (1 -22 XY). Dos brazos por cromosoma (p y q). Cada brazo del cromosoma posee una o dos áreas definidas por bandas (21 q 1 y 21 q 2). 21 q 1 Cada area posee subdivisiones inferiores (21 q 11. 1 y 21 q 11. 2) 20

Sesión #4 Cromosomas eucariotas - centrómero Permiten mapear chromosoma físico. Base del ISCN (International System for Cytogenetic Nomenclature) Cada cromosoma designado con un número (1 -22 XY). Dos brazos por cromosoma (p y q). Cada brazo del cromosoma posee una o dos áreas definidas por bandas (21 q 1 y 21 q 2). 21 q 1 Cada area posee subdivisiones inferiores (21 q 11. 1 y 21 q 11. 2) Permite resumir anomalias citogenéticas 46, XX, del(21 q 11. 2) 21

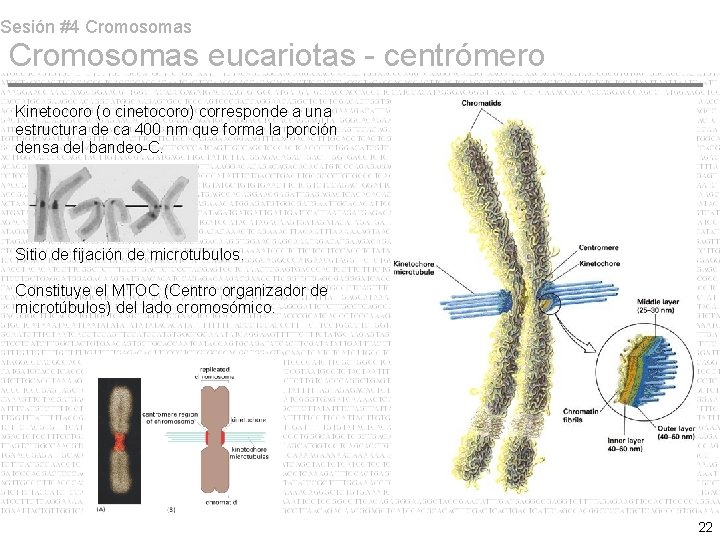
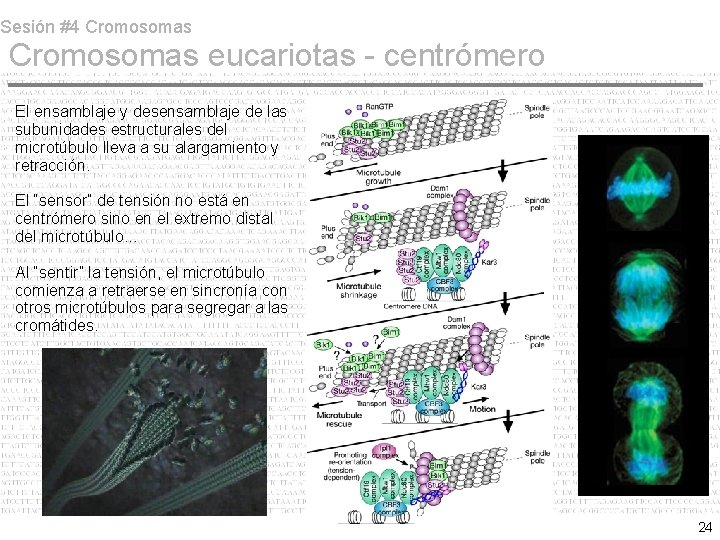
Sesión #4 Cromosomas eucariotas - centrómero Kinetocoro (o cinetocoro) corresponde a una estructura de ca 400 nm que forma la porción densa del bandeo-C. Sitio de fijación de microtubulos. Constituye el MTOC (Centro organizador de microtúbulos) del lado cromosómico. 22

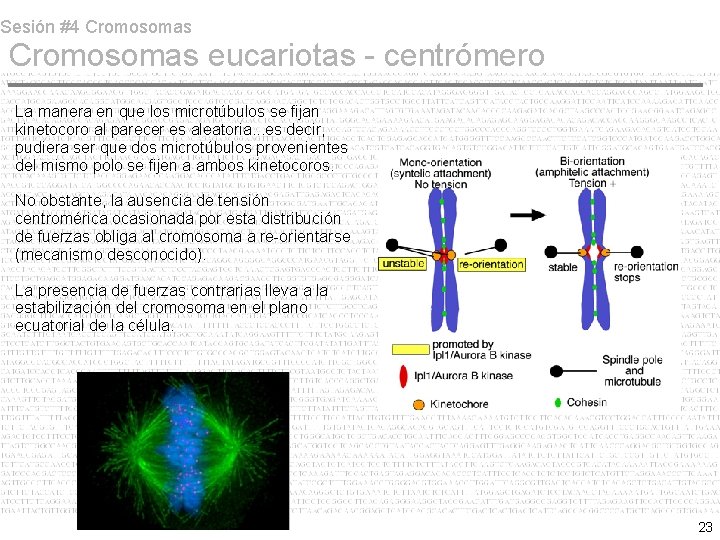
Sesión #4 Cromosomas eucariotas - centrómero La manera en que los microtúbulos se fijan kinetocoro al parecer es aleatoria. . . es decir, pudiera ser que dos microtúbulos provenientes del mismo polo se fijen a ambos kinetocoros. No obstante, la ausencia de tensión centromérica ocasionada por esta distribución de fuerzas obliga al cromosoma a re-orientarse (mecanismo desconocido). La presencia de fuerzas contrarias lleva a la estabilización del cromosoma en el plano ecuatorial de la célula. 23

Sesión #4 Cromosomas eucariotas - centrómero El ensamblaje y desensamblaje de las subunidades estructurales del microtúbulo lleva a su alargamiento y retracción. El “sensor” de tensión no está en centrómero sino en el extremo distal del microtúbulo. . . Al “sentir” la tensión, el microtúbulo comienza a retraerse en sincronía con otros microtúbulos para segregar a las cromátides. 24

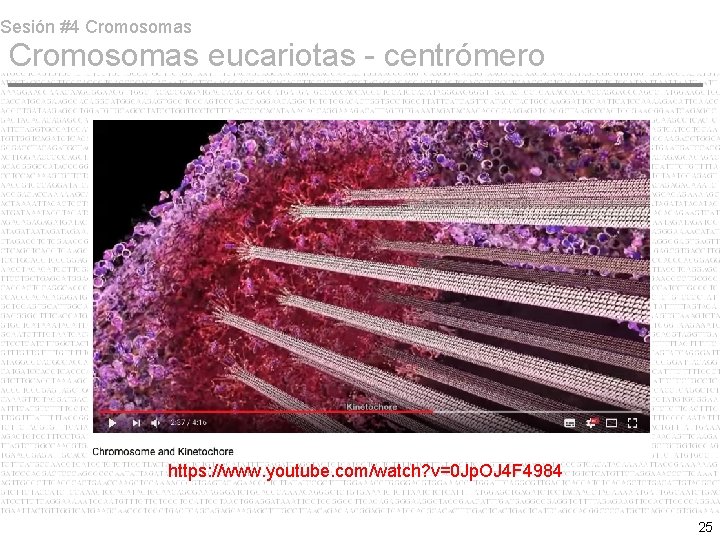
Sesión #4 Cromosomas eucariotas - centrómero https: //www. youtube. com/watch? v=0 Jp. OJ 4 F 4984 25

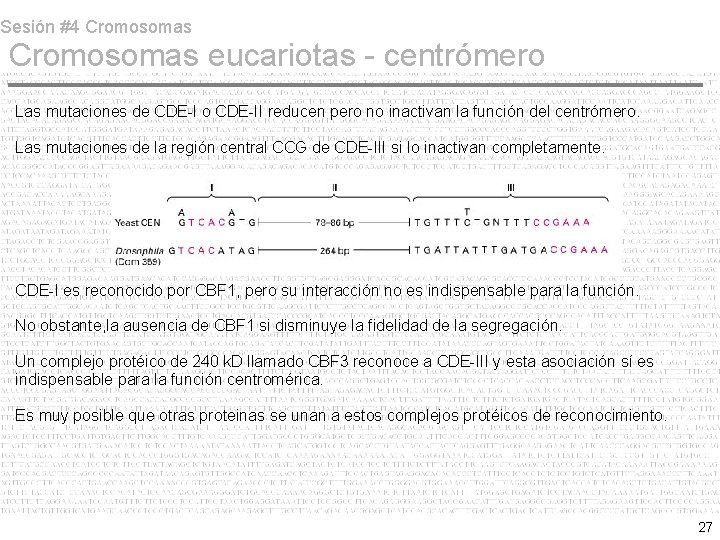
Sesión #4 Cromosomas eucariotas - centrómero Los centrómeros poseen fragments de DNA llamados secuencias o elementos CEN inicialmente caracterizados en S. cerevisiae. La levadura que no poseía kinetocoros pero que sí presentaba segregación y migración mitótica. Una región de 120 bp resistente a nucleasas. Tres regiones indispensables para el funcionamiento del centrómero. Región I, elemento CDE-I : Secuencia de 9 bp conservada en extremo 5’. Región II, elemento CDE-II: Secuencia variable pero tamaño constante, 80 a 90 bp, >90% AT Función más asociada a tamaño que secuencia Contiene secuencias parecidas a STR (DNA satélite). Secuencia de DNA genera contorsiones helicoidales (z. DNA) Región III, elemento CDE-III: Secuencia de 11 bp, altamente conservada. 26

Sesión #4 Cromosomas eucariotas - centrómero Las mutaciones de CDE-I o CDE-II reducen pero no inactivan la función del centrómero. Las mutaciones de la región central CCG de CDE-III si lo inactivan completamente. CDE-I es reconocido por CBF 1, pero su interacción no es indispensable para la función. No obstante, la ausencia de CBF 1 si disminuye la fidelidad de la segregación. Un complejo protéico de 240 k. D llamado CBF 3 reconoce a CDE-III y esta asociación sí es indispensable para la función centromérica. Es muy posible que otras proteinas se unan a estos complejos protéicos de reconocimiento. 27

Sesión #4 Cromosomas eucariotas - telómero Estructura encargada de sellar extremos del cromosoma. Los extremos de moléculas de DNA lineares son “pegajosos” e inestables lo que ocasiona las estructuras anómalas observadas cuando se rompen los cromosomas. Localizados en los extremos terminales de los cromosomas. Brindan estabilidad a las moléculas de DNA lineares que son nuestros cromosomas. P. Ej. Sobrevida de moléculas de DNA en S. cerevisiae. Los plasmidos lineares son inestables y rápidamente degradados dentro de S. cerevisiae. La transferencia de secuencias teloméricas de otro organismo estabiliza a las moléculas lineares. 28

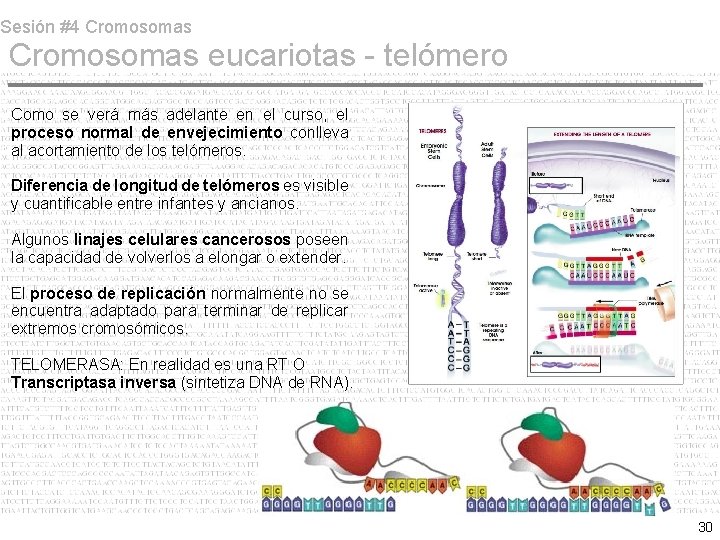
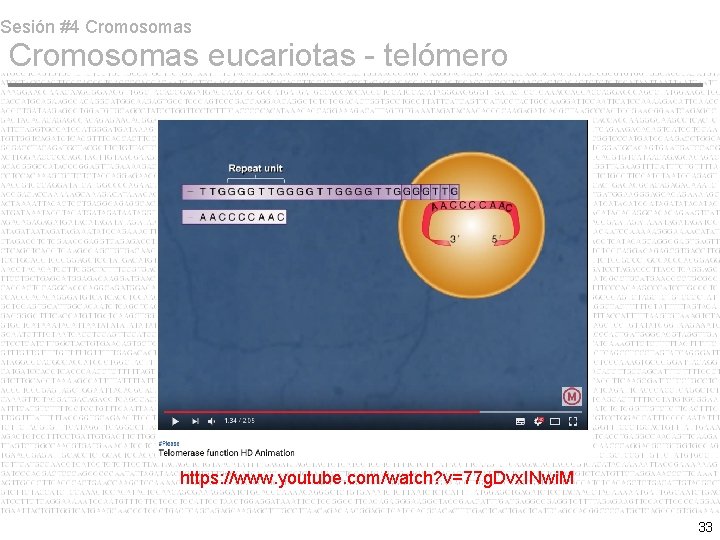
Sesión #4 Cromosomas eucariotas - telómero Varias secuencias teloméricas se han aislado a partir de organismos tan diversos como flagelados (Tetrahymena) y humanos. El mismo tipo de secuencia se encuentra presente en plantas y animales. Por ello la construcción y la función de los telómeros parece seguir un principio universal que ha sido conservado a lo largo de la evolución. Secuencias de DNA teloméricas consisten en repeticiones cortas en serie (tandem) o STRs. De hecho la secuencia telomérica del r. DNA extracromosómico linear natural de Tetrahymena fue la responsable de la estabilización de plasmidos lineares en S. cerevisiae. Función telomérica depende de dos propiedades: Capacidad de alargarse y acortarse. Adopción de conformaciones extrañas (loops y tetraplex). 29

Sesión #4 Cromosomas eucariotas - telómero Como se verá más adelante en el curso, el proceso normal de envejecimiento conlleva al acortamiento de los telómeros. Diferencia de longitud de telómeros es visible y cuantificable entre infantes y ancianos. Algunos linajes celulares cancerosos poseen la capacidad de volverlos a elongar o extender. El proceso de replicación normalmente no se encuentra adaptado para terminar de replicar extremos cromosómicos. TELOMERASA: En realidad es una RT O Transcriptasa inversa (sintetiza DNA de RNA). 30

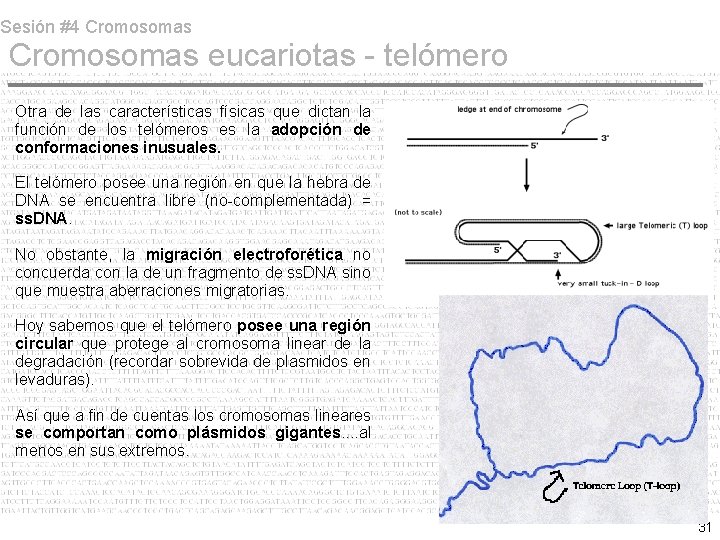
Sesión #4 Cromosomas eucariotas - telómero Otra de las características físicas que dictan la función de los telómeros es la adopción de conformaciones inusuales. El telómero posee una región en que la hebra de DNA se encuentra libre (no-complementada) = ss. DNA. No obstante, la migración electroforética no concuerda con la de un fragmento de ss. DNA sino que muestra aberraciones migratorias. Hoy sabemos que el telómero posee una región circular que protege al cromosoma linear de la degradación (recordar sobrevida de plasmidos en levaduras). Así que a fin de cuentas los cromosomas lineares se comportan como plásmidos gigantes. . al menos en sus extremos. 31

Sesión #4 Cromosomas eucariotas - telómero Este lazo (loop) de 5 a 10 kb no le permite el acceso a nucleasas que degraden el extremo del DNA linear y su formación es catalizada por proteínas (complejos TRF 1 y TRF 2). 32

Sesión #4 Cromosomas eucariotas - telómero https: //www. youtube. com/watch? v=77 g. Dvx. INwi. M 33

Sesión #4 Cromosomas Y para que tantos cromosomas? https: //www. youtube. com/watch? v=n. Ku. JZy. ZUh 2 M 34
 Moleculas de adhesion
Moleculas de adhesion Genomas
Genomas Garca la carolina
Garca la carolina 13 linhas para viver
13 linhas para viver Andrea garca
Andrea garca Mrquez
Mrquez Eligas garça
Eligas garça Ricardo garca
Ricardo garca Ricardo garca
Ricardo garca Gabriel garca
Gabriel garca Repuestos garca
Repuestos garca Andrea garca
Andrea garca Garça tipos
Garça tipos Rhizomer
Rhizomer Colorado garca
Colorado garca 13 conseil de la vie
13 conseil de la vie Ricardo garca
Ricardo garca A mayor conocimiento mayor sufrimiento
A mayor conocimiento mayor sufrimiento Definicion de garca
Definicion de garca Garca la carolina
Garca la carolina Sergio garca
Sergio garca Miguel garca
Miguel garca Partes del hombro
Partes del hombro Jorge garca
Jorge garca Citogentica
Citogentica Genetica humana
Genetica humana Cromosomas
Cromosomas Tabla de los latidos según las semanas de embarazo
Tabla de los latidos según las semanas de embarazo The function of the chromosomes
The function of the chromosomes Dotacion
Dotacion Centriolos y centrosomas es lo mismo
Centriolos y centrosomas es lo mismo Cromosomas homologos
Cromosomas homologos No disyuncion
No disyuncion Genes holandricos
Genes holandricos Poros nucleares
Poros nucleares