Gene finding Software de prediccin de genes Miquel



















- Slides: 30

Gene finding: Software de predicción de genes Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

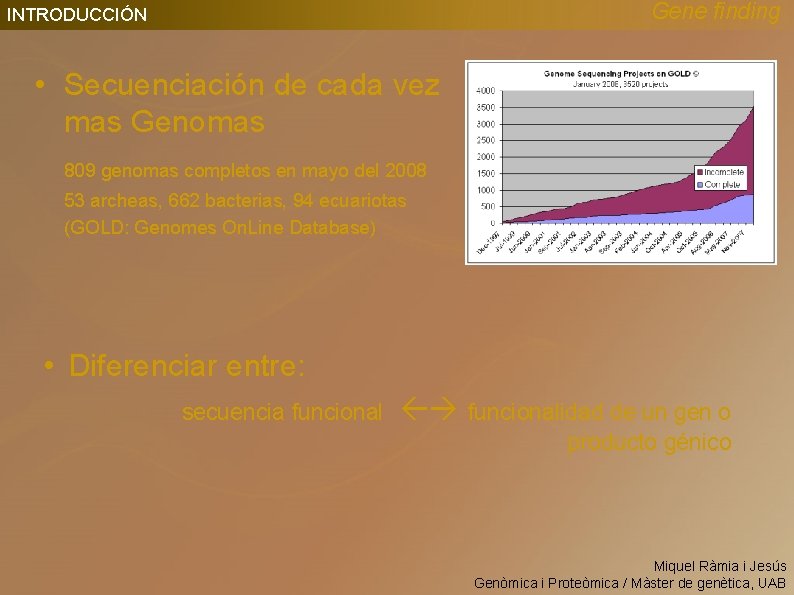
Gene finding INTRODUCCIÓN • Secuenciación de cada vez mas Genomas 809 genomas completos en mayo del 2008 53 archeas, 662 bacterias, 94 ecuariotas (GOLD: Genomes On. Line Database) • Diferenciar entre: secuencia funcionalidad de un gen o producto génico Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding INTRODUCCIÓN PREomics POSTomics Experimental: Computacional: • Cultivos • Hibridaciones • Recombinación y Mapas Genéticos • Grandes bases de datos • Algoritmos de búsqueda • Automatización Regiones Concretas Organismos Concretos Genes Concretos Regiones grandes (Genomas) Muchos organismos Muchos (posibles) genes Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding INTRODUCCIÓN POSTomics Experimental: Computacional: • Cultivos • Hibridaciones • Recombinación y Mapas Genéticos • Grandes bases de datos • Algoritmos de búsqueda • Automatización Regiones Concretas Organismos Concretos Genes Concretos + Regiones grandes (Genomas) Muchos organismos Muchos (posibles) genes Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding ESTRATEGIAS ACTUALES • Métodos de similaridad • Métodos Ab Initio • Genómica Comparativa Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

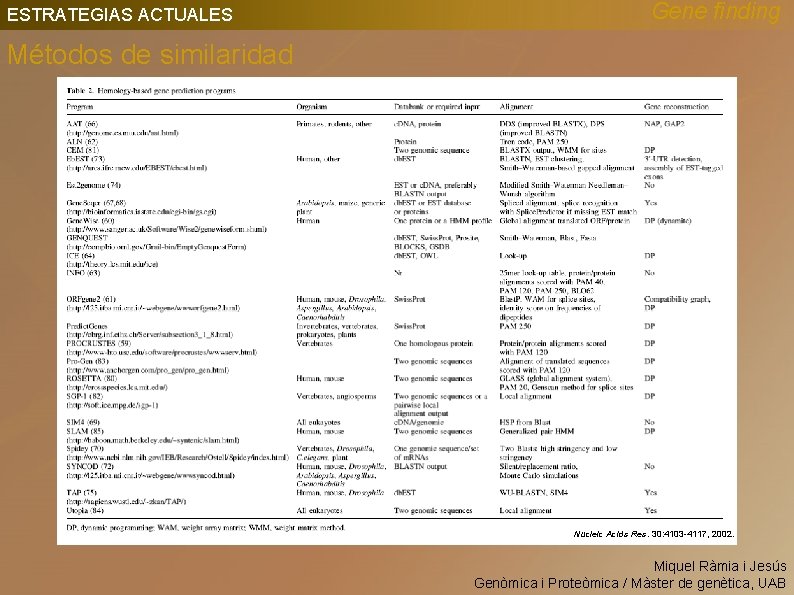
ESTRATEGIAS ACTUALES Gene finding Métodos de similaridad ( Lookup , Evidence based or Extrinsec Methods ) • Comparación de Secuencia y Búsquedas por similaridad (Blast, CLUSTAL) • Alineamiento con Proteínas • Alineamiento con m. RNA/c. DNA • Alineamiento con ESTs • Comparación Intra-genómica • Comparación Inter-genomica (Genómica Comparativa) Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

ESTRATEGIAS ACTUALES Gene finding Métodos de similaridad Nucleic Acids Res. 30: 4103 -4117, 2002. Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding ESTRATEGIAS ACTUALES Métodos de similaridad ✗ • Capacidad limitada para encontrar nuevos genes, se estima que se pueden Encontrar el 50% de genes. “Nada será encontrado si la base de datos no contiene suficientes secuéncias similares” • ESTs solo dan información parcial ya que solo reflejan una parte de un m. RNA • Exones pequeños no son detectados (problema también en ab initio) ✓ • Resultados bastante sólidos al estar basados en evidencia • Comparaciones intra-genómicas aportan información de genes parecidos (familias génicas) • ESTs / c. DNAs dan mucha información sobre divisiones Exon/Intron, y exones usados o no en splicing alternativo Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding ESTRATEGIAS ACTUALES • Métodos de similaridad • Métodos Ab Initio • Genómica Comparativa Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding ESTRATEGIAS ACTUALES Métodos Ab Initio ( Intrinsec, Signal , Template Search ) Métodos totalmente computacionales basados en la busca de patrones o secuencias consenso. El “Gene Prediction” real. • Típos de software: - Predicción de estructura - Detección de Exones/Intrones - Detección de lugares de splicing - Detección de otras senyales • Estratégias: - Secuencias consenso y ORFs - Estadísticos (Codon Bias) y Matrices de puntuación - Arboles de decisión (HMM - Hiden Marcov Models) - Otros… Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

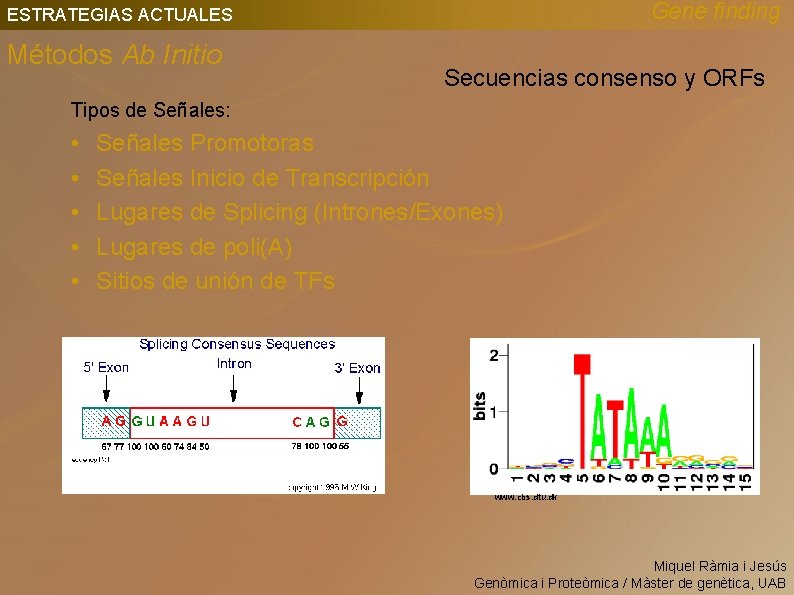
Gene finding ESTRATEGIAS ACTUALES Métodos Ab Initio Secuencias consenso y ORFs Tipos de Señales: • • • Señales Promotoras Señales Inicio de Transcripción Lugares de Splicing (Intrones/Exones) Lugares de poli(A) Sitios de unión de TFs www. cbs. dtu. dk Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding ESTRATEGIAS ACTUALES Métodos Ab Initio ✗ Secuencias consenso y ORFs • Métodos muy útiles en procariotas, pero no tanto en eucariotas con estructuras de genes complejas (ejemplo ORFs) • Incluso en procariotas, podemos encontrar genes solapando con otros genes • Secuencias consenso , por si solas no son 100% fiables en todas las bases ✓ • Aproximación muy rápida, que puede definir posteriores procesos • Paso previo de ORFs necesario Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding ESTRATEGIAS ACTUALES Métodos Ab Initio Estadísticos y Matrices Estadísticos: • Codon Bias • K-meros: Hexameros, etc… • Otros… ✓ • Puntuaciones, índices, valores…de fácil tratamiento computacional • Codon Bias puede ser muy indicativo para separar regiones codificantes del resto ✗ Las matrices y estadísticos provienen de datos previos, se deben de optimizar. Se puede hacer a partir de los datos propios pero requiere iteraciones • Trainig Sets (matrices, codones, secuencias señal…) Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

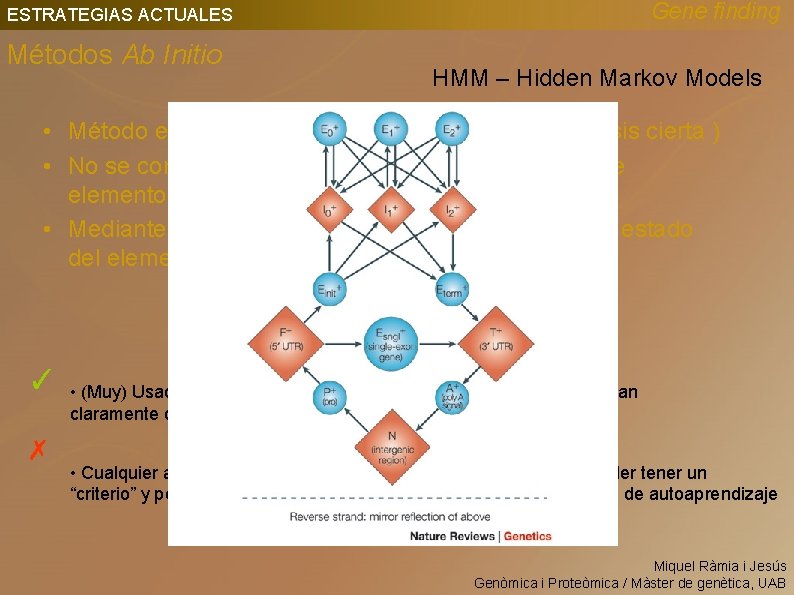
Gene finding ESTRATEGIAS ACTUALES Métodos Ab Initio HMM – Hidden Markov Models • Método estadístico (bayesiano demostrar hipotesis cierta ) • No se conoce previamente el estado de un grupo de elementos observado (Hidden state) • Mediante arboles de decisiones, se intenta inferir el estado del elemento a partir de lo observado ✓ • (Muy) Usado en eucariotas, donde las señales consenso no están tan claramente definidas ✗ • Cualquier algoritmo de este tipo necesita de datos previos para poder tener un “criterio” y poder hacer decisiones. Resultan necesarios mecanismos de autoaprendizaje Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

ESTRATEGIAS ACTUALES Métodos Ab Initio • • Gene finding Otras Aproximaciones Autoaprendizaje (SPV – Suport Vector Machines) Redes Neurales Estructuras secundarias del m. RNA … Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

ESTRATEGIAS ACTUALES Gene finding Métodos Ab Initio • • Autoaprendizaje (SPV – Suport Vector Machines) Redes Neurales Estructuras secundarias del m. RNA … Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding ESTRATEGIAS ACTUALES • Métodos de similaridad • Métodos Ab Initio • Genómica Comparativa Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding ESTRATEGIAS ACTUALES Genómica Comparativa • Algún software especializado (Rosetta para Humano y Ratón, SPG 1 para Vertebrados y Angiospermas) • Aproximación Mixta: (intentar buscar genes representativos del grupo de organismos estudiado) 1 - Usar evidencia experimental (por homologia) · Protein Coding c. DNA · Similaridad con proteinas 2 - Predicción ab Initio 3 - Filtro de los resultados para descartar redundancia, pseudogeneso errores 4 - Ampliar con otras fuentes de evidencia (ESTs, …) Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

ESTRATEGIAS ACTUALES ✗ Gene finding • Software principalmente centrado en regiones que codifican para proteina Por el momento otras secuencias que transcriben son poco tratadas. • Definido a priori que un gen es la región que transcribe entre señal de inicio y final • Poco consenso entre los desarrolladores, falta de standards definidos (GFF – General Feature Format). Programas confusos para el usuario • Detalles a mejorar su detección: - Genes solapantes (por extremo 3’ o genes dentro de intrones) - Genes Policistrónicos (varios genes codificados en un m. RNA) - Frameshifts (perdida de nucleótidos en la secuenciación o bases de datos) - Intrones en secciones no codificantes / Exones no traducidos - Exones pequeños (estructuras secundarias) - Sitios de splicing no canonicos - Procesados alternativos: - Otros inicios de transcripción - Splicing alternativo - Sitios de Poly(A) diferentes - Otros inicios de traducción: AUG alternativos / no-AUG (poca consideración a casos “menores”) Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding ESTRATEGIAS ACTUALES ✓ • Combinación de métodos ofrece resultados más fiables • Puede facilitar pasos previos a la experimentación • Imprescindible para genomas que , quizás, NUNCA serán estudiados experimentalmente Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding TEST DE SOFTWARE Programas a prueba: • Gen. Mark-hmm • GENSCAN • Gene. ID • Genscan se usó en el Proyecto Genoma Humano y en el Genoma del Ratón • Genscan y Gen. Mark-hmm tienen las mejores valoraciones en pruebas de este tipo de software • Los tres tienen interface web Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

TEST DE SOFTWARE Gene finding • Gen. Mark-hmm - Desde 1993 - Instituto tecnológico de Georgia, patrocinado por IBM - Varios programas: Procariotas, Eucariotas, Heuristicos, EST y c. DNA … Gen. Mark-hmm : Usa Hidden Markov Models y selftraining Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding TEST DE SOFTWARE • GENSCAN - Desde 1997 - Dep. Matemáticas de la Universidad De Stanford - (web) Vertebrados, arabidopsis, maíz. Usa Hidden Markov Models y se basa en información de doble cadena Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding TEST DE SOFTWARE • GENEID - Desde 2000 (paper en Drosophila) - Universitat Pompeu Fabra / CRG - Datos para varios organismos, van publicando nuevos sets periodicamente - Permite añadir datos externos (GFF) - Opcion de output en formato GFF - Web muy explicativa Usa 3 passos: 1. Deteccion de señales: Splicing, start-stop codons, matrices de puntuación 2. Usa los datos anteriores en un modelo HMM 3. Integrar todo en una predicción de la estructura Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

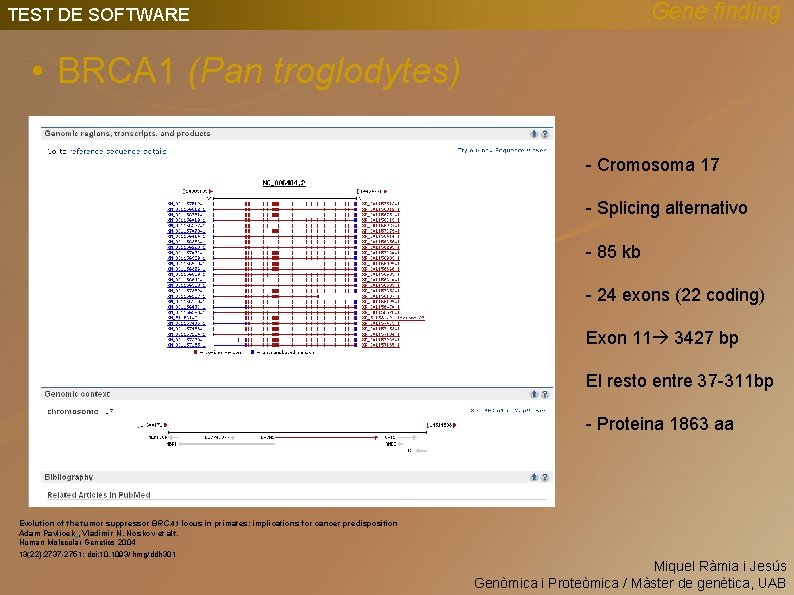
Gene finding TEST DE SOFTWARE • BRCA 1 (Pan troglodytes) - Cromosoma 17 - Splicing alternativo - 85 kb - 24 exons (22 coding) Exon 11 3427 bp El resto entre 37 -311 bp - Proteina 1863 aa Evolution of the tumor suppressor BRCA 1 locus in primates: implications for cancer predisposition Adam Pavlicek , Vladimir N. Noskov et alt. Human Molecular Genetics 2004 13(22): 2737 -2751; doi: 10. 1093/ hmg/ddh 301 Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

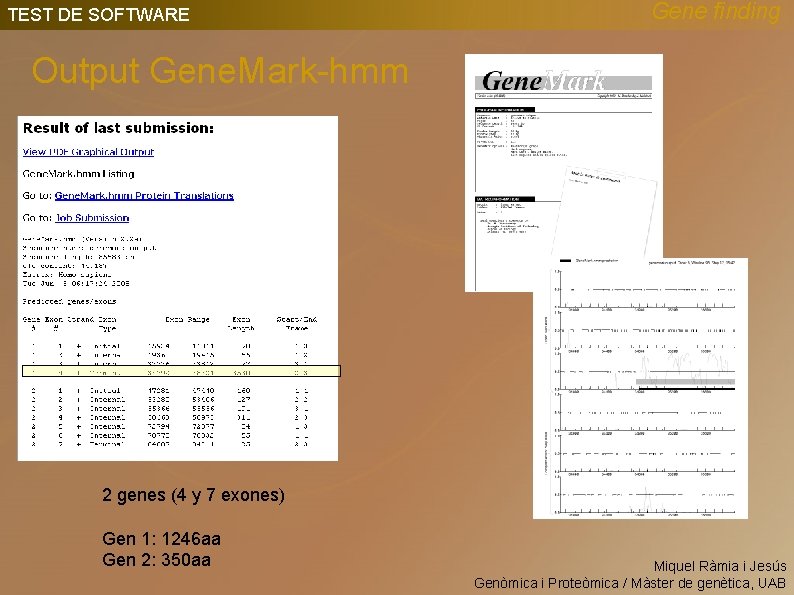
TEST DE SOFTWARE Gene finding Output Gene. Mark-hmm 2 genes (4 y 7 exones) Gen 1: 1246 aa Gen 2: 350 aa Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

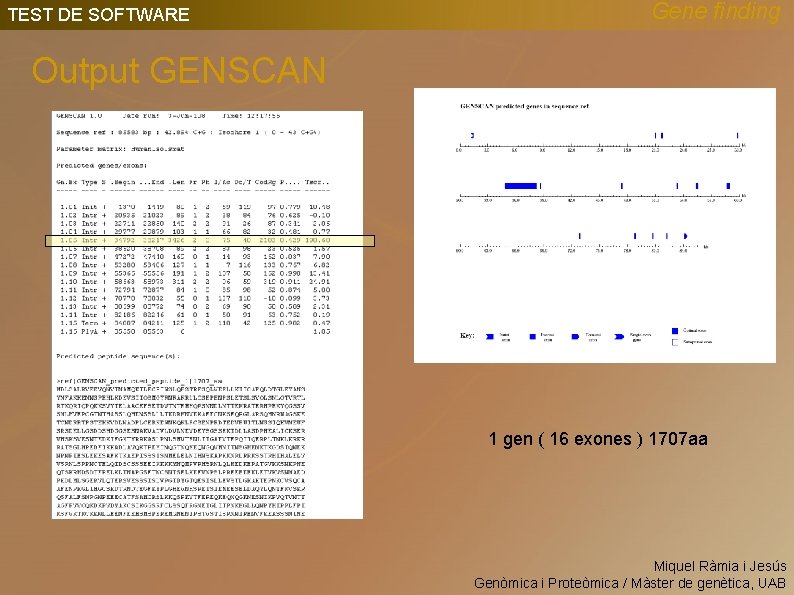
TEST DE SOFTWARE Gene finding Output GENSCAN 1 gen ( 16 exones ) 1707 aa 2 genes (4 y 7 exones) Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

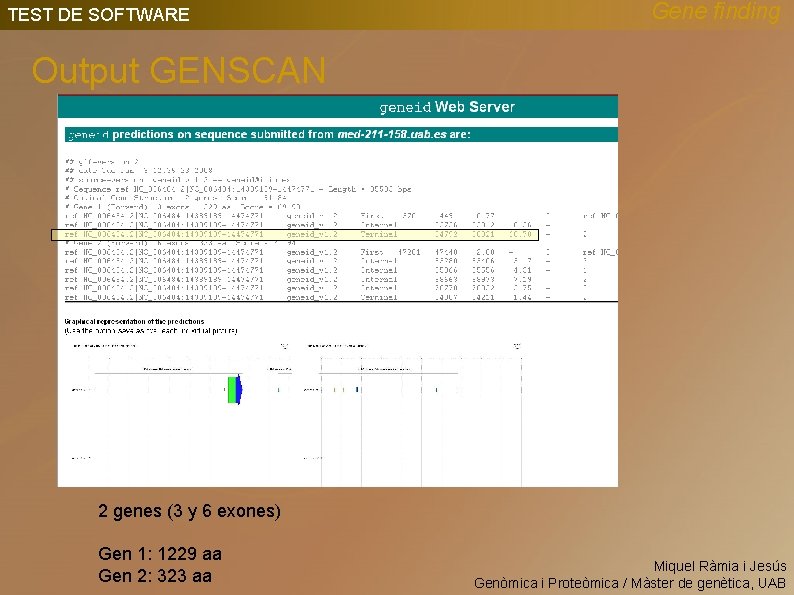
TEST DE SOFTWARE Gene finding Output GENSCAN 2 genes (3 y 6 exones) Gen 1: 1229 aa Gen 2: 323 aa Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding INFORMACIÓN COMPLEMENTARIA http: //www. genefinding. org/ http: //www. nslij-genetics. org/gene/programs. html Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB

Gene finding REFERENCIAS Evaluation of gene prediction software using genomic dataset: application to Arabidopsis thaliana sequences. Pavy N, Rombauts S et alt. Bioinformatics Vol 15 no 11 1999. 887 -899 Evaluation of Gene-Finding Programs on Mammalian Sequences Sanja Rogic, Alan K. Mackworth and Francis B. F. Ouellette. Genome Res. 2001 11: 817 -832 Current methods of gene prediction, their strengths and weaknesses Catherine Mathé, Marie-France Sagot, Thomas Schiex and Pierre Rouzé. Nucleic Acids Res. 30: 4103 -4117, 2002. In search of the small ones: improved prediction of short exons in vertebrates, plants, fungi and protists. Yvan Saeys, Pierre Rouze and Yves Van de Peer. Vol. 23 no. 4 2007, pages 414– 420 doi: 10. 1093/bioinformatics/btl 639 Miquel Ràmia i Jesús Genòmica i Proteòmica / Màster de genètica, UAB