FEG FEG 1PM 3 QMSerAlaMM 100 stepsMDFEG Sample












- Slides: 17



FEG最適化の利用 FEGのテスト計算 1 (PM 3) QM領域は、Serのみで、AlaはMM領域 100 stepsのMDによるFEG(水分子なし) Sample NEBルーチンには、IMIN=1, INEB=1でないと入らない。 INEB=0がdefault &cntrl NTORP=2, NCHAIN=2, MTSTAT(1)=3, MTSTAT(2)=3, NHCPAR=0, NTT=5, NTF=1, CUT=8. 0, SCEE=1. 2, IMIN=1, NTMIN=4, NSTLIM=100, NRUN=1, NSNB=10, INIT=3, DT=0. 001, MAXCYC=100, NTB=0, NTF=1, NTC=1, NTX=1, INIT=3, TEMPI=300. 0, TIMLIM=99999. , NTPR=1, NTWX=1, NTWV=000, NTWE=1, NTWXM=0, NTWVM=0, NTWEM=0, IBELLY=0, IFQT=1, NQT=13, MODCHG=1, KMAX=0, INEB=1, nbeads=1, &end 1 2 3 4 5 6 7 8 9 10 11 12 13 14 0. 0000 nbeads=1もしくは、2の場合、FEG計算を実行する。 nbeads=3以上で、2個の構造を結ぶNEB計算を実行できる。 IMIN=0: standard MD IMIN=1, INEB=0: standard optimization IMIN=1, INEB=1, nbeads=1: FEG optimization IMIN=1, INEB=1, nbeads>=3: FEG-NEB optimization INEB=2, 3: 全ページ参照 NSTLIM回のMD平均力を用いたMAXCYC回の最適化を実行する。 最適化が、MAXCYCステップに到達するか、もしくは構造が収束 した場合に、実行が終了する。 keywordファイル GEO-OK GRAD ANALYT PRECISE PM 3 CHARGE=1 MMOK


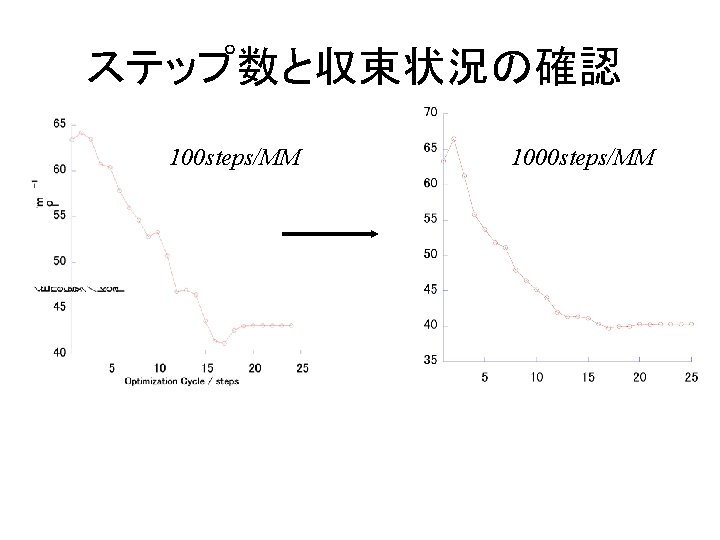
ステップ数と収束状況の確認 100 steps/MM 1000 steps/MM

非結合性の相互作用を含め、かつQM領域 の計算にはGaussian 03を用いて確認 FEGのテスト計算 2 (RHF/STO-3 G) QM領域は、Serのみで、Ala+52 waterはMM領域 100 stepsのMDによるFEG Sample 2 &cntrl gaussianのヘッダー NTORP=2, NCHAIN=2, MTSTAT(1)=3, MTSTAT(2)=3, %nosave NHCPAR=0, NTT=5, NTF=1, CUT=8. 0, SCEE=1. 2, IMIN=1, NTMIN=4, NSTLIM=100, NRUN=1, NSNB=10, INIT=3, %nproc=2 DT=0. 001, MAXCYC=100, NTB=1, %rwf=elec. rwf NTF=1, NTC=1, NTX=1, INIT=3, TEMPI=10. 0, %mem=200 MB TIMLIM=99999. , NTPR=1, NTWX=1, NTWV=000, NTWE=1, NTWXM=0, NTWVM=0, NTWEM=0, IBELLY=0, IFQT=1, NQT=13, MODCHG=1, #p rhf/sto-3 g charge density=current nosymm prop=(read, field) force iop(6/13=1) KMAX=0, INEB=1, nbeads=1, &end 1 2 3 4 5 6 7 8 9 10 11 12 13 14 QM/MM 0. 0000 11


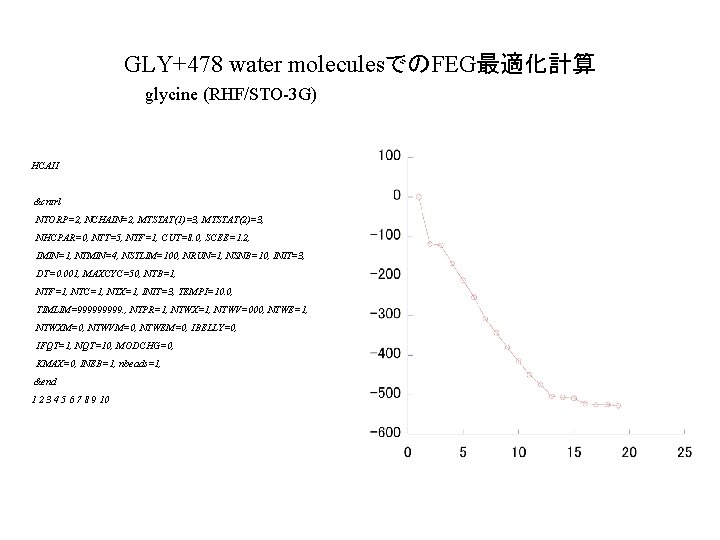
GLY+478 water moleculesでのFEG最適化計算 glycine (RHF/STO-3 G) HCAII &cntrl NTORP=2, NCHAIN=2, MTSTAT(1)=3, MTSTAT(2)=3, NHCPAR=0, NTT=5, NTF=1, CUT=8. 0, SCEE=1. 2, IMIN=1, NTMIN=4, NSTLIM=100, NRUN=1, NSNB=10, INIT=3, DT=0. 001, MAXCYC=50, NTB=1, NTF=1, NTC=1, NTX=1, INIT=3, TEMPI=10. 0, TIMLIM=99999. , NTPR=1, NTWX=1, NTWV=000, NTWE=1, NTWXM=0, NTWVM=0, NTWEM=0, IBELLY=0, IFQT=1, NQT=10, MODCHG=0, KMAX=0, INEB=1, nbeads=1, &end 1 2 3 4 5 6 7 8 9 10



nebの初期ビーズと環境生成 (imin=1, ineb=2, nbeads=4, maxcyc=20) 1 2 3 4


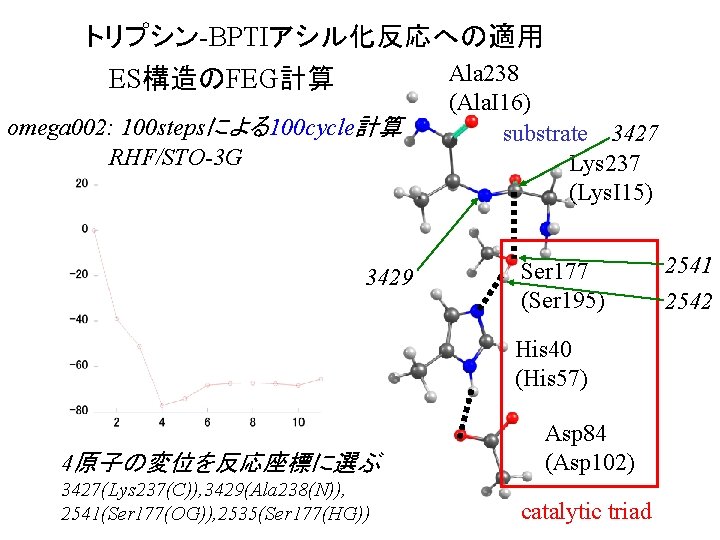
トリプシン-BPTIアシル化反応への適用 Ala 238 ES構造のFEG計算 omega 002: 100 stepsによる100 cycle計算 RHF/STO-3 G 3429 (Ala. I 16) substrate 3427 Lys 237 (Lys. I 15) Ser 177 (Ser 195) His 40 (His 57) 4原子の変位を反応座標に選ぶ Asp 84 (Asp 102) 3427(Lys 237(C)), 3429(Ala 238(N)), 2541(Ser 177(OG)), 2535(Ser 177(HG)) catalytic triad 2541 2542




 100 100 100 100 100
100 100 100 100 100 Feg t
Feg t Friedrich ebert gymnasium sandhausen
Friedrich ebert gymnasium sandhausen Moodle feg
Moodle feg 100 + 100 200
100 + 100 200 50 nin 40 ı kaçtır
50 nin 40 ı kaçtır 100 + 100 + 200
100 + 100 + 200 1453-1337
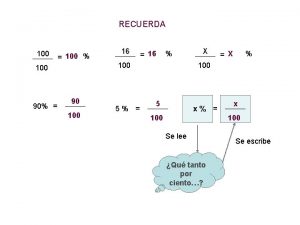
1453-1337 C/100=f-32/180=k-273/100
C/100=f-32/180=k-273/100 200+200+100+100
200+200+100+100 100+100=200
100+100=200 What's 100 + 100
What's 100 + 100 Box plots gcse
Box plots gcse 100 iops/gb and 100,000 iops per volume oci
100 iops/gb and 100,000 iops per volume oci Numeros romanos del 1 al 500
Numeros romanos del 1 al 500 300 + 200 + 200
300 + 200 + 200 Malloc lab 100/100
Malloc lab 100/100 A sample poll of 100 voters chosen at random
A sample poll of 100 voters chosen at random