ENZIMI DI RESTRIZIONE 1 Enzimi che interagiscono con
















- Slides: 29

ENZIMI DI RESTRIZIONE 1

Enzimi che interagiscono con il DNA 1. DNA polimerasi 2. DNA ligasi 3. Chinasi e Fosfatasi alcaline 4. Nucleasi 5. Topoisomerasi 6. Metilasi

Nucleasi • Esonucleasi: – Rimuovono un nucleotide alla volta dalla molecola di DNA iniziando da una delle estremità libere e proseguendo verso l’estremità opposta • Endonucleasi: – Rompono i legami fosfodiesterici all’interno di molecole di DNA lineari o circolari – Comprendono gli enzimi di restrizione

Classificazione delle endonucleasi • Tipo 1: proteine con diverse subunità che funzionano sottoforma di complesso multimerico; necessitano di energia per il taglio. Provocano tagli casuali sul DNA (riconoscono sequenze di 3/4 bp ma possono tagliare anche a 1000 basi di distanza rispetto alle sequenze riconosciute) • Tipo 3: composte da due domini (endonucleasi + metilasi). Riconoscono sequenze di 5/6 bp ma tagliano fino a 25 basi di distanza. Necessitano di energia per il taglio. • Tipo 4: endonucleasi che riconosco DNA modificato (metilato, idrossi-metilato, glucosil-idrossi-metilato) Le endonucleasi di tipo 1, 3 e 4 NON sono utili nelle tecniche molecolari di laboratorio (clonaggi, discriminazione allelica, ecc. )

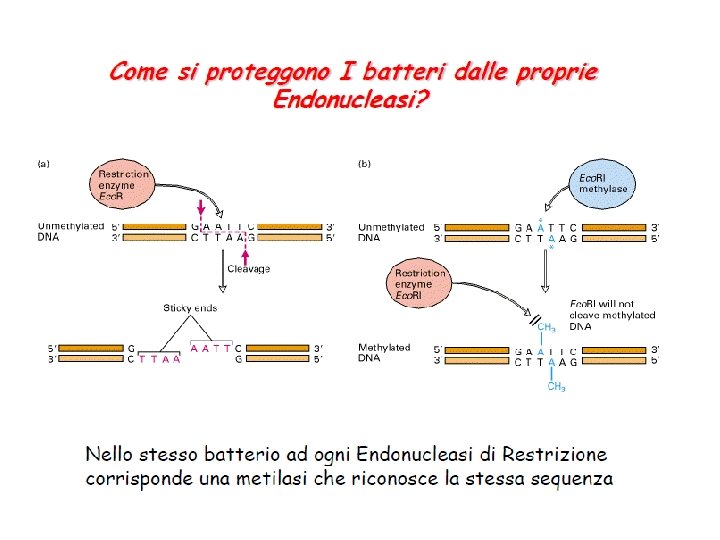
• Tipo 2: chiamati enzimi di restrizione (ER) tagliano il DNA all’interno di specifiche regioni di riconoscimento producendo al 5’ estremità fosforilate e 3’ idrossilate. Le sequenze riconosciute sono composte da 4 a 10 bp e palindromiche (sequenze di basi a simmetria binaria: la sequenza è sempre la stessa quando i due filamenti di DNA sono letti dal 5’ al 3’). Indispensabili in molte tecniche di laboratorio! - caratterizzati 3500 diversi ER di cui oltre 200 sono commerciali - identificati in ceppi batterici resistenti ai batteriofagi (possibilità di distruggere il DNA del fago ma non il proprio grazie alla metilazione delle proprie sequenze palindromiche!)


- ogni specie batterica produce un diverso ER il cui nome è un acronimo di 3 lettere (I lettera = genere microrganismo, II e III = specie; altri numeri o lettere = ceppo batt. ) Es. l’enzima Hind. II è stato isolato da Haemophilus influenzae ceppo d, II enzima isolato

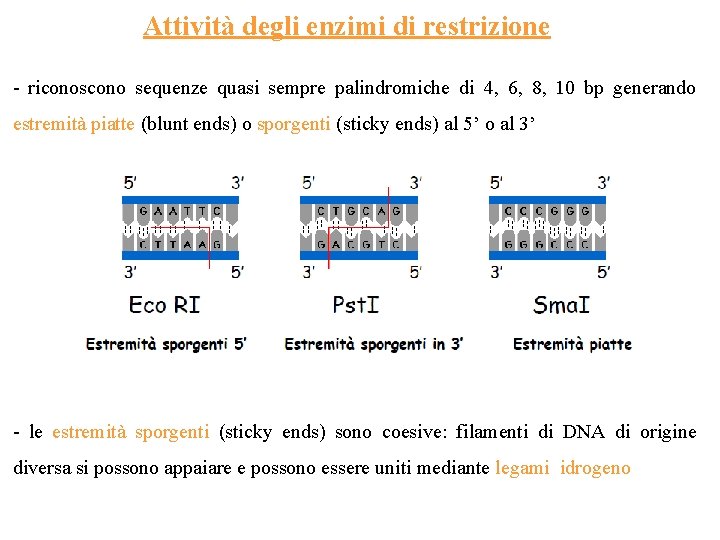
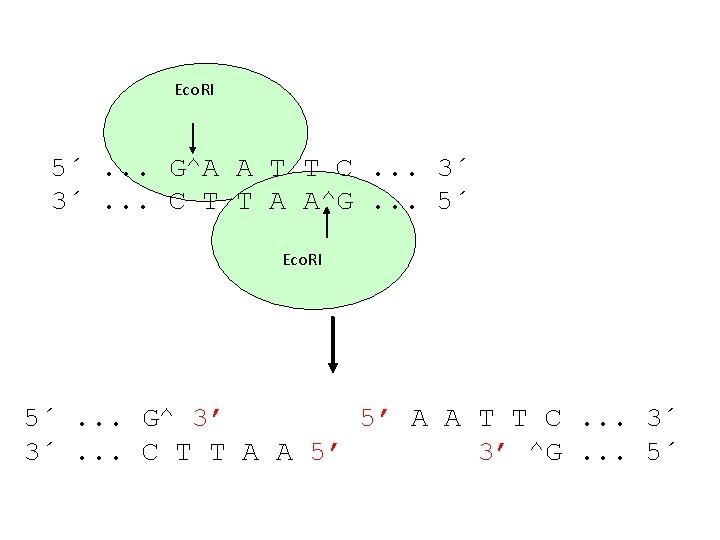
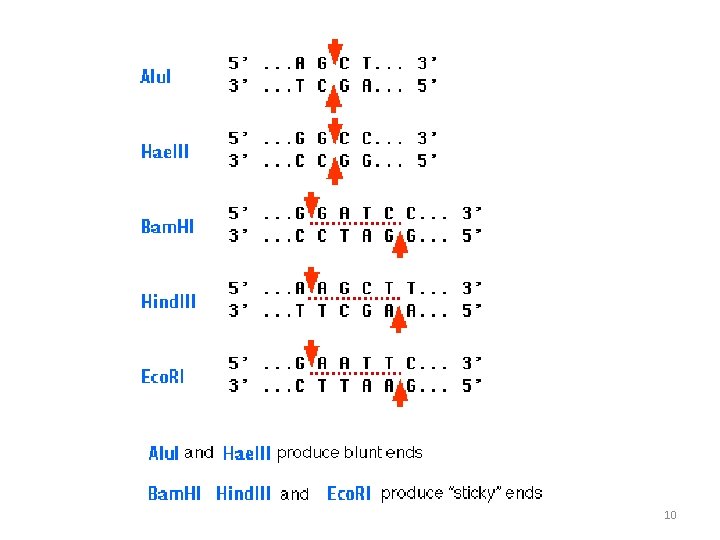
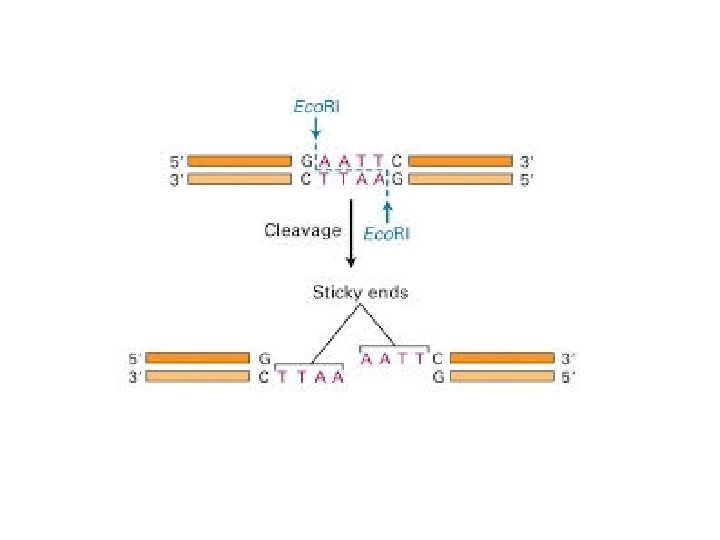
Attività degli enzimi di restrizione - riconoscono sequenze quasi sempre palindromiche di 4, 6, 8, 10 bp generando estremità piatte (blunt ends) o sporgenti (sticky ends) al 5’ o al 3’ - le estremità sporgenti (sticky ends) sono coesive: filamenti di DNA di origine diversa si possono appaiare e possono essere uniti mediante legami idrogeno


10


Esempi di sequenze riconosciute dagli ER 4 -basi: Alu. I 5´. . . AG^CT. . . 3´ blunt ends Msp. I 5´. . . C^CGG. . . 3´ 5’ overhang (2 bp) 6 -basi Pvu. II 5´. . . CAG^CTG. . . 3´ blunt ends Kpn. I 5´. . . GGTAC^C. . . 3´ 3’ overhang (4 bp) 5´. . . GC^GGCCGC. . . 3´ 5’ overhang (4 bp) 8 -basi Not. I Sequenze particolari Mwo. I 5´. . . GCNNNNN^NNGC. . . 3´ 3´. . . CGNN^NNNNNCG. . . 5´ 3’ overhang (3 bp)

Quali sono le probabilità che un ER di restrizione tagli una sequenza prescelta? A) Se si assume che il genoma umano sia formato da sequenze randomizzate delle quattro basi (A, T, G, C) e che ognuna delle quattro basi è presente con la stessa frequenza, si può stimare (calcolo ipotetico) che un ER può tagliare il DNA: - ogni 256 bp (ER che riconosce sequenze di 4 bp -> 44 = 256) - ogni 4096 bp (ER che riconosce sequenze di 6 bp -> 46 = 4096) - ogni 65536 bp (ER che riconosce sequenze di 8 bp -> 48 = 65536) Però le sequenze non sempre sono distribuite random B) Se la sequenza è conosciuta posso cercare la sua distribuzione nel genoma con www. rebase. neb. com C) La regione in cui è inserita la sequenza ha una certa importanza! (alcuni siti sono preferiti dagli ER rispetto ad altri)

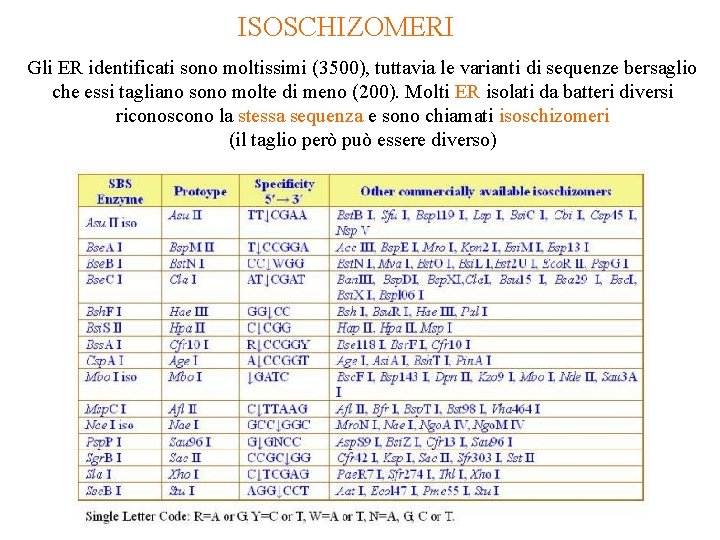
ISOSCHIZOMERI Gli ER identificati sono moltissimi (3500), tuttavia le varianti di sequenze bersaglio che essi tagliano sono molte di meno (200). Molti ER isolati da batteri diversi riconoscono la stessa sequenza e sono chiamati isoschizomeri (il taglio però può essere diverso)

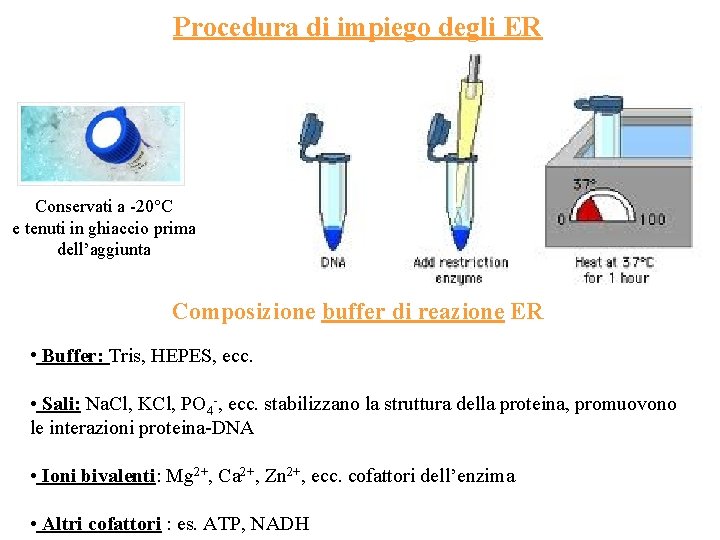
Procedura di impiego degli ER Conservati a -20°C e tenuti in ghiaccio prima dell’aggiunta Composizione buffer di reazione ER • Buffer: Tris, HEPES, ecc. • Sali: Na. Cl, KCl, PO 4 -, ecc. stabilizzano la struttura della proteina, promuovono le interazioni proteina-DNA • Ioni bivalenti: Mg 2+, Ca 2+, Zn 2+, ecc. cofattori dell’enzima • Altri cofattori : es. ATP, NADH

Composizione buffer di conservazione ER • Buffer: Tris, HEPES, ecc. • Sali: Na. Cl, KCl, PO 4 -, ecc. stabilizzano la struttura della proteina, promuovono le interazioni proteina-DNA • Glicerolo: per la conservazione dell’enzima, stabilizza la sua struttura • EDTA: interagisce con gli ioni bivalenti cofattori importanti per l’attività delle proteasi (conservazione dell’enzima) • Beta mercaptoetanolo o ditiotreitolo (DTT): agenti riducenti che prevengono la formazione di legami disolfuro non idonei • Proteine per la conservazione: Bovine serum albumin (BSA) Per definizione 1 unità di ER rappresenta la quantità di enzima richiesta per digerire completamente 1 microgrammo di DNA substrato in un’ora

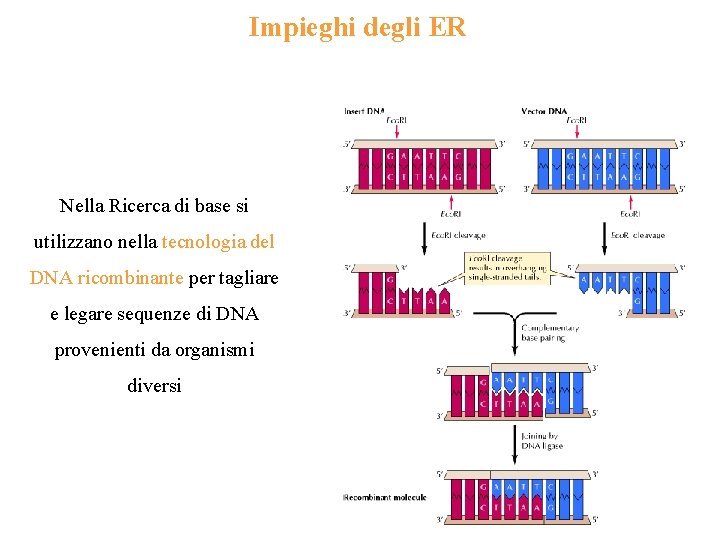
Impieghi degli ER Nella Ricerca di base si utilizzano nella tecnologia del DNA ricombinante per tagliare e legare sequenze di DNA provenienti da organismi diversi

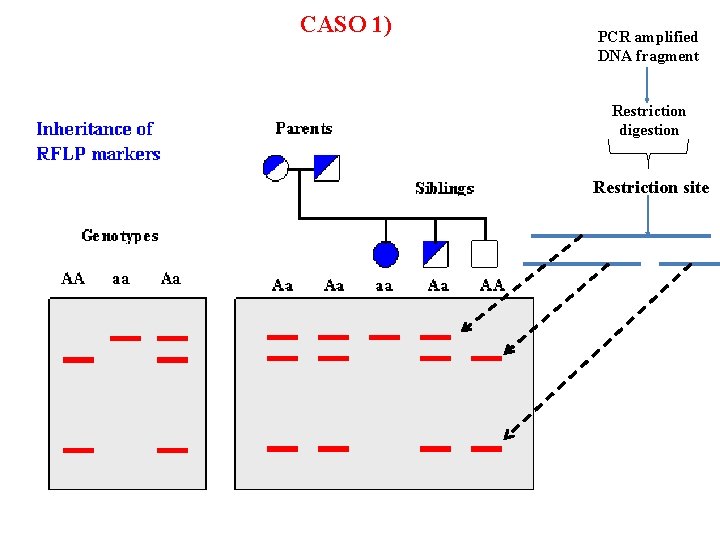
In Diagnostica molecolare gli ER si utilizzano per lo studio di mutazioni puntiformi o di polimorfismi a singolo nucleotide (SNP, variazioni a carico di un unico nucleotide che si riscontrano in soggetti di una stessa specie) RFLP: “Polimorfismi di lunghezza dei frammenti di restrizione” - Più del 50% del genoma umano presenta sequenze che sono riconosciute dagli enzimi di restrizione a disposizione - E’ possibile individuare mutazioni o SNPs nel momento in cui la base interessata cade all’interno di una sequenza di riconoscimento per un ER - Se un allele contiene un sito di riconoscimento per un ER e l’altro no, la digestione enzimatica produrrà frammenti di dimensione differente

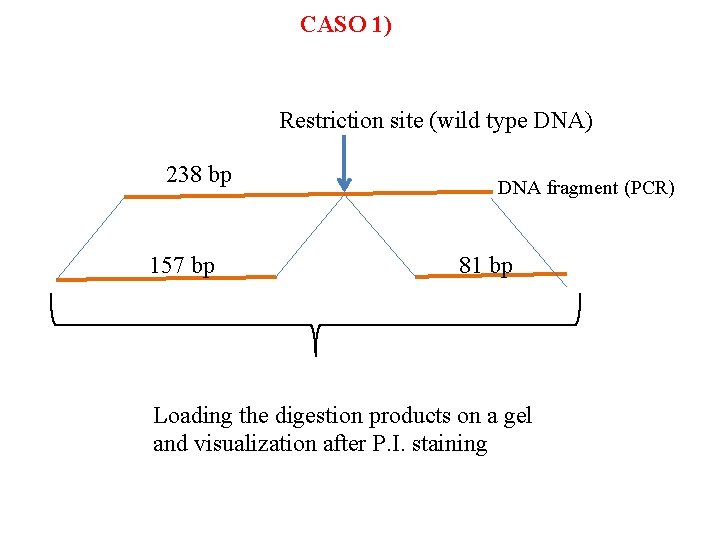
- in seguito alla mutazione o alla comparsa di uno SNP può non esistere più la sequenza per l’ER (presente nel DNA wild type) oppure può crearsi una sequenza per l’ER (non presente nel DNA wild type) Caso 1 -> la mutazione (responsabile della patologia) fa scomparire un sito di restrizione presente nella sequenza wild type a) si amplifica con PCR la regione di DNA da studiare per poter avere abbastanza templato su cui lavorare (campione controllo wild type e campione sospetto +) b) si digeriscono gli amplificati con l’ER specifico per il riconoscimento della sequenza al cui interno cade la mutazione c) dopo elettroforesi si osserveranno due frammenti nel controllo (taglio avvenuto) mentre un solo frammento nel campione omozigote per la mutazione. 3 frammenti si osservano nel campione di un soggetto portatore (eterozigote)

CASO 1) Restriction site (wild type DNA) 238 bp 157 bp DNA fragment (PCR) 81 bp Loading the digestion products on a gel and visualization after P. I. staining

CASO 1) PCR amplified DNA fragment Restriction digestion Restriction site

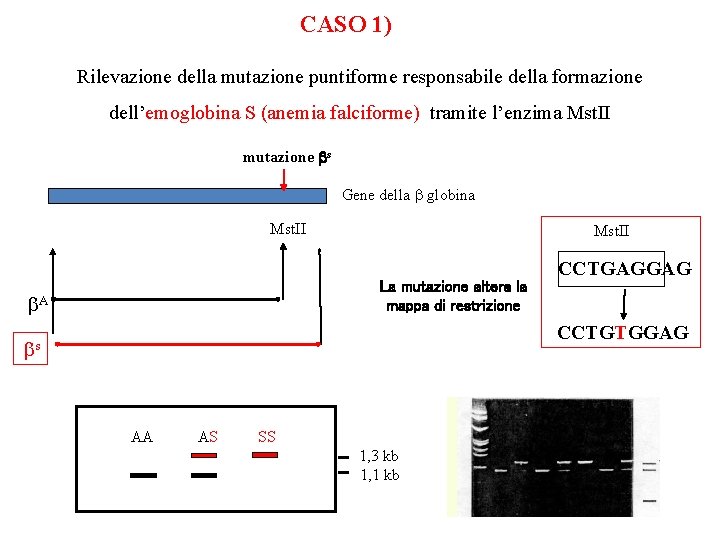
CASO 1) Rilevazione della mutazione puntiforme responsabile della formazione dell’emoglobina S (anemia falciforme) tramite l’enzima Mst. II mutazione s Gene della globina Mst. II La mutazione altera la mappa di restrizione A CCTGAGGAG CCTGTGGAG s AA AS SS 1, 3 kb 1, 1 kb

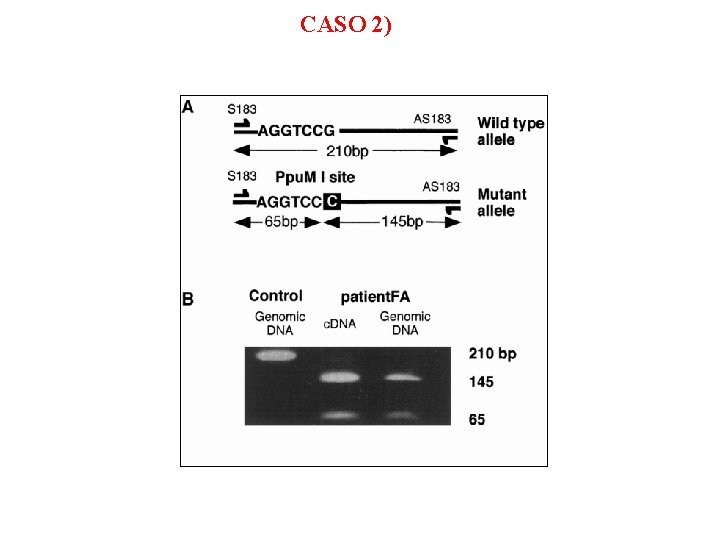
Caso 2 -> la mutazione (responsabile della patologia) fa comparire un sito di restrizione prima non presente nella sequenza wild type a) si amplifica con PCR la regione di DNA da studiare per poter avere abbastanza templato su cui lavorare (campione controllo wild type e campione sospetto +) b) si digeriscono gli amplificati con l’ER specifico per il riconoscimento della sequenza al cui interno cade la mutazione c) dopo elettroforesi si osserveranno un frammento nel controllo (nessun taglio avvenuto) mentre due frammenti nel campione omozigote per la mutazione. 3 frammenti si osservano nel campione di un soggetto portatore (eterozigote)

CASO 2)

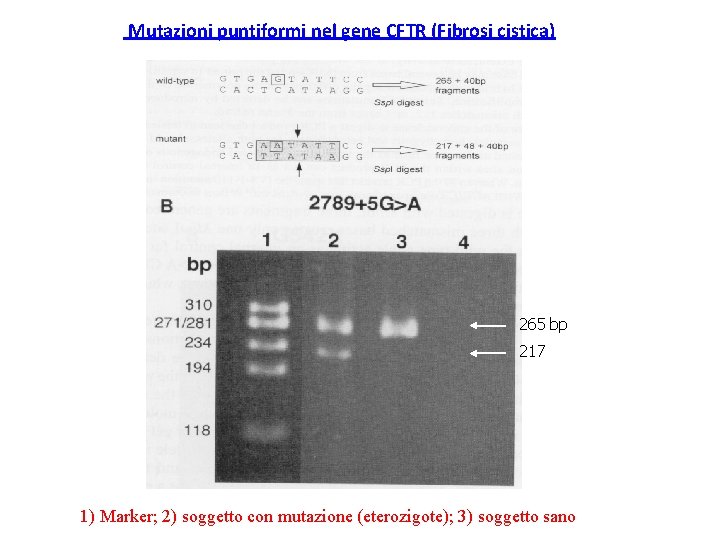
Mutazioni puntiformi nel gene CFTR (Fibrosi cistica) 265 bp 217 1) Marker; 2) soggetto con mutazione (eterozigote); 3) soggetto sano


ER e “Star activity” Quando le condizioni di reazione non sono ottimali, alcuni ER acquisiscono la capacità di riconoscere e tagliare sequenze NON CANONICHE simili ma non identiche alle originali. Questa mancanza di specificità enzimatica prende il nome di STAR ACTIVITY Eco. R 1→GAATTC Eco. R 1 con star activity →NAATTN (N=qualsiasi base)

Condizioni che contribuiscono alla Star activity Alta concentrazione di glicerolo (> 10% v/v) Suggerimenti per evitare Star activity Gli ER sono conservati in una soluzione con il 50% di glicerolo, per questo motivo la quantità di ER non deve superare il 10% del vol. totale di reazione. Un volume di 50 µl di reazione può ridurre il rischio di superare qs rapporto causa evaporazioni. Si impiega la minore quantità possibile di Alta concentrazione di enzima/µg di DNA enzima per la reazione. Ciò evita digestioni (es. rapporto enzima/DNA =100 unità/1µg troppo spinte e riduce la quantità di anzichè la canonica 10 unità/1µg ) glicerolo nel volume di reazione Buffer non ottimale Le reazione vanno condotte nel tampone consigliato. Buffer che differiscono per forza ionica e p. H contribuiscono alla star activity

Condizioni che contribuiscono alla Star activity Presenza di solventi organici [DMSO, etanolo(4), ethylene glycol, dimethylacetamide, dimethylformamide, sulphalane (5)] Sostituzione del Mg 2+con altri cationi bivalenti (Mn 2+, Cu 2+, Co 2+, Zn 2+) Suggerimenti per evitare Star activity Questi reagenti possono esser presenti dopo eluizione del DNA nella procedura di estrazione. Purificare su colonnine apposite per togliere i contaminanti Impiegare il Mg 2+ come ione bivalente. Altri cationi bivalenti non si inseriscono correttamente nel sito attivo dell’enzima, ciò interferisce con l’attività di riconoscimento dell’enzima