ENTREZ Genomes Map Viewer I Map Viewer II
















































![Query: gattacaccccgattaca (29 letters) Példa [2 mins] Database: All Gen. Bank+EMBL+DDBJ+PDB sequences (but no Query: gattacaccccgattaca (29 letters) Példa [2 mins] Database: All Gen. Bank+EMBL+DDBJ+PDB sequences (but no](https://slidetodoc.com/presentation_image_h2/d77d6c734a43654e91143bd09433b4b8/image-76.jpg)
![Query: Human atoh enhancer, 179 letters [1. 5 min] Példa Result: 57 blast hits Query: Human atoh enhancer, 179 letters [1. 5 min] Példa Result: 57 blast hits](https://slidetodoc.com/presentation_image_h2/d77d6c734a43654e91143bd09433b4b8/image-77.jpg)
- Slides: 77

ENTREZ - Genomes

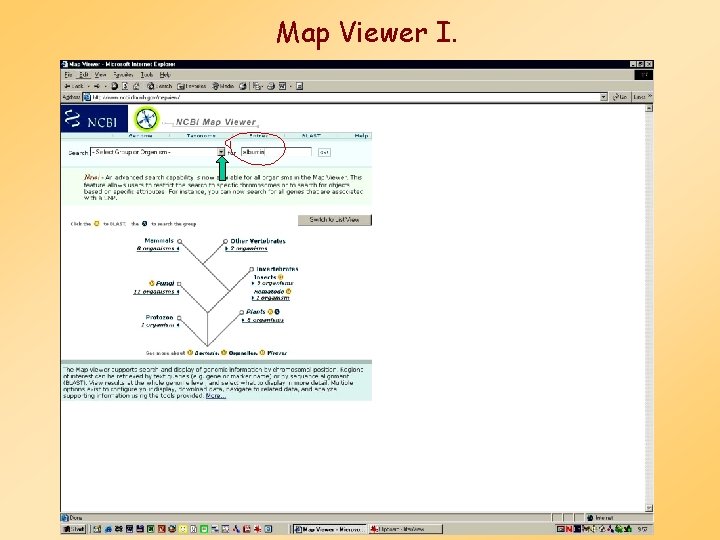
Map Viewer I.

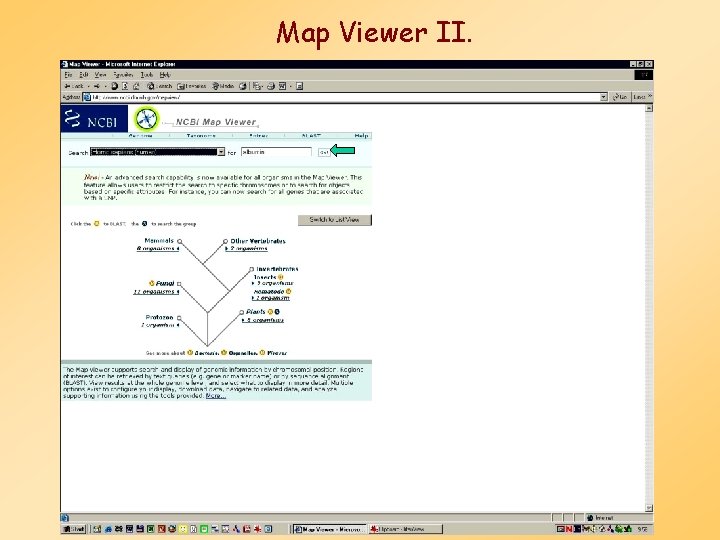
Map Viewer II.

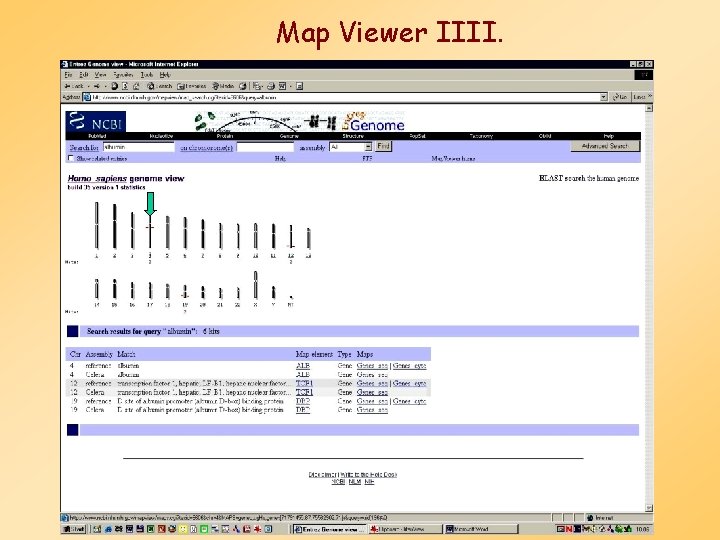
Map Viewer IIII.

Map Viewer IV.

European Bioinformatics Institute (EBI)

European Bioinformatics Institute (EBI)

Readseq: szekvencia formátum konvertáló

Szekvencia formátumok I. FASTA GCG >nameless_1 457 bp GGCGAAGATTCGGCCAGGCAAAGAAGAGCGCGACGAATGGGAGCATGTAGCCA TGGCTGTATTCCTCCGTACCCCATGCCCCAACCATGCGAGTCAAACCTTCGTGA AAGATCACCACGAGCAGCCCGAAGACGATGGCCATCCACACGACGTGAA CCGCGCCCTCCATGCGGTCCGCGGTTTTTCAATGATCGAAAGGGAATCGGCAAC TTGAGTGGAGTCATATCAGGAGTCCCTTTGGGAGATGCTTCAGAGAGCAA GGTTCGTTGCCTCGGACCTGCATCACCCAACCATACAGATGCTCGGTTCGCGAC GGCCTGCGTTGATTGCGCTGAGGATACCCGGTTCCAGTCCGTGCGACGACCATT AATAAGGCGCTCCACAGTTCCCGCGGGACACTAGCCAACCGGGCAGTGTCCAC TGGGCAGCGGGCAGGGTCTCCCCCGGGA nameless_1 1 GGCGAAGATT 51 CCATGGCTGT 101 TTCGTGAAAG 151 CGTGAA 201 AGGGAATCGG 251 AGATGCTTCA 301 ATACAGATGC 351 CGGTTCCAGT 401 GGGACACTAG 451 CCCGGGA Length: 457 Nov 15, 2004 10: 24 CGGCCAGGCA AAGAAGAGCG CGACGAATGG ATTCCTCCGT ACCCCATGCC CCAACCATGC ATCACCACGA GCAGCCCGAA GACGATGGCC CCGCGCCCTC CATGCGGTCC GCGGTTTTTC CAACTTGAGT GGAGTC ATATCAGGAG GAGAGCAAGG TTCGTTGCCT CGGACCTGCA TCGGTTCGCG ACGGCCTGCG TTGATTGCGC CCGTGCGACG ACCATTAATA AGGCGCTCCA CCAACCGGGC AGTGTCCACT GGGCAGCGGG Check: 7178 GAGCATGTAG GAGTCAAACC ATCCACACGA AATGATCGAA TCCCTTTGGG TCACCCAACC TGAGGATACC CAGTTCCCGC CAGGGTCTCC . .

Szekvencia formátumok II. Gen. Bank EMBL

Readseq: szekvencia formátum konvertáló, fehérje

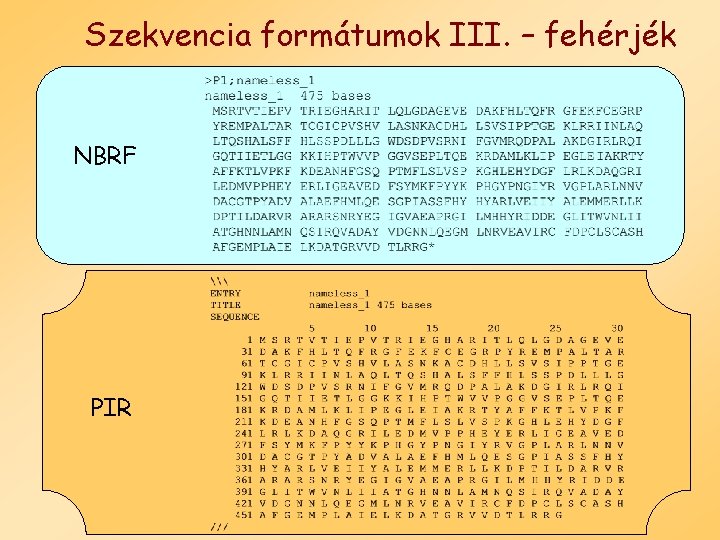
Szekvencia formátumok III. – fehérjék NBRF PIR

KERESÉS AZ ADATBNKOKBAN: HASONLÓSÁG Elsődleges DNS vagy fehérje szekvencia összehasonlítása más elsődleges szekvenciákhoz abban a reményben, hogy annak a funkciója ismert a kísérletek szükségessége analogikus gondolkodás ha valamilyen fehérje hasonlít valami ismert funkiójú fehérjéhez, akkor a funkció is hasonló kérdés: mi hordozza a funkciót? fehérje, vagy fehérje rész, hány funkciója van egy fehérjének? globalitás - lokalitás

Szekvencia illesztés

Illesztés - héttér “For many protein sequences, evolutionary history can be traced back 1 -2 billion years” -William Pearson • When we align sequences, we assume that they share a common ancestor – They are then homologous • Protein fold is much more conserved than protein sequence • DNA sequences tend to be less informative than protein sequences

Szekvenciák illesztése • Nagyon sok illesztés, alignment lehetséges. • Két szekvenciát mindig lehet illeszteni Kérdés: jó-e, valós hasonlóságot mutat-e az illesztése. Ehhez • az illesztések “jóságát” pontozni kell • Gyakran több illeszkedés is jó, ugyanolyan ponttal

Szekvenciák illesztése…. actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact Szekvencia 1 Szekvencia 2 actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact

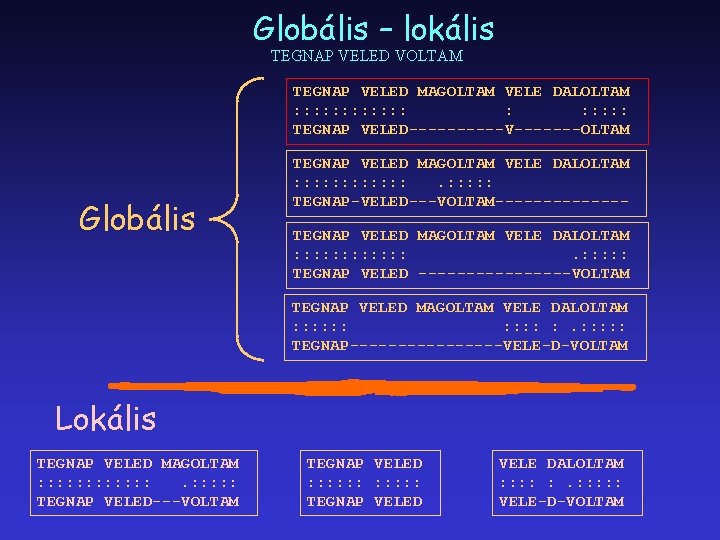
Globális – lokális TEGNAP VELED VOLTAM TEGNAP VELED MAGOLTAM VELE DALOLTAM : : : : : TEGNAP VELED-----V-------OLTAM Globális TEGNAP VELED MAGOLTAM VELE DALOLTAM : : : TEGNAP-VELED---VOLTAM-------TEGNAP VELED MAGOLTAM VELE DALOLTAM : : : TEGNAP VELED --------VOLTAM TEGNAP VELED MAGOLTAM VELE DALOLTAM : : : TEGNAP--------VELE-D-VOLTAM Lokális TEGNAP VELED MAGOLTAM : : : TEGNAP VELED---VOLTAM TEGNAP VELED : : : TEGNAP VELED VELE DALOLTAM : : : VELE-D-VOLTAM

Pontozás • Szekvencia szerkesztés: AGGCCTC – Mutációk AGGACTC – Inszerciók AGGGCCTC – Deléciók AGG. CTC Pontozás: Illeszkedés: Eltérés: Lyuk: +m -s -d Pont: F = (# illeszkedés) m - (# eltérés) s – (#lyukak) d

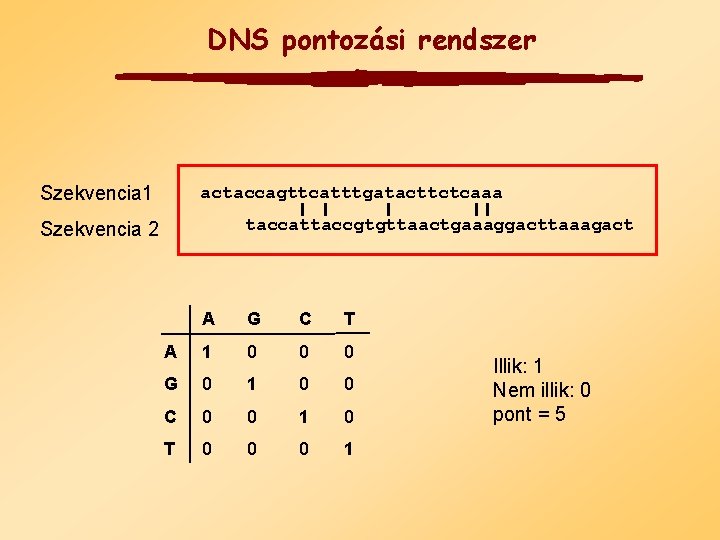
DNS pontozási rendszer Szekvencia 1 actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact Szekvencia 2 A G C T A 1 0 0 0 G 0 1 0 0 C 0 0 1 0 T 0 0 0 1 Illik: 1 Nem illik: 0 pont = 5

Szekvenciák illesztése…. actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact Szekvencia 1 Szekvencia 2 actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact

DNS pontozási rendszer Szekvencia 1 actaccagttcatttgatacttctcaaa taccattaccgtgttaactgaaaggacttaaagact Szekvencia 2 Negatív érték bünteti az eltéréseket: A T C G A 5 -4 -4 -4 T -4 5 -4 -4 C -4 -4 G -4 -4 -4 5 Illik: 5 Nem illik: 19 Score: 5 x 5 + 19 x (-4) = - 51

Dotplots Illeszkedési Mátrix A T G C S W R Y K M B V H D N U A 5 -4 -4 1 1 -4 -4 1 -4 -1 -1 - 2 -4 T -4 5 -4 -4 -4 1 1 -4 -1 - 1 -2 5 A G -4 -4 5 -4 1 -4 -1 -2 -4 C -4 -4 -4 5 1 -4 - 4 1 -1 -1 -1 -4 -2 -4 S -4 -4 1 1 -1 - 4 - 2 -2 -1 - 1 -3 -3 -1 -4 W 1 1 -4 -4 -4 -1 -2 -2 -3 -3 -1 -1 -1 1 R 1 -4 -2 -2 -1 -4 -2 -2 -3 -1 - 3 - 1 -1 -4 Y -4 1 -2 -2 -4 -1 -2 -2 -1 -3 -1 1 K -4 1 1 -4 -2 -2 -1 -4 -1 -3 -3 -1 -1 -1 M 1 -4 -4 -1 -2 -2 -4 -1 -3 -1 - 4 B -4 -1 -1 -3 -3 -1 -1 -3 -1 -2 -2 -2 -1 -1 V -1 -4 -1 -1 -1 -3 -3 -1 -2 -1 - 2 -2 -1 -4 H -1 -1 -1 -4 -3 -1 -1 -3 -2 -2 -2 -1 -1 -1 D 1 1 1 4 3 1 1 3 2 2 2 1 1 1 N -2 -2 -1 -1 - 1 -1 -1 -2 U -4 5 -4 -4 -4 1 1 -4 -1 -1 -2 5 A T G C 5 -4 -4 -4 T -4 5 -4 -4 G – 4 -4 5 -4 C -4 -4 -4 5

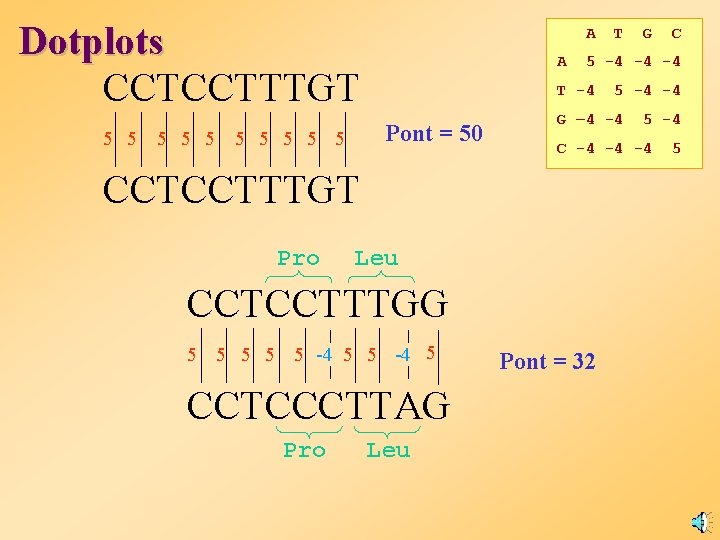
Dotplots CCTCCTTTGT 5 5 5 A A CCTCCTTTGG CCTCCCTTAG Pro Leu 5 -4 C -4 -4 -4 Leu 5 5 5 -4 5 C 5 -4 -4 G – 4 -4 CCTCCTTTGT Pro G 5 -4 -4 -4 T -4 Pont = 50 5 5 5 T Pont = 32 5

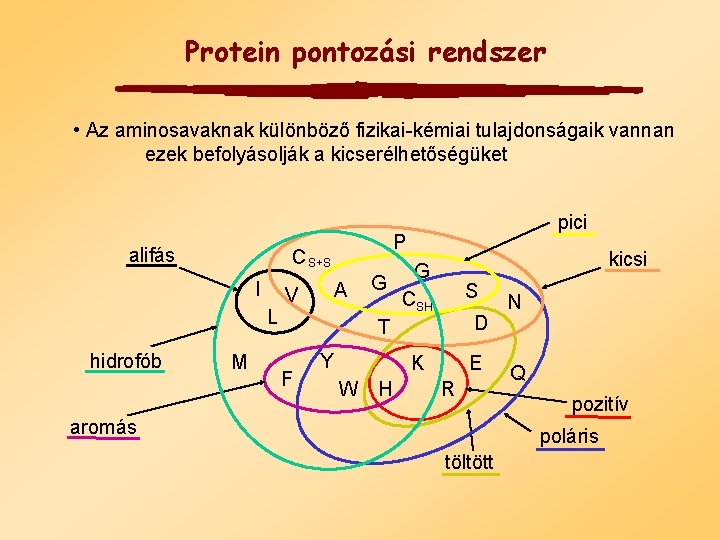
Protein pontozási rendszer • Az aminosavaknak különböző fizikai-kémiai tulajdonságaik vannan ezek befolyásolják a kicserélhetőségüket alifás L hidrofób P C S+S I M V F pici A kicsi G G CSH T Y S D K W H E R aromás N Q pozitív poláris töltött

Fehérje pontozási rendszer • Az aminosavaknak különböző fizikai-kémiai tulajdonságaik vannan ezek befolyásolják a kicserélhetőségüket • Pontozó mátrixnak tükröznie kell • a kölcsönös szubsztitúciók valószínűségét • az aminosavak előfordulási valószínűségét • Általánosan használt mátrixok: • PAM • BLOSUM

PAM (Percent Accepted Mutations) mátrixok • Fehérje családokból globál illesztéséből származik • A család tagjai legalább 85%-osan azonosak (Dayhoff et al. , 1978) • Filogenetikus fa konstrukciója és ősi eredő szekvencia minden fehérje családra • aminosav cserék számítógépes analízise

PAM 250 A R N D C Q E G H I L K M F P S T W Y V B Z W A 2 -2 0 0 1 -1 -1 -2 -1 -1 -3 1 1 1 -6 -3 0 2 1 R -2 6 0 -1 -4 1 -1 -3 2 -2 -3 3 0 -4 0 0 -1 2 -4 -2 1 2 N 0 0 2 2 -4 1 1 0 2 -2 -3 1 -2 -3 0 1 0 -4 -2 -2 4 3 D 0 -1 2 4 -5 2 3 1 1 -2 -4 0 -3 -6 -1 0 0 -7 -4 -2 5 4 C C -2 -4 -4 -5 12 -5 -5 -3 -3 -2 -6 -5 -5 -4 -3 0 -2 -8 0 -2 -3 -4 Q 0 1 1 2 -5 4 2 -1 3 -2 -2 1 -1 -5 0 -1 -1 -5 -4 -2 3 5 -8 E 0 -1 1 3 -5 2 4 0 1 -2 -3 0 -2 -5 -1 0 0 -7 -4 -2 4 5 G 1 -3 0 1 -3 -1 0 5 -2 -3 -4 -2 -3 -5 0 1 0 -7 -5 -1 2 1 H -1 2 2 1 -3 3 1 -2 6 -2 -2 0 -1 -1 -3 0 -2 3 3 I -1 -2 -2 -2 -3 -2 5 2 -2 2 1 -2 -1 0 -5 -1 4 -1 -1 L -2 -3 -3 -4 -6 -2 -3 -4 -2 2 6 -3 4 2 -3 -3 -2 -2 -1 K -1 3 1 0 -5 1 0 -2 -3 5 0 -5 -1 0 0 -3 -4 -2 2 2 M -1 0 -2 -3 -5 -1 -2 -3 -2 2 4 0 6 0 -2 -2 -1 -4 -2 2 -1 0 F -3 -4 -3 -6 -4 -5 -5 -5 -2 1 2 -5 0 9 -5 -3 -3 0 7 -1 -3 -4 P 1 0 0 -1 -3 0 -1 0 0 -2 -3 -1 -2 -5 6 1 0 -6 -5 -1 1 1 S 1 0 0 -1 0 1 -1 -1 -3 0 -2 -3 1 2 1 -2 -3 -1 2 1 T 1 -1 0 0 -2 -1 0 0 -1 0 -2 0 -1 -3 0 1 3 -5 -3 0 2 1 WY W -6 2 -4 -7 -8 -5 -7 -7 -3 -5 -2 -3 -4 0 -6 -2 -5 17 0 -6 -4 -4 -3 -4 -2 -4 0 -4 -4 -5 0 -1 -1 -4 -2 7 -5 -3 -3 0 10 -2 -2 -3 17 V 0 -2 -2 -2 -1 -2 4 2 -2 2 -1 -1 -1 0 -6 -2 4 0 0 B 2 1 4 5 -3 3 4 2 3 -1 -2 2 -1 -3 1 2 2 -4 -2 0 6 5 Z 1 2 3 4 -4 5 5 1 3 -1 -1 2 0 -4 1 1 1 -4 -3 0 5 6

BLOSUM (Blocks Substitution Matrix) • Távoli rokonságban álló fehérjék doménjeinek összehasonlításából (Henikoff & Henikoff, 1992). A A C E C • Minden blokk minden oszlopjában minden aminosav előfordulását számolják • Az összes blokkból származtatott számokat használják a BLOSUM mátrixokhoz A A C E C A-E C-E A-A C-C =4 =2 =2 =1 =1

BLOSUM (Blocks Substitution Matrix) • A szekvenciákat a blokkokban csoportosítják az azonossági szintjüknek megfelelően. • A klasztereket egy szekvenciaként kezelik. • A különböző BLOSUM mátrixok különböznek abban, hogy hány százalékos szekvenciaazonosságot használtak a klaszterezés során. • A mátrix neve mögötti szám (62 BLOSUM 62 esetén) a százalékos szekvencia azonosságra utal a mátrix képzése során. • Nagyobb számok kisebb evolúciós távolságra utalnak

BLOSUM 50 mátrix H E A G A W G H E E P -2 -1 -1 -2 -1 -4 -2 -2 -1 -1 A -2 -1 5 0 5 -3 0 -2 -1 -1 W -3 -3 -3 15 -3 -3 H 10 0 -2 -2 -2 -3 -2 10 0 0 E 0 6 -1 -3 -3 0 6 6 A -2 -1 5 0 5 -3 0 -2 -1 -1 E 0 6 -1 -3 -3 0 6 6

Melyik mátrixot használjuk ? • Általában lokális hasonlósg keresés során a BLOSUM mátrixok jobban használhatóak, mint PAM mátrixok (Henikoff & Henikoff, 1993). • Amikor közeli rokonságban álló fehérjéket hasonlítunk össze alacsonyabb számú PAM vagy magasabb számú BLOSUM mátrixok ajánlottak, távoli kapcsolatban álló fehérjék esetén a mátrix száma magasabb legyen PAM alacsonyabb BLOSUM mátrix esetén. • A BLOSUM 62 az “alapmátrix” (default) adatbázis kutatás esetén

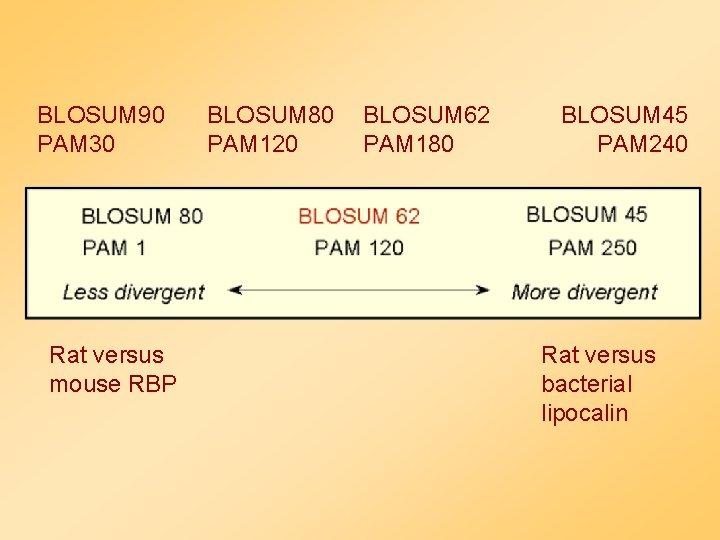
BLOSUM 90 PAM 30 Rat versus mouse RBP BLOSUM 80 PAM 120 BLOSUM 62 PAM 180 BLOSUM 45 PAM 240 Rat versus bacterial lipocalin

Blosum 62 scoring matrix

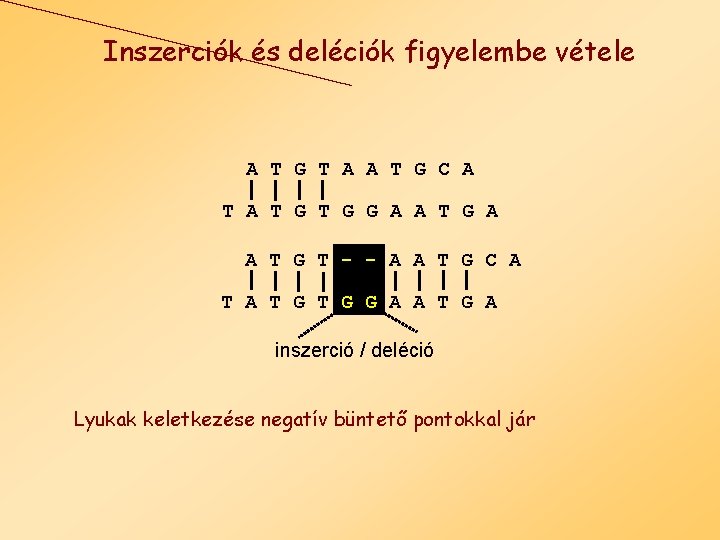
Inszerciók és deléciók figyelembe vétele A T G T A A T G C A T G T G G A A T G T - - A A T G C A T G T G G A A T G A inszerció / deléció Lyukak keletkezése negatív büntető pontokkal jár

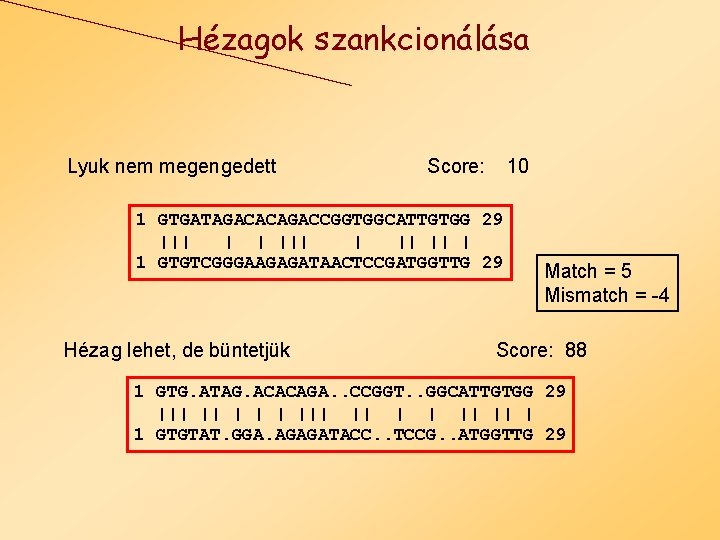
Hézagok szankcionálása Lyuk nem megengedett Score: 10 1 GTGATAGACACAGACCGGTGGCATTGTGG 29 ||| || | 1 GTGTCGGGAAGAGATAACTCCGATGGTTG 29 Hézag lehet, de büntetjük Match = 5 Mismatch = -4 Score: 88 1 GTG. ATAG. ACACAGA. . CCGGT. . GGCATTGTGG 29 ||| || || | 1 GTGTAT. GGA. AGAGATACC. . TCCG. . ATGGTTG 29

Hézagok büntetése • Két szekvencia optimális alignmentje általában • maximálja az illeszkedések • minimalizálja a lyukak számát. • Inszerciók megengedése túl sok magas pontszámú illesztéshez vezetne fals következtetés • Néhány hézag viszont jót tesz az illesztésnek.

Hézagok büntetése matematikailag Lineáris: (g) = - gd Két lépcsős büntetés (Affine gap) : (g) = -d - (g -1)e (g) = g hosszúságú lyuk büntetőpontja d = lyuk nyitás e = lyuk hosszabbítás büntetétőpontja g = hézag hossz

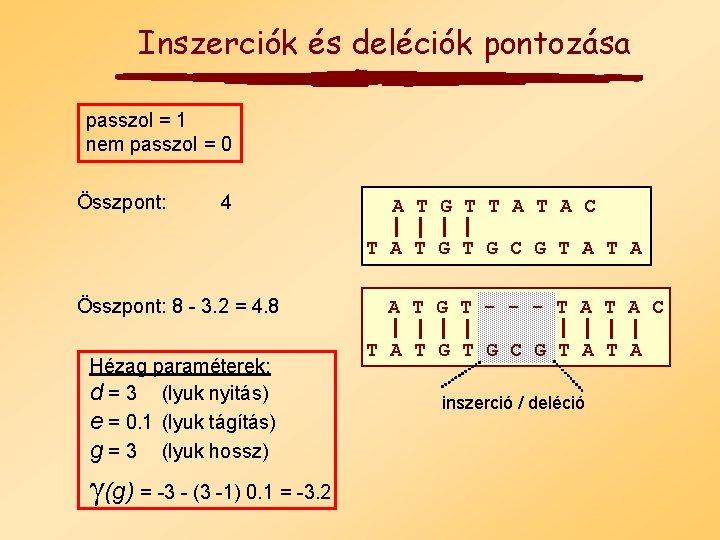
Inszerciók és deléciók pontozása passzol = 1 nem passzol = 0 Összpont: 4 A T G T T A C T A T G C G T A Összpont: 8 - 3. 2 = 4. 8 Hézag paraméterek: d = 3 (lyuk nyitás) e = 0. 1 (lyuk tágítás) g = 3 (lyuk hossz) (g) = -3 - (3 -1) 0. 1 = -3. 2 A T G T - - - T A C T A T G C G T A inszerció / deléció

Alignment típusok • Szigorú algoritmusok - időigényes – Needleman-Wunsch – Smith-Waterman • Heurisztikus algoritmusok – BLAST – FASTA - gyors

A dinamikus programozás alapelvei - Alignment mátrix létrehozása - Pontszámok lépésenként kalkulációja - Visszanyomozás (backtracking) (az optimális út megállapítása)

Az alignment additív Két szekvenciarészlet összevetése x 1…xi y 1…yj xi+1…x. M yj+1…y. N A két pontszám összeadódik: F(x[1: M], y[1: N]) = F(x[1: i], y[1: j]) + F(x[i+1: M], y[j+1: N])

Dinamikus programozás I. • dinamikus programozási algoritmus Tegyük fel, hogy az alábbi két szekvenciát már illesztettük x 1……x. M y 1……y. N Legyen F(i, j) = az illesztés optimális értéke x 1……xi y 1……yj

Dinamikus programozás II. Három lehetséges eset van: 1. xi passzintható yj x 1……xi-1 xi y 1……yj-1 yj 2. xi hézaghoz illik x 1……xi-1 xi y 1……yj - 3. yj hézaghoz illik x 1……xi y 1……yj-1 yj m, ha xi = yj F(i, j) = F(i-1, j-1) + s, ha nem F(i, j) = F(i-1, j) - d F(i, j) = F(i, j-1) - d

Dinamikus programozás III. • Honnan tudjuk, mi a korrekt? Induktív feltételezés: F(i, j-1), F(i-1, j-1) optimális F(i-1, j-1) F(i, j-1) s(xi , yj) F(i-1, j) -d -d F(i, j) Ekkor, F(i, j) = max Ahol F(i-1, j-1) + s(xi, yj) F(i-1, j) – d F( i, j-1) – d s(xi, yj) = m, s(xi, yj) = s, ha xi = yj; ha xi yj ld. mátrixok

Needleman-Wunsch Algoritmus 1. Kezdeti paraméterek. a. b. c. 2. F(0, 0) F(0, j) F(i, 0) = 0 =-j d =-i d Fő iterációk. A mátrix kitöltése a. Minden i = 1……M Minden j = 1……N F(i, j) Ptr(i, j) = max = F(i-1, j-1) + s(xi, yj) F(i-1, j) – d F(i, j-1) – d átló, bal, fel, 3. [1. eset] [2. eset] [3. eset] Termináció. F(M, N) az optimális pont, és Ptr(M, N)-ből az optimális alignment visszanyomozható [1. eset] [2. eset] [3. eset]

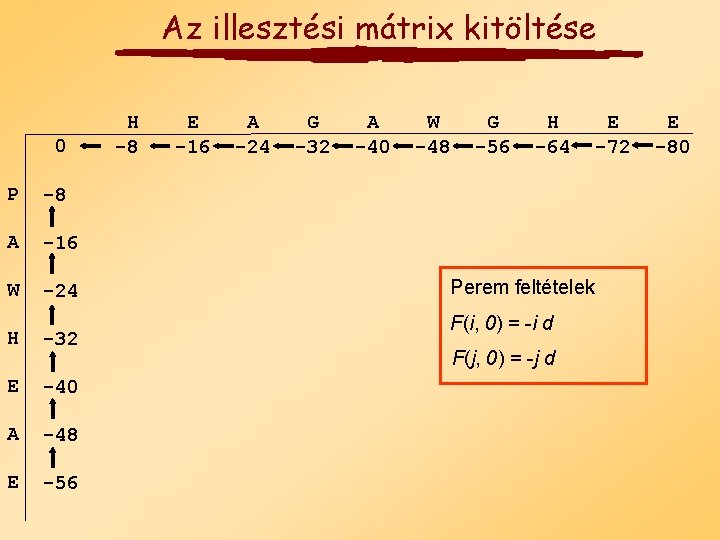
Az illesztési mátrix kitöltése 0 P -8 A -16 W -24 H -32 E -40 A -48 E -56 H -8 E -16 A -24 G -32 A -40 W -48 G -56 H -64 Perem feltételek F(i, 0) = -i d F(j, 0) = -j d E -72 E -80

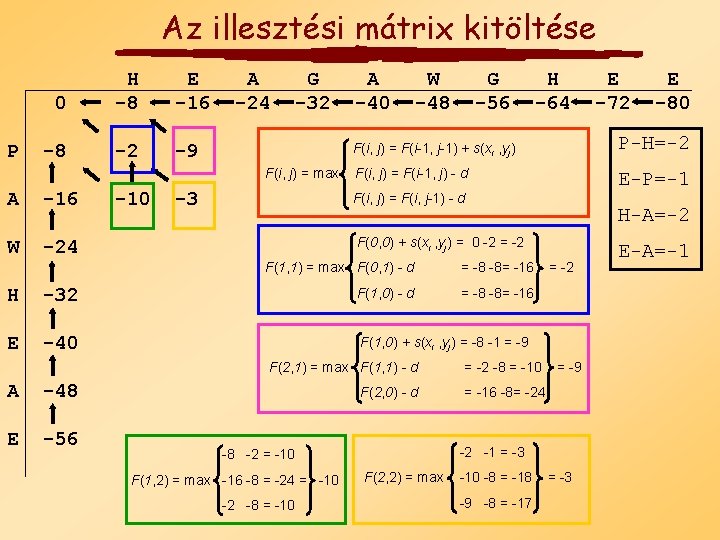
Az illesztési mátrix kitöltése P 0 H -8 E -16 -8 -2 -9 A -24 G -32 F(i, j) = max A -16 W -24 -10 -3 A -40 W -48 G -56 F(i, j) = F(i-1, j) - d E-P=-1 F(i, j) = F(i, j-1) - d F(1, 1) = max F(0, 1) - d = -8 -8= -16 E -40 F(1, 0) + s(xi , yj) = -8 -1 = -9 -56 H-A=-2 F(0, 0) + s(xi , yj) = 0 -2 = -2 F(1, 0) - d E F(2, 1) = max F(1, 1) - d = -2 -8 = -10 F(2, 0) - d = -16 -8= -24 -2 -8 = -10 = -2 = -9 -2 -1 = -3 -8 -2 = -10 F(1, 2) = max -16 -8 = -24 = -10 E -80 P-H=-2 -32 -48 E -72 F(i, j) = F(i-1, j-1) + s(xi , yj) H A H -64 F(2, 2) = max -10 -8 = -18 -9 -8 = -17 = -3 E-A=-1

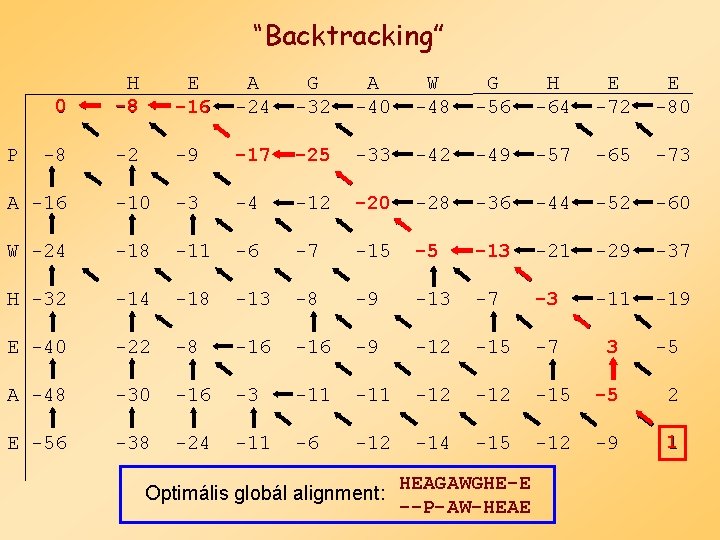
“Backtracking” 0 H -8 E -16 A -24 G -32 A -40 W -48 G -56 H -64 E -72 E -80 -8 -2 -9 -17 -25 -33 -42 -49 -57 -65 -73 A -16 -10 -3 -4 -12 -20 -28 -36 -44 -52 -60 W -24 -18 -11 -6 -7 -15 -5 -13 -21 -29 -37 H -32 -14 -18 -13 -8 -9 -13 -7 -3 -11 -19 E -40 -22 -8 -16 -9 -12 -15 -7 3 -5 A -48 -30 -16 -3 -11 -12 -15 -5 2 E -56 -38 -24 -11 -6 -12 -14 -15 -12 -9 1 P Optimális globál alignment: HEAG AWGHE-E --P- AW-HEAE

Smith - Waterman (lokális alignment) Két különbség: 0 1. F(i, j) = max F(i, j) = F(i-1, j-1) + s(xi , yj) F(i, j) = F(i-1, j) - d F(i, j) = F(i, j-1) - d 2. Az alignment bárhol befejeződhet a mátrixban Példa: Szekvencia 1 Szekvencia 2 HEAGAWGHEE PAWHEAE Mátrix: Lyuk büntetés: BLOSUM Lineáris, d=8

Smith - Waterman alignment 0 H 0 E 0 A 0 G 0 A 0 W 0 G 0 H 0 E 0 P 0 0 0 A 0 0 0 5 0 0 0 W 0 0 2 0 20 12 4 0 0 H 0 10 2 0 0 0 12 18 22 14 6 E 0 2 16 8 0 0 4 10 18 28 20 A 0 0 8 21 13 5 0 4 10 20 27 E 0 0 6 13 18 12 4 0 4 16 26 Optimal local alignment: A WGH E A W-H E

Extended Smith & Waterman Több lokális alignment kapható: • a legjobb útvonal körüli régió törlése • ismételt visszanyomozás (backtracking)

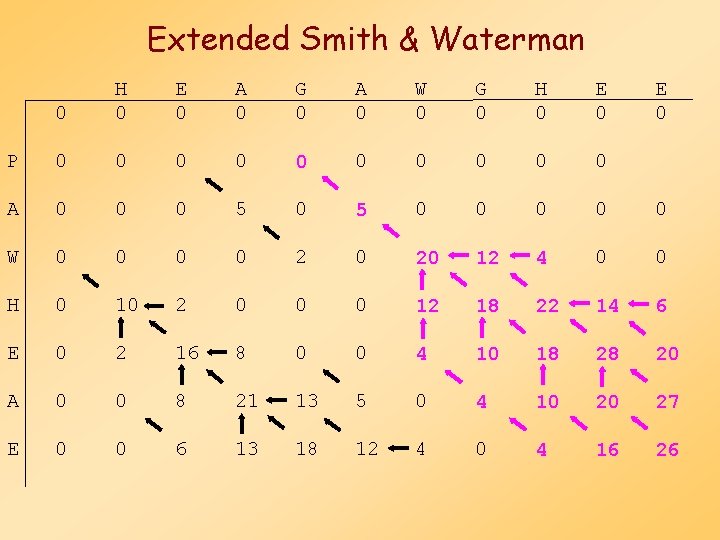
Extended Smith & Waterman 0 H 0 E 0 A 0 G 0 A 0 W 0 G 0 H 0 E 0 P 0 0 0 0 0 A 0 0 0 5 0 0 0 W 0 0 2 0 20 12 4 0 0 H 0 10 2 0 0 0 12 18 22 14 6 E 0 2 16 8 0 0 4 10 18 28 20 A 0 0 8 21 13 5 0 4 10 20 27 E 0 0 6 13 18 12 4 0 4 16 26

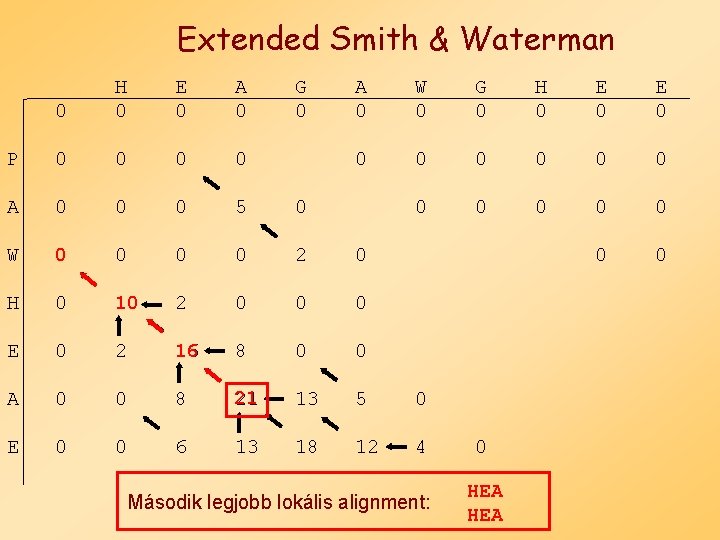
Extended Smith & Waterman 0 H 0 E 0 A 0 G 0 A 0 W 0 G 0 H 0 E 0 P 0 0 0 0 0 A 0 0 0 5 0 0 0 W 0 0 2 0 0 0 H 0 10 2 0 0 0 E 0 2 16 8 0 0 A 0 0 8 21 13 5 0 E 0 0 6 13 18 12 4 Második legjobb lokális alignment: 0 HEA

Heuristic Methods • Fast. A (Pearson and Lipman) • Blast / Blast 2 (Altschul)

Fast. A (Pearson and Lipman) 1. Rövid, rögzített hosszúságú azonos betűsor keresése 2. 3. Minden átló pontszámát meghatározzuk. 4. 5. A 10 legmagasabb pontszámú átlós régiót újrapontozzuk a pontszám táblázatok alapján (kezdeti régiók). A legmagasabb pontszám (score) init 1. Szomszédos kezdeti átlók összekötése. A legmagasabb pontszám (score) initn. Azokra a szekvenciákra, ahol az initn pontszám meghalad egy küszöbértéket (treshold) opt-score, z-score, és E() értéket számolunk. 6. Azokat a szekvenciákat listázzuk, amiknek az E() értéke 7. kisebb, mint egy adott küszöbérték

Példa: Fast. A 1 lépés Rögzített hosszúságú azonos szavak keresése adatbázis szekvencia Szó hossz: DNS: 6 Protein: 2 kereső szekvencia

Fast. A (Pearson and Lipman) 1. Rövid, rögzített hosszúságú azonos betűsor keresése 2. 3. Minden átló pontszámát meghatározzuk. 4. 5. A 10 legmagasabb pontszámú átlós régiót újrapontozzuk a pontszám táblázatok alapján (kezdeti régiók). A legmagasabb pontszám (score) init 1. Szomszédos kezdeti átlók összekötése. A legmagasabb pontszám (score) initn. Azokra a szekvenciákra, ahol az initn pontszám meghalad egy küszöbértéket (treshold) opt-score, z-score, és E() értéket számolunk. 6. Azokat a szekvenciákat listázzuk, amiknek az E() értéke 7. kisebb, mint egy adott küszöbérték

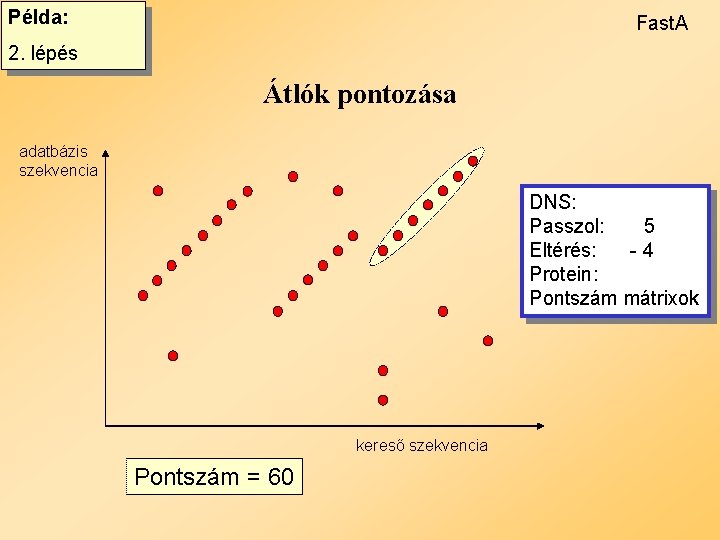
Példa: Fast. A 2. lépés Átlók pontozása adatbázis szekvencia DNS: Passzol: 5 Eltérés: -4 Protein: Pontszám mátrixok kereső szekvencia Pontszám = 60

Fast. A (Pearson and Lipman) 1. Rövid, rögzített hosszúságú azonos betűsor keresése 2. 3. Minden átló pontszámát meghatározzuk. 4. 5. A 10 legmagasabb pontszámú átlós régiót újrapontozzuk a pontszám táblázatok alapján (kezdeti régiók). A legmagasabb pontszám (score) init 1. Szomszédos kezdeti átlók összekötése. A legmagasabb pontszám (score) initn. Azokra a szekvenciákra, ahol az initn pontszám meghalad egy küszöbértéket (treshold) opt-score, z-score, és E() értéket számolunk. 6. Azokat a szekvenciákat listázzuk, amiknek az E() értéke 7. kisebb, mint egy adott küszöbérték

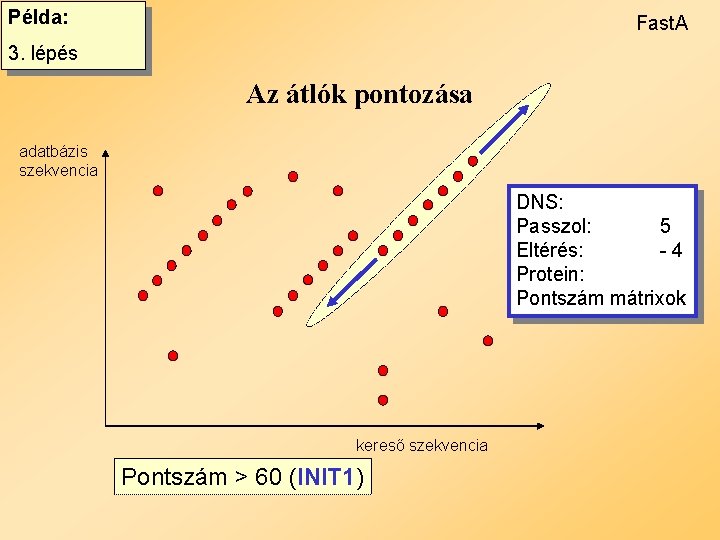
Példa: Fast. A 3. lépés Az átlók pontozása adatbázis szekvencia DNS: Passzol: 5 Eltérés: -4 Protein: Pontszám mátrixok kereső szekvencia Pontszám > 60 (INIT 1)

Fast. A (Pearson and Lipman) 1. Rövid, rögzített hosszúságú azonos betűsor keresése 2. 3. Minden átló pontszámát meghatározzuk. 4. 5. A 10 legmagasabb pontszámú átlós régiót újrapontozzuk a pontszám táblázatok alapján (kezdeti régiók). A legmagasabb pontszám (score) init 1. Szomszédos kezdeti átlók összekötése. A legmagasabb pontszám (score) initn. Azokra a szekvenciákra, ahol az initn pontszám meghalad egy küszöbértéket (treshold) opt-score, z-score, és E() értéket számolunk. 6. Azokat a szekvenciákat listázzuk, amiknek az E() értéke 7. kisebb, mint egy adott küszöbérték

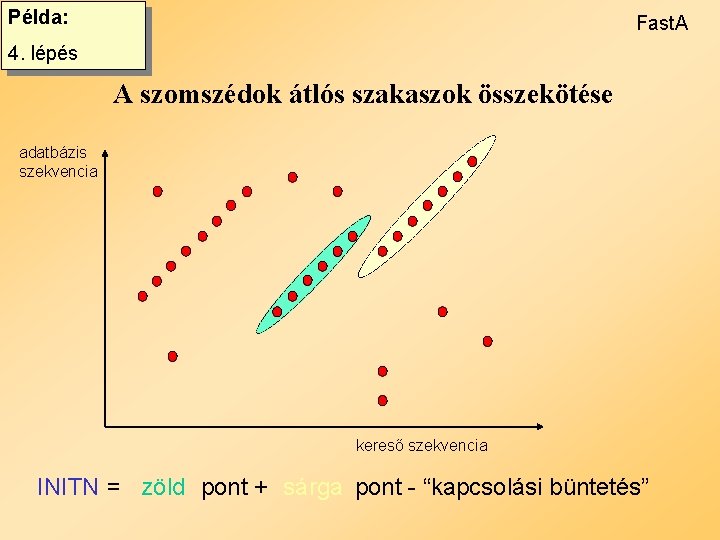
Példa: Fast. A 4. lépés A szomszédok átlós szakaszok összekötése adatbázis szekvencia kereső szekvencia INITN = zöld pont + sárga pont - “kapcsolási büntetés”

Fast. A (Pearson and Lipman) 1. Rövid, rögzített hosszúságú azonos betűsor keresése 2. 3. Minden átló pontszámát meghatározzuk. 4. 5. A 10 legmagasabb pontszámú átlós régiót újrapontozzuk a pontszám táblázatok alapján (kezdeti régiók). A legmagasabb pontszám (score) init 1. Szomszédos kezdeti átlók összekötése. A legmagasabb pontszám (score) initn. Azokra a szekvenciákra, ahol az initn pontszám meghalad egy küszöbértéket (treshold) opt-score, z-score, és E() értéket számolunk. 6. Azokat a szekvenciákat listázzuk, amiknek az E() értéke 7. kisebb, mint egy adott küszöbérték

Fast. A Pontszám kalkuláció 5. lépés Opt-score: Smith-Waterman pontszám Z-score: normalizált az adatbázis szekvencia hosszára Mi az oka a jó pontszámnak? A sorrend vagy az összetétel? Z= (Sc – MSc) / σ E() value A pontszám várható értéke Mi az oka a jó pontszámnak? A homológia vagy a nagy adatbázis? E: annak a valószínűsége, hogy az adott (homológiájú) szekvencia véletlen szerűen szerepel az adatbázisban; Az ilyen homológiát mutató szekvenciák várható száma

Fast. A (Pearson and Lipman) 1. Rövid, rögzített hosszúságú azonos betűsor keresése 2. 3. Minden átló pontszámát meghatározzuk. 4. 5. A 10 legmagasabb pontszámú átlós régiót újrapontozzuk a pontszám táblázatok alapján (kezdeti régiók). A legmagasabb pontszám (score) init 1. Szomszédos kezdeti átlók összekötése. A legmagasabb pontszám (score) initn. Azokra a szekvenciákra, ahol az initn pontszám meghalad egy küszöbértéket (treshold) opt-score, z-score, és E() értéket számolunk. 6. Azokat a szekvenciákat listázzuk, amiknek az E() értéke 7. kisebb, mint egy adott küszöbérték

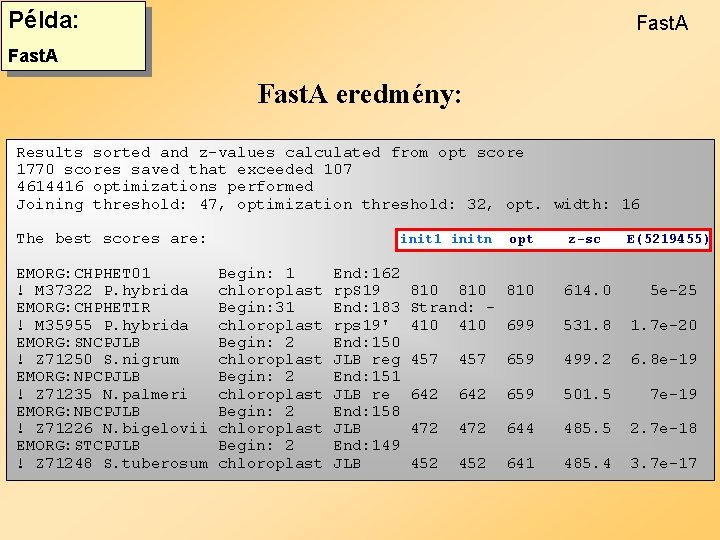
Példa: Fast. A eredmény: Results sorted and z-values calculated from opt score 1770 scores saved that exceeded 107 4614416 optimizations performed Joining threshold: 47, optimization threshold: 32, opt. width: 16 The best scores are: EMORG: CHPHET 01 ! M 37322 P. hybrida EMORG: CHPHETIR ! M 35955 P. hybrida EMORG: SNCPJLB ! Z 71250 S. nigrum EMORG: NPCPJLB ! Z 71235 N. palmeri EMORG: NBCPJLB ! Z 71226 N. bigelovii EMORG: STCPJLB ! Z 71248 S. tuberosum init 1 initn Begin: 1 chloroplast Begin: 31 chloroplast Begin: 2 chloroplast End: 162 rp. S 19 End: 183 rps 19' End: 150 JLB reg End: 151 JLB re End: 158 JLB End: 149 JLB opt z-sc E(5219455) 810 810 Strand: 410 699 614. 0 5 e-25 531. 8 1. 7 e-20 457 659 499. 2 6. 8 e-19 642 659 501. 5 7 e-19 472 644 485. 5 2. 7 e-18 452 641 485. 4 3. 7 e-17

FASTA programok: Fast. A TFast. A Fast. X TFast. X hasonlóság keresés kereső szekvencia és bármilyen típusú szekvencia között(DNS és Protein). peptid szekvenciákat nukleotid szekvenciákkal szemben. nukleotidek szekvenciákat fehérje adatbázissal szemben “frameshift“-eket figyelembe véve. nukleotid szekvenciákat nukleotid szekvencia adatbázissal fehérje szinten.

BLAST (Basic Local Alignment Search Tool) Alapok: 1. query A kereső szekvencia összes lehetséges szavából létrehoz egy szótárat 2. Lokális alignmentet indít minden szóra ami talál párt az adatbázisban Futási idő: O(MN) Nagyságrendekkel gyorsabb, mint a Smith-Waterman DB

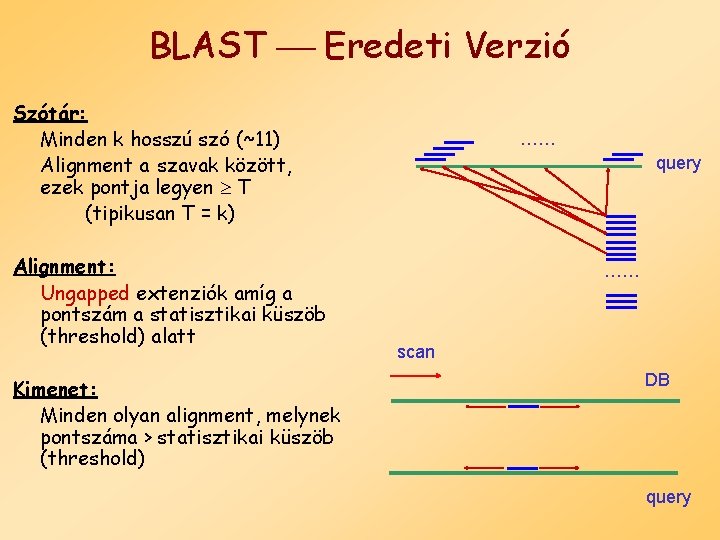
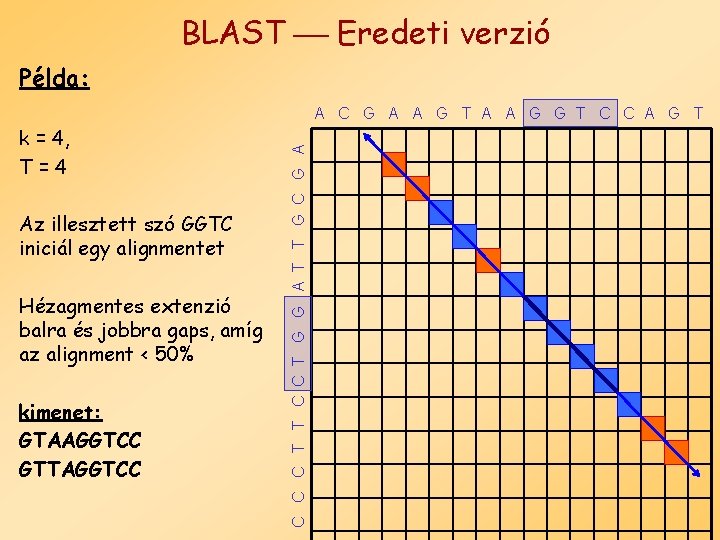
BLAST Eredeti Verzió Szótár: Minden k hosszú szó (~11) Alignment a szavak között, ezek pontja legyen T (tipikusan T = k) Alignment: Ungapped extenziók amíg a pontszám a statisztikai küszöb (threshold) alatt Kimenet: Minden olyan alignment, melynek pontszáma > statisztikai küszöb (threshold) …… query …… scan DB query

BLAST Eredeti verzió Példa: k = 4, T=4 Az illesztett szó GGTC iniciál egy alignmentet Hézagmentes extenzió balra és jobbra gaps, amíg az alignment < 50% kimenet: GTAAGGTCC GTTAGGTCC C T T C C T G G A T T G C G A A G T A A G G T C C A G T

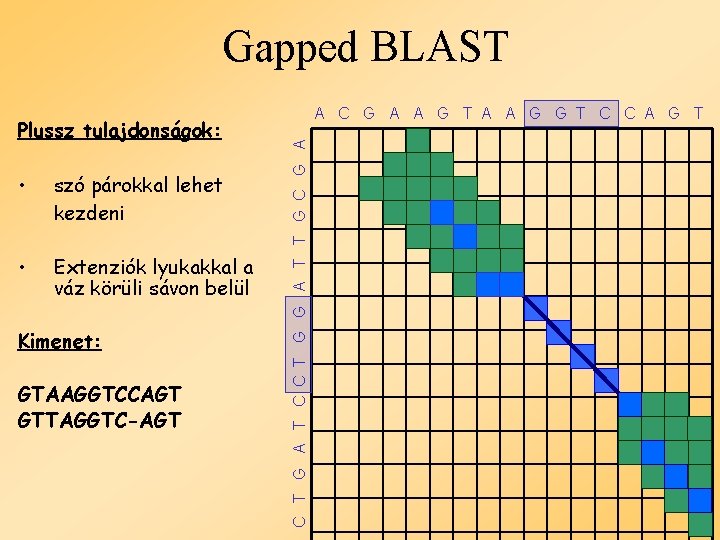
Gapped BLAST • szó párokkal lehet kezdeni • Extenziók lyukakkal a váz körüli sávon belül Kimenet: GTAAGGTCCAGT GTTAGGTC-AGT C T G A T C C T G G A T T G C G A Plussz tulajdonságok: A C G A A G T A A G G T C C A G T

• szó párokkal lehet kezdeni • Közeli alignmentek összeolvasztva • Extenziók hézagokkal amíg a pontszám < T az addigi legjobb pontszám alá kerül Kimenet: GTAAGGTCCAGT GTTAGGTC-AGT A C G A A G T A A G G T C C A G T C T G A T C C T G G A T T G C G A Plussz tulajdonságok: Gapped BLAST

BLAST variációk • MEGABLAST: – Nagyon hasonló szekvenciák összahasonlítására van optimalizálva • Legjobban működik, ha k = 4 i 16 • Lineáris lyuk szankció • PSI-BLAST: – BLAST-tal sok találat – ezeket illesztjük, és mintázatot (pattern) kreálunk – ezt a mintázatot használjuk a következő kereséshez ezeket a lépéseket iteratíve ismételjük • WU-BLAST: (Wash U BLAST) – Optimilizált, extra tulajdonságok • Blast. Z – BLAST/Pattern. Hunter metódus kombinációja

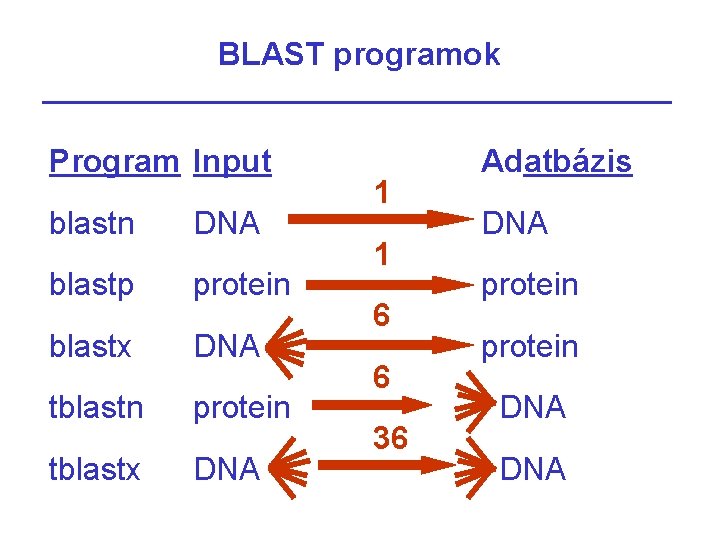
BLAST programok Program Input blastn DNA blastp protein blastx DNA tblastn protein tblastx DNA 1 1 6 6 36 Adatbázis DNA protein DNA
![Query gattacaccccgattaca 29 letters Példa 2 mins Database All Gen BankEMBLDDBJPDB sequences but no Query: gattacaccccgattaca (29 letters) Példa [2 mins] Database: All Gen. Bank+EMBL+DDBJ+PDB sequences (but no](https://slidetodoc.com/presentation_image_h2/d77d6c734a43654e91143bd09433b4b8/image-76.jpg)
Query: gattacaccccgattaca (29 letters) Példa [2 mins] Database: All Gen. Bank+EMBL+DDBJ+PDB sequences (but no EST, STS, GSS, or phase 0, 1 or 2 HTGS sequences) 1, 726, 556 sequences; 8, 074, 398, 388 total letters >gi|28570323|gb|AC 108906. 9| Oryza sativa chromosome 3 BAC OSJNBa 0087 C 10 genomic sequence, complete sequence Length = 144487 Score = 34. 2 bits (17), Expect = 4. 5 Identities = 20/21 (95%) Strand = Plus / Plus Query: Sbjct: 4 tacaccccgattacaccccga 24 ||||||||||||| 125138 tacacccagattacaccccga 125158 Score = 34. 2 bits (17), Expect = 4. 5 Identities = 20/21 (95%) Strand = Plus / Plus Query: Sbjct: 4 tacaccccgattacaccccga 24 ||||||||||||| 125104 tacacccagattacaccccga 125124 >gi|28173089|gb|AC 104321. 7| Oryza sativa chromosome 3 BAC OSJNBa 0052 F 07 genomic sequence, complete sequence Length = 139823 Score = 34. 2 bits (17), Expect = 4. 5 Identities = 20/21 (95%) Strand = Plus / Plus Query: Sbjct: 4 tacaccccgattacaccccga 24 ||||||||||||| 3891 tacacccagattacaccccga 3911
![Query Human atoh enhancer 179 letters 1 5 min Példa Result 57 blast hits Query: Human atoh enhancer, 179 letters [1. 5 min] Példa Result: 57 blast hits](https://slidetodoc.com/presentation_image_h2/d77d6c734a43654e91143bd09433b4b8/image-77.jpg)
Query: Human atoh enhancer, 179 letters [1. 5 min] Példa Result: 57 blast hits 1. gi|7677270|gb|AF 218259. 1|AF 218259 Homo sapiens ATOH 1 enhanc. . . gi|22779500|gb|AC 091158. 11| Mus musculus Strain C 57 BL 6/J ch. . . gi|7677269|gb|AF 218258. 1|AF 218258 Mus musculus Atoh 1 enhanc. . . gi|28875397|gb|AF 467292. 1| Gallus gallus CATH 1 (CATH 1) gene. . . gi|27550980|emb|AL 807792. 6| Zebrafish DNA sequence from clo. . . gi|22002129|gb|AC 092389. 4| Oryza sativa chromosome 10 BAC O. . . gi|22094122|ref|NM_013676. 1| Mus musculus suppressor of Ty. . . gi|13938031|gb|BC 007132. 1| Mus musculus, Similar to suppres. . . 2. 3. 4. 5. 6. 7. 8. 355 1 e-95 264 4 e-68 256 9 e-66 78 5 e-12 54 7 e-05 44 0. 068 42 0. 27 gi|7677269|gb|AF 218258. 1|AF 218258 Mus musculus Atoh 1 enhancer sequence Length = 1517 Score = 256 bits (129), Expect = 9 e-66 Identities = 167/177 (94%), Gaps = 2/177 (1%) Strand = Plus / Plus Query: 3 tgacaatagagggtctggcagaggctcctggccgcggtgcggagcgtctggagca 62 ||||||||||||||||||| Sbjct: 1144 tgacaatagaggggctggcagaggctcctggccccggtgcggagcgtctggagca 1203 Query: 63 cgcgctgtcagctggtgagcgcactctcctttcaggcagctccccggggagctgtgcggc 122 |||||||||||||||| Sbjct: 1204 cgcgctgtcagctggtgagcgcactc-gctttcaggccgctccccggggagctgagcggc 1262 Query: 123 cacatttaacaccatcatcacccctccccggcctcctcaacctcggcctcctcctcg 179 ||||||| || ||||||||||| Sbjct: 1263 cacatttaacaccgtcgtca-ccctccccggcctcctcaacatcggcctcctcctcg 1318