DNS replikci A DNS szerkezete A DNS kmiai


































- Slides: 67

DNS replikáció

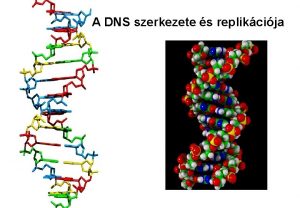
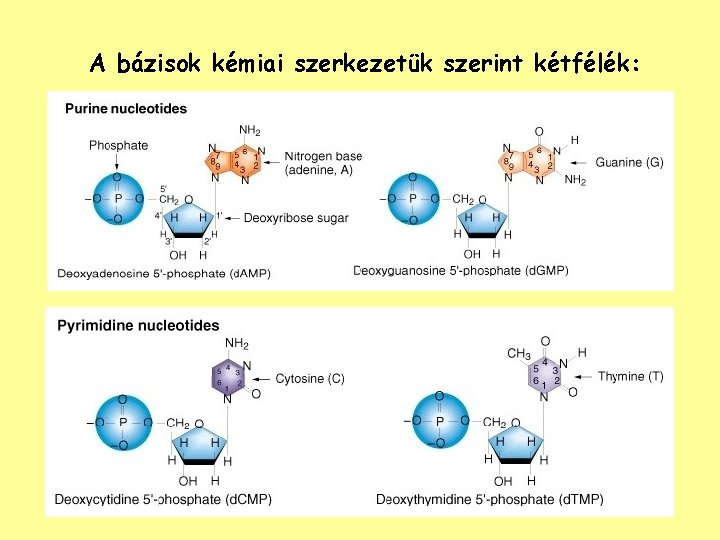
A DNS szerkezete A DNS kémiai felépítésének alapegysége a NUKLEOTID. A nukleotid foszfátot, deoxiribóz cukrot és négy szerves bázisból egyet tartalmaz. Szerkezete: bázis-cukor-foszfát. A négy bázis az adenin, a guanin, a citozin és a timin. A nukleotidok teljes kémiai neve: deoxiadenozin 5’-monofoszfát, d. AMP deoxiguanozin 5’-monofoszfát, d. GMP deoxicitidin 5’-monofoszfát, d. CMP deoxitimidin 5’-monofoszfát, d. TMP A cukor és a bázis alkotta egység a NUKLEOZID: deoxiadenozin, deoxiguanozin, deoxicitidin, deoxitimidin.

A bázisok kémiai szerkezetük szerint kétfélék:

Chargaff szabályok Kölönböző élőlényekből kivonható DNS összetételének vizsgálata érdekes törvényszerűségeket tárt fel. 1. Az élőlényekből származó DNS-ekben a pirimidin nukleotidok (T + C) mennyisége egyenlő a purin (A + G) nukleotidok mennyiségével. 2. a T mennyisége egyenlő az A-val, és C egyenlő G-vel. 3. Azonban A + T és C + G mennyiségek nem feltétlenül egyenlők, azok aránya jellemző az élőlényre amiből a DNS származik.

A DNS szerkezete A kettős spirál 1953 - Watson és Crick. A modell kidolgozása részben a DNS-ről készült u. n. röntgen diffrakciós adatokon, a Chragaff szabályokon, és a DNS és alkotórészeiről felhalmozódott kémiai ismereteken alapul. A DNS B forma röntgen diffrakciós képe

A Watson Crick modell a DNS úgynevezett B formáját írja le. Felismertek más DNS formákat is. Ezek az A és Z formák. Az A forma a DNS vízmentesítése során keletkezik, lényegében a B forma kissé torzult változata. A Z forma különleges nukleotid sorrendek esetén, vagy különleges összetételű oldatban hozható létre. Ebben a szálak nem jobbra, hanem balra tekerednek, lefutásuk nem egyenletes, hanem zegzugos.

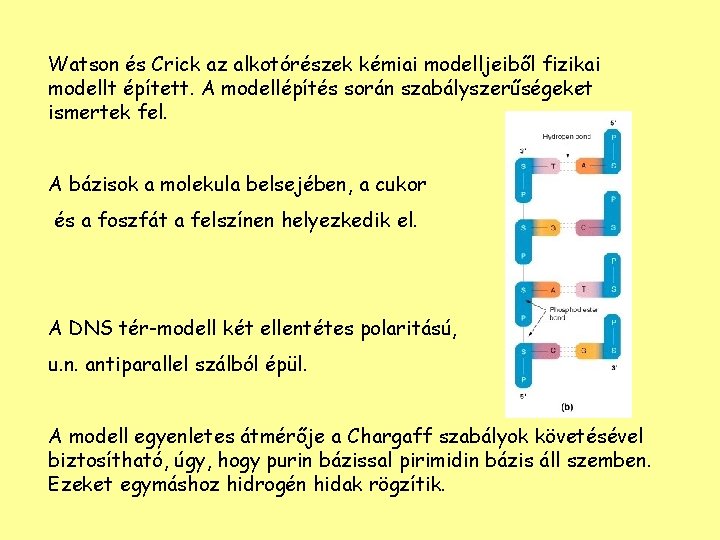
Watson és Crick az alkotórészek kémiai modelljeiből fizikai modellt épített. A modellépítés során szabályszerűségeket ismertek fel. A bázisok a molekula belsejében, a cukor és a foszfát a felszínen helyezkedik el. A DNS tér-modell két ellentétes polaritású, u. n. antiparallel szálból épül. A modell egyenletes átmérője a Chargaff szabályok követésével biztosítható, úgy, hogy purin bázissal pirimidin bázis áll szemben. Ezeket egymáshoz hidrogén hidak rögzítik.

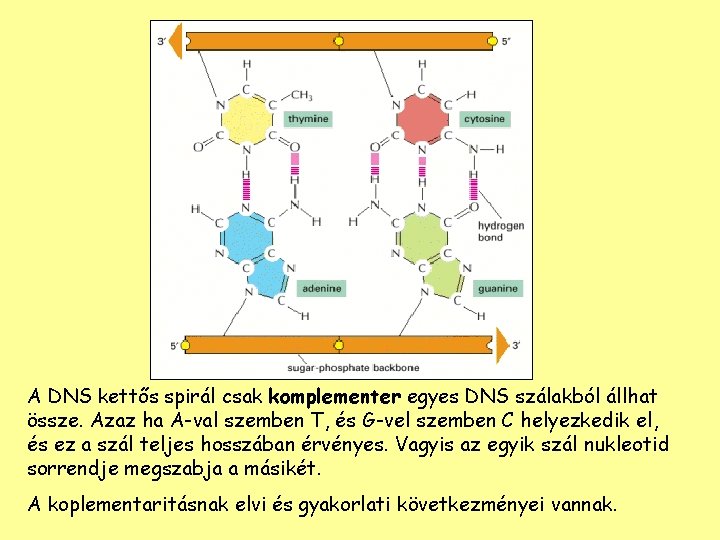
A DNS kettős spirál csak komplementer egyes DNS szálakból állhat össze. Azaz ha A-val szemben T, és G-vel szemben C helyezkedik el, és ez a szál teljes hosszában érvényes. Vagyis az egyik szál nukleotid sorrendje megszabja a másikét. A koplementaritásnak elvi és gyakorlati következményei vannak.

A DNS kettős spirál stabil szerkezet. Az egyik a bázisok közötti hidrogén hidak. A hidrogén híd kötés összetartó ereje ugyan a kovalens kötéseknek csak néhány százaléka, azonban sok van belőle, és a kötést létesítő bázisok rögzítettek, ami fokozza a kapcsolat stabilitását. Az egymás felett elhelyezkedő, lapos, hidrofób bázisok között hidrofób kölcsönhatás, a „stacking” is fontos tényező. Ez úgy működik, mit akkor tapasztalunk, amikor két egymásrafektetett üveglapot próbálunk víz alatt szétválasztani. Ezeket könnyebb egymáson elcsúsztatni, mint szétválasztani. További kölcsönhatás a cukorfoszfát gerinc foszfát csoportjainak egymásra gyakorolt elektrosztatikus taszító hatása.

A szerkezet sajátságaiból két fontos dolog következik: A kettős spirál alakja vagy stabilitása független a nukleotidok sorrendjétől. Kitűnően alkalmas ezért információ tárolásra. A szerkezet alapján könnyen elképzelhető annak megkettőződése, olyan módon, hogy széttekeredik, és az új szálak a régiek nukleotid sorrendjével komplementer módon jön létre.

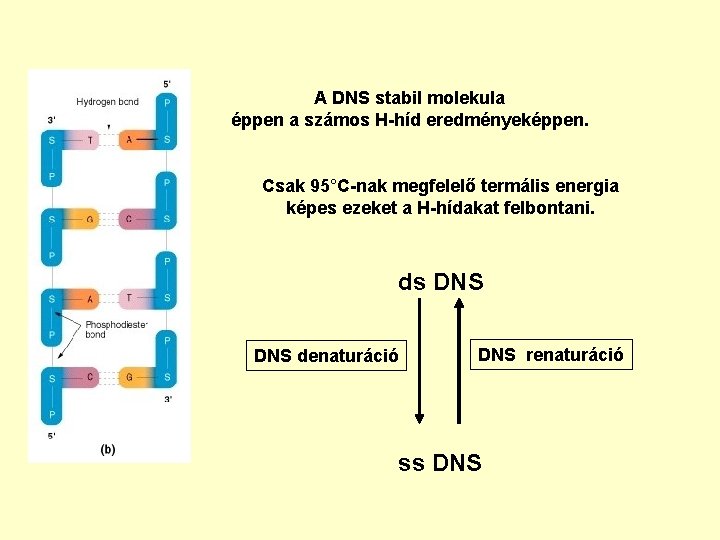
A DNS stabil molekula éppen a számos H-híd eredményeképpen. Csak 95°C-nak megfelelő termális energia képes ezeket a H-hídakat felbontani. ds DNS denaturáció DNS renaturáció ss DNS

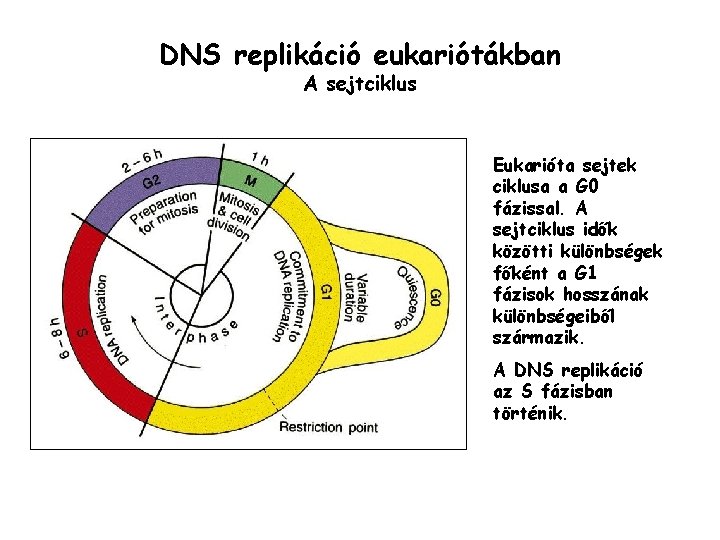
DNS replikáció eukariótákban A sejtciklus Eukarióta sejtek ciklusa a G 0 fázissal. A sejtciklus idők közötti különbségek főként a G 1 fázisok hosszának különbségeiből származik. A DNS replikáció az S fázisban történik.

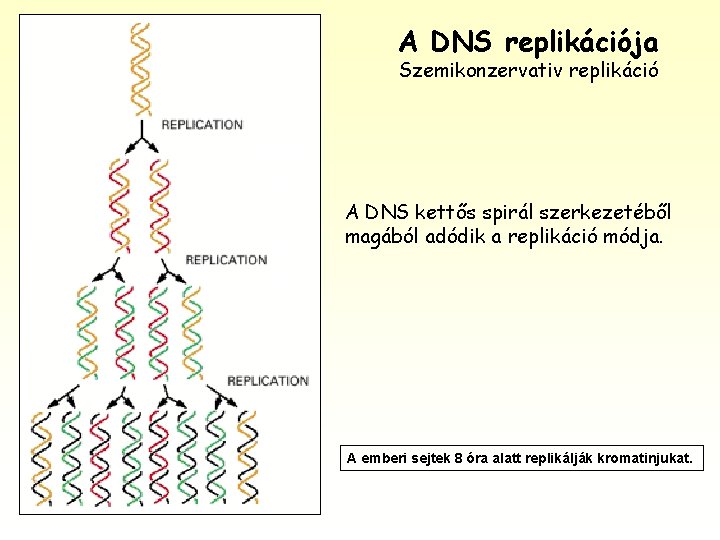
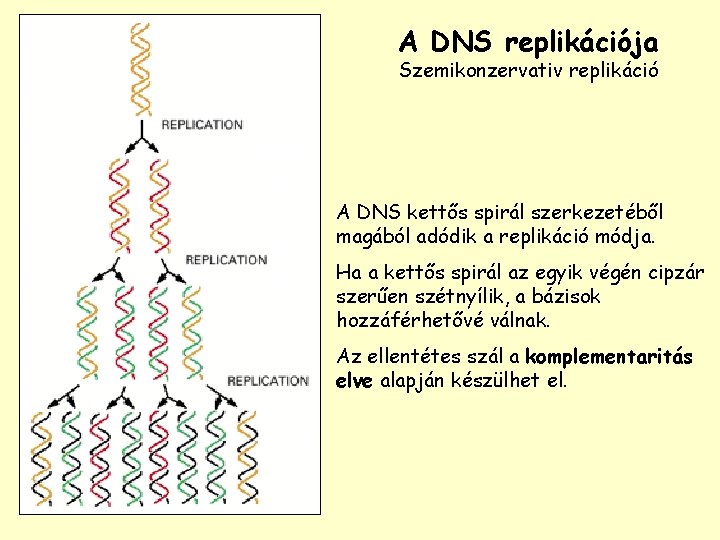
A DNS replikációja Szemikonzervativ replikáció A DNS kettős spirál szerkezetéből magából adódik a replikáció módja. A emberi sejtek 8 óra alatt replikálják kromatinjukat.

A DNS replikációja Szemikonzervativ replikáció A DNS kettős spirál szerkezetéből magából adódik a replikáció módja. Ha a kettős spirál az egyik végén cipzár szerűen szétnyílik, a bázisok hozzáférhetővé válnak. Az ellentétes szál a komplementaritás elve alapján készülhet el.

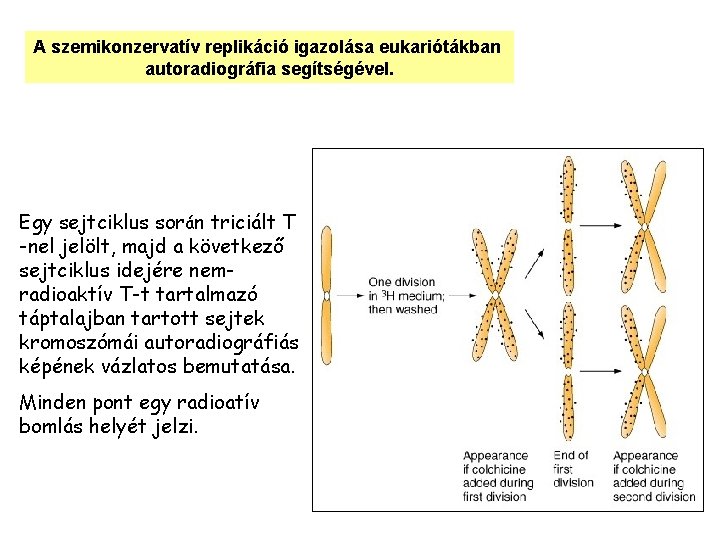
A szemikonzervatív replikáció igazolása eukariótákban autoradiográfia segítségével. Egy sejtciklus során triciált T -nel jelölt, majd a következő sejtciklus idejére nemradioaktív T-t tartalmazó táptalajban tartott sejtek kromoszómái autoradiográfiás képének vázlatos bemutatása. Minden pont egy radioatív bomlás helyét jelzi.

Az autoradiográfiás kép értelmezése. Az első sejtciklusban végén minden DNS kettős spirál egy 3 H-mal jelölt és egy nem jelölt szálból áll. Az ilyen kromoszómák autoradiogramja egyenletes radioaktív jelöltséget mutat. A második sejtciklusban a jelölt és nem jelölt templát DNS szálon szintetizálódik nem jelölt új szál. Egy kromoszóma leánykromatídáinak egyike triciált, a másika nem triciált lesz. A tapasztalt kép teljes összhangban van a DNS replikációja szemikonzervatív jellegével. Ezért úgy véljük az eukarióták DNS-e is szemikonzervatív módon replikálódik.

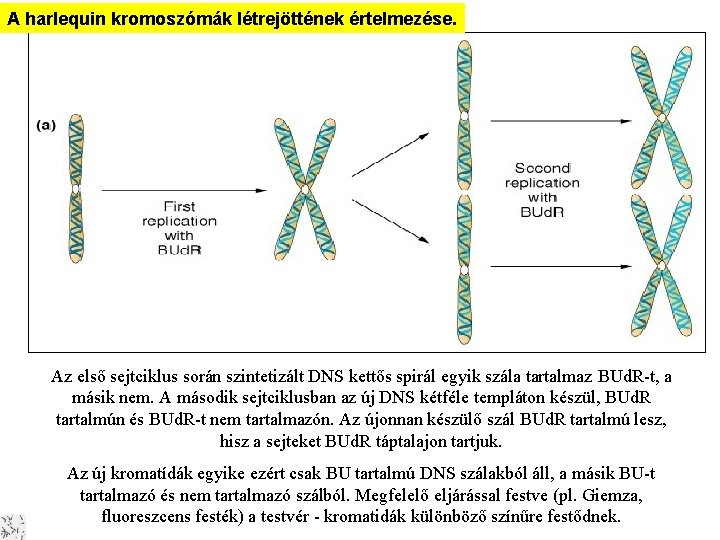
A DNS replikációja Harlekin kromoszómák Az eukarióta DNS szintézis módjának citológiai kimutatására egy másik módszer is kínálkozik. A módszer egy nukleotid analóg a brom-deodxi-uridin (BUd. R) beépülésén alapszik. A replikációs rendszer a BUd. R-t timidinként kezeli és építi be a DNS-be. A mindkét szálában BUd. R-el jelölt kromoszóma azonban nem képes olyan szorosan csomagolódni, mint a normális, és ezért DNS festékekkel fakón festődik a normálishoz képest. Ha két sejtcikluson át BUd. R-en tartjuk a sejteket, kolhicin kezelés után úgynevezett bohóc (harlequin) kromoszómákat kapunk, amelyek egyik leány kromatídája erősen, a másik gyengén festődik.

A harlequin kromoszómák létrejöttének értelmezése. A DNS replikációja Harlekin kromoszómák Az első sejtciklus során szintetizált DNS kettős spirál egyik szála tartalmaz BUd. R-t, a másik nem. A második sejtciklusban az új DNS kétféle templáton készül, BUd. R tartalmún és BUd. R-t nem tartalmazón. Az újonnan készülő szál BUd. R tartalmú lesz, hisz a sejteket BUd. R táptalajon tartjuk. Az új kromatídák egyike ezért csak BU tartalmú DNS szálakból áll, a másik BU-t tartalmazó és nem tartalmazó szálból. Megfelelő eljárással festve (pl. Giemza, fluoreszcens festék) a testvér - kromatidák különböző színűre festődnek.

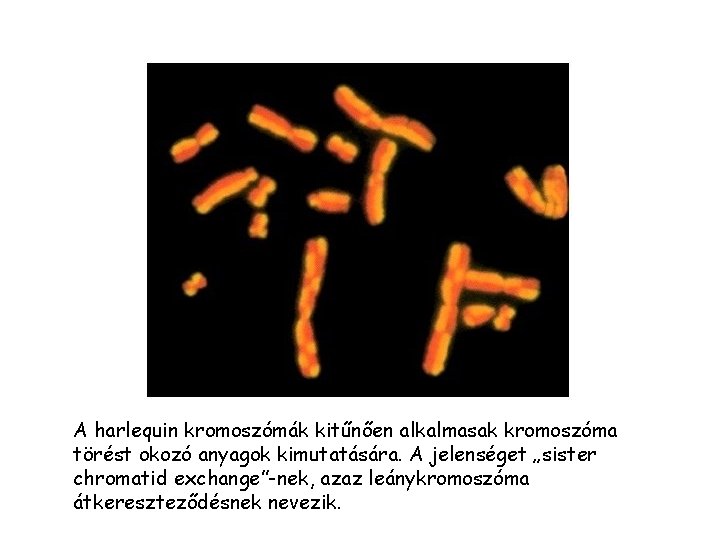
A harlequin kromoszómák kitűnően alkalmasak kromoszóma törést okozó anyagok kimutatására. A jelenséget „sister chromatid exchange”-nek, azaz leánykromoszóma átkereszteződésnek nevezik.

A DNS replikációja Szemikonzervativ replikáció A DNS kettős spirál szerkezetéből magából adódik a replikáció módja. Ha a kettős spirál az egyik végén cipzár szerűen szétnyílik, a bázisok hozzáférhetővé válnak. Az ellentétes szál a komplementaritás elve alapján készülhet el.

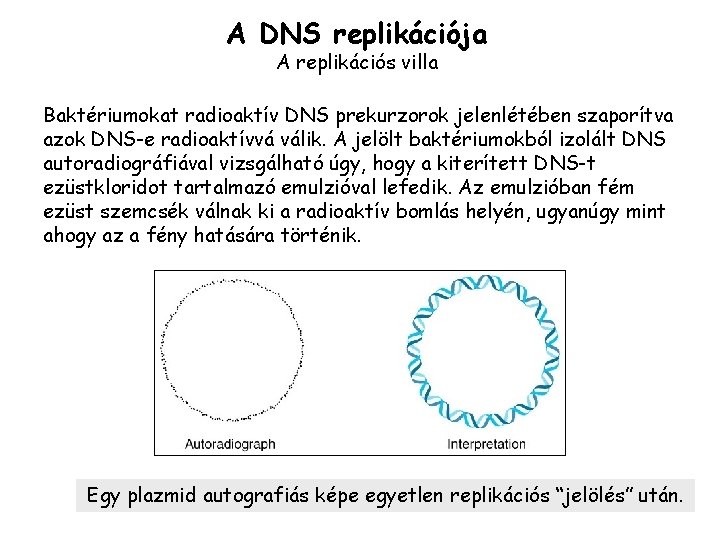
A DNS replikációja A replikációs villa Baktériumokat radioaktív DNS prekurzorok jelenlétében szaporítva azok DNS-e radioaktívvá válik. A jelölt baktériumokból izolált DNS autoradiográfiával vizsgálható úgy, hogy a kiterített DNS-t ezüstkloridot tartalmazó emulzióval lefedik. Az emulzióban fém ezüst szemcsék válnak ki a radioaktív bomlás helyén, ugyanúgy mint ahogy az a fény hatására történik. Egy plazmid autografiás képe egyetlen replikációs “jelölés” után.

Egy replikációs időnél hosszabban tartó jelölés jellegzetes q (téta) szerkezetet eredményez. A belső jelben mindkét szál jelölt. A kör és a belső szál találkozásai a replikációs villák, a DNS szintézis helyei.

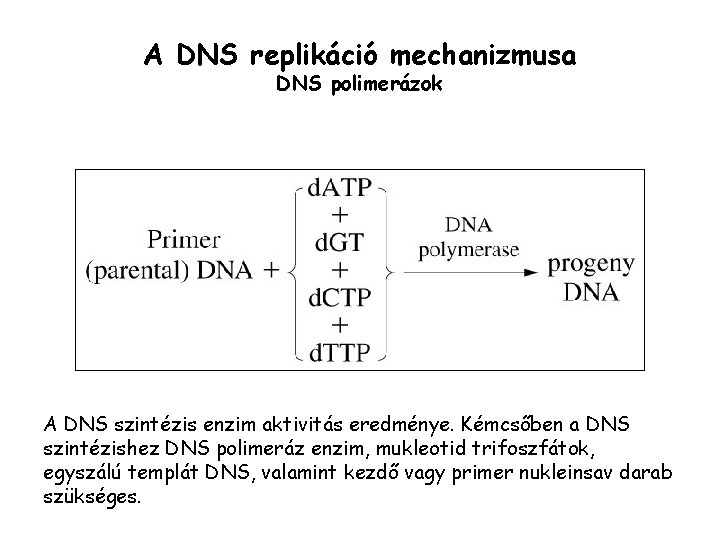
A DNS replikáció mechanizmusa DNS polimerázok A DNS szintézis enzim aktivitás eredménye. Kémcsőben a DNS szintézishez DNS polimeráz enzim, mukleotid trifoszfátok, egyszálú templát DNS, valamint kezdő vagy primer nukleinsav darab szükséges.

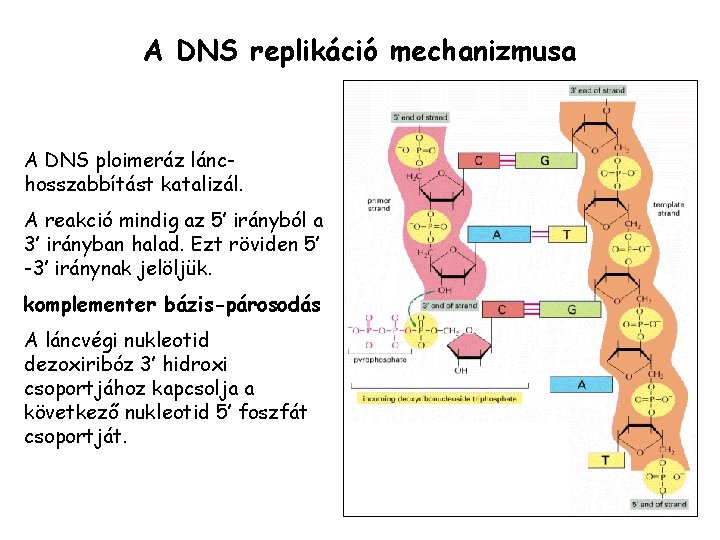
A DNS replikáció mechanizmusa A DNS ploimeráz lánchosszabbítást katalizál. A reakció mindig az 5’ irányból a 3’ irányban halad. Ezt röviden 5’ -3’ iránynak jelöljük. komplementer bázis-párosodás A láncvégi nukleotid dezoxiribóz 3’ hidroxi csoportjához kapcsolja a következő nukleotid 5’ foszfát csoportját.

A DNS szintézis a replikációs origókban kezdődik el. A baktérium kromoszómákon egyetlen replikációs origó működik. Az eukarióták kromoszómáin sok működő replikációs origó mutatható ki. Az E. coli replikációs origója, az ori. C, 245 nukleotidpár hosszú. Közel azonos 13 nukleotid hosszú sorrendek tandem ismétlődéseit, és négy dna. A fehérje kötő helye tartalmaz.

A DNS replikáció mechanizmusa A replikációs origo, a kezdőpont A origóba kezdő szálak, primerek kötnek be. Ezeket hosszabbítják a polimerázok. A replikáció két irányba halad.

The “winding problem” that arises during DNA replication. For a bacterial replication fork moving at 500 nucleotides/second, the parental DNA helix ahead of the fork must rotate at 50 revolutions/second

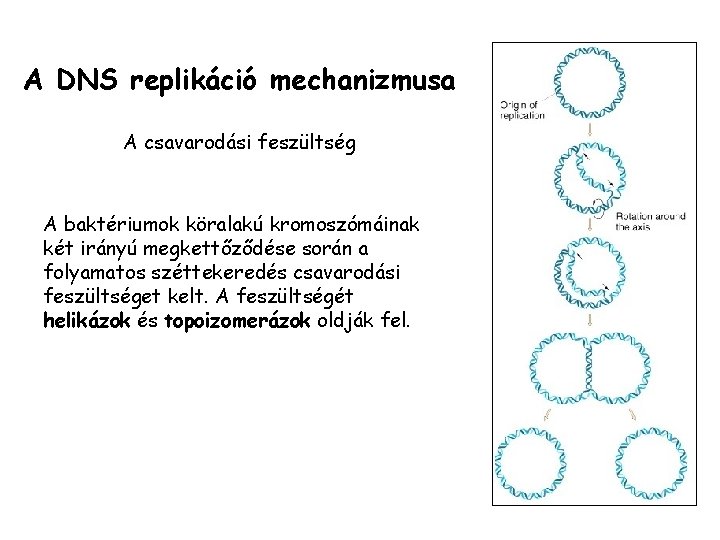
A DNS replikáció mechanizmusa A csavarodási feszültség A baktériumok köralakú kromoszómáinak két irányú megkettőződése során a folyamatos széttekeredés csavarodási feszültséget kelt. A feszültségét helikázok és topoizomerázok oldják fel.


A DNS replikáció mechanizmusa Topoizomerázok Ahogy a replikációs villában szétválnak a szülői szálak, a villa előtti szakasz pozitív irányban túltekeredik. A topoizomeráz oldja fel a feszültséget, hatásának eredménye olyan, mintha egy forgó-láncszem lenne a villa előtt.

DNS replikáció eukariótákban Replikációs origók Autoradiográfiával kimutatható DNS replikációs mintázat. A sejteket rövid 3 H-timidin jelölésnek (pulzus) teszik ki, majd sok nem-radioaktív timidint adva (chase) tartják. Autoradiográfiával kimutatható replikációs mintázat a Drosophila politén kromoszómáján.

DNS replikáció eukariótákban

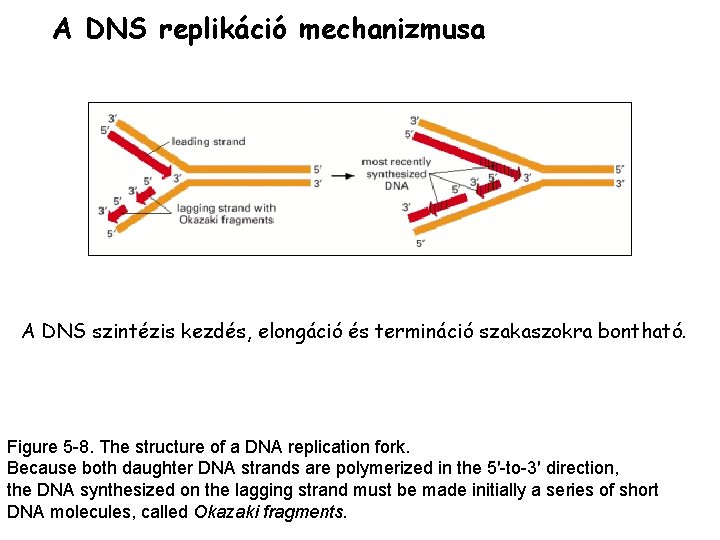
A DNS replikáció mechanizmusa A DNS szintézis kezdés, elongáció és termináció szakaszokra bontható. Figure 5 -8. The structure of a DNA replication fork. Because both daughter DNA strands are polymerized in the 5′-to-3′ direction, the DNA synthesized on the lagging strand must be made initially a series of short DNA molecules, called Okazaki fragments.

A DNS replikáció mechanizmusa A DNS kettős sprál széttekerésében, a replikációs villa kinyílásában topoizomeráz és helikáz enzimek vesz részt. Az egyszálú DNS stabilitását és nukleázok elleni védelmét az SSB (Single Strand Binding) fehérje biztosítja. A kezdő, primer szálak RNS szálak. Ezeket az RNS primáz emzim készíti, ami egy nagy komplex, a primoszóma része. A kezdő szálakat DNS polimeráz hosszabbítja.

A DNS replikáció mechanizmusa A DNS szintézis kezdése, priming A DNS szintézist RNS primer iniciálja

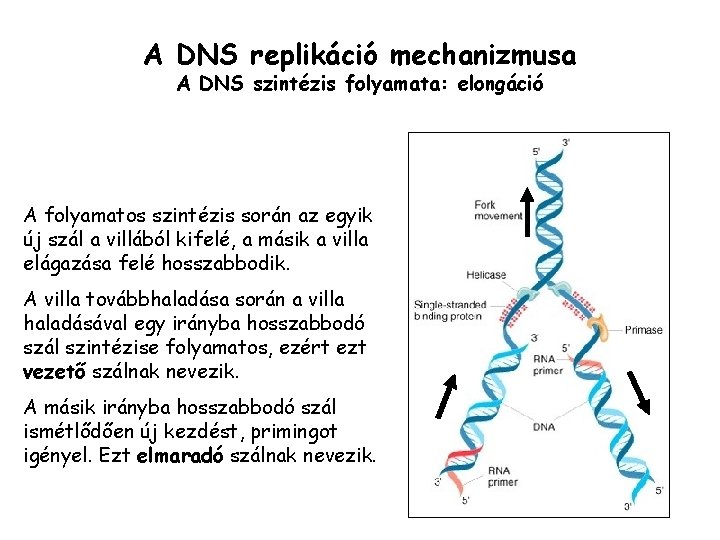
A DNS replikáció mechanizmusa A DNS szintézis folyamata: elongáció A folyamatos szintézis során az egyik új szál a villából kifelé, a másik a villa elágazása felé hosszabbodik. A villa továbbhaladása során a villa haladásával egy irányba hosszabbodó szál szintézise folyamatos, ezért ezt vezető szálnak nevezik. A másik irányba hosszabbodó szál ismétlődően új kezdést, primingot igényel. Ezt elmaradó szálnak nevezik.

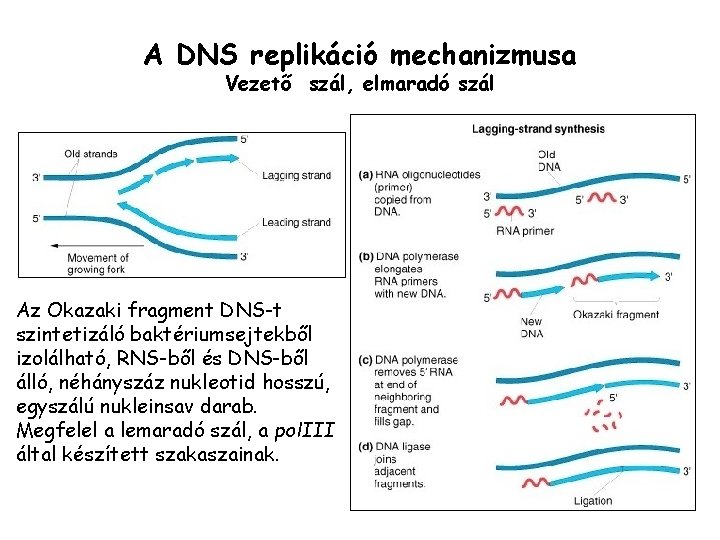
A DNS replikáció mechanizmusa Vezető szál, elmaradó szál Az Okazaki fragment DNS-t szintetizáló baktériumsejtekből izolálható, RNS-ből és DNS-ből álló, néhányszáz nukleotid hosszú, egyszálú nukleinsav darab. Megfelel a lemaradó szál, a pol. III által készített szakaszainak.

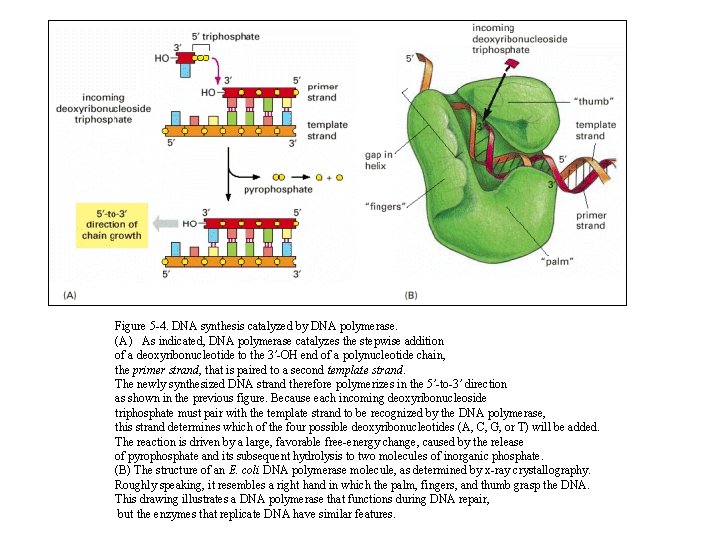
Figure 5 -4. DNA synthesis catalyzed by DNA polymerase. (A) As indicated, DNA polymerase catalyzes the stepwise addition of a deoxyribonucleotide to the 3′-OH end of a polynucleotide chain, the primer strand, that is paired to a second template strand. The newly synthesized DNA strand therefore polymerizes in the 5′-to-3′ direction as shown in the previous figure. Because each incoming deoxyribonucleoside triphosphate must pair with the template strand to be recognized by the DNA polymerase, this strand determines which of the four possible deoxyribonucleotides (A, C, G, or T) will be added. The reaction is driven by a large, favorable free-energy change, caused by the release of pyrophosphate and its subsequent hydrolysis to two molecules of inorganic phosphate. (B) The structure of an E. coli DNA polymerase molecule, as determined by x-ray crystallography. Roughly speaking, it resembles a right hand in which the palm, fingers, and thumb grasp the DNA. This drawing illustrates a DNA polymerase that functions during DNA repair, but the enzymes that replicate DNA have similar features.

A DNS replikáció mechanizmusa A polimerázok Az E. coli DNS replikációjában két polimeráz vesz részt, a DNS polimeráz I (pol I vagy Kornberg enzim) és a DNS polimeráz III (pol III). A pol. III végzi a primerek meghosszabbítását. Addig dolgozik, míg neki nem fut a következő primernek. Ekkor leválik a szubsztrátról. A szintézis zömét a pol. III végzi. A pol. I képes a szabad szálvéghez kapcsolódni, és 5’-3’ exonukleáz aktivitásánál fogva képes maga előtt a primert lebontani, eközben maga mögött szintetizálja az új szálat. Editing by DNA polymerase. Outline of the structures of DNA polymerase complexed with the DNA template in the polymerizing mode (left) and the editing mode (right). The catalytic site for the exonucleolytic (E) and the polymerization (P) reactions are indicated.

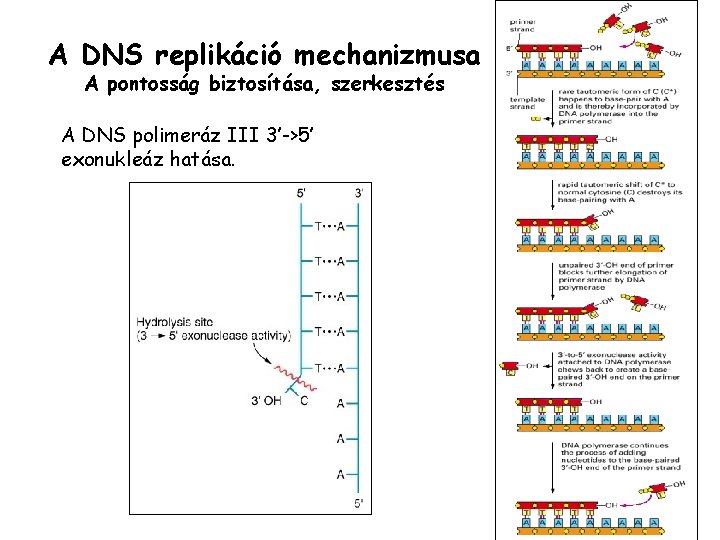
A DNS replikáció mechanizmusa A pontosság biztosítása, szerkesztés A DNS polimeráz III 3’->5’ exonukleáz hatása.

A DNS replikáció mechanizmusa A polimerázok Az E. coli a kromoszómális DNS replikációjában résztvevő polimerázainak főbb tulajdonságai. pol. III 5’->3’ polimerizáció + + 3’->5’ exonukleáz (korrektura) + + 5’->3’ exonukleáz + - 400 10 életképes letális molekula/sejt null allél

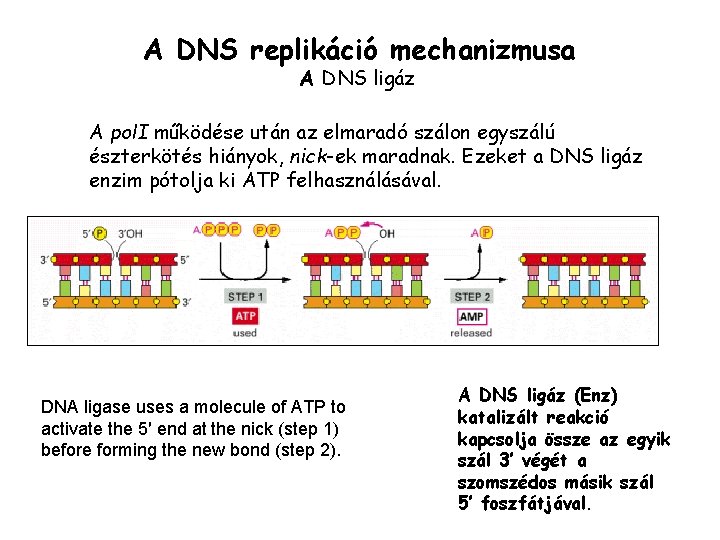
A DNS replikáció mechanizmusa A DNS ligáz A pol. I működése után az elmaradó szálon egyszálú észterkötés hiányok, nick-ek maradnak. Ezeket a DNS ligáz enzim pótolja ki ATP felhasználásával. DNA ligase uses a molecule of ATP to activate the 5′ end at the nick (step 1) before forming the new bond (step 2). A DNS ligáz (Enz) katalizált reakció kapcsolja össze az egyik szál 3’ végét a szomszédos másik szál 5’ foszfátjával.

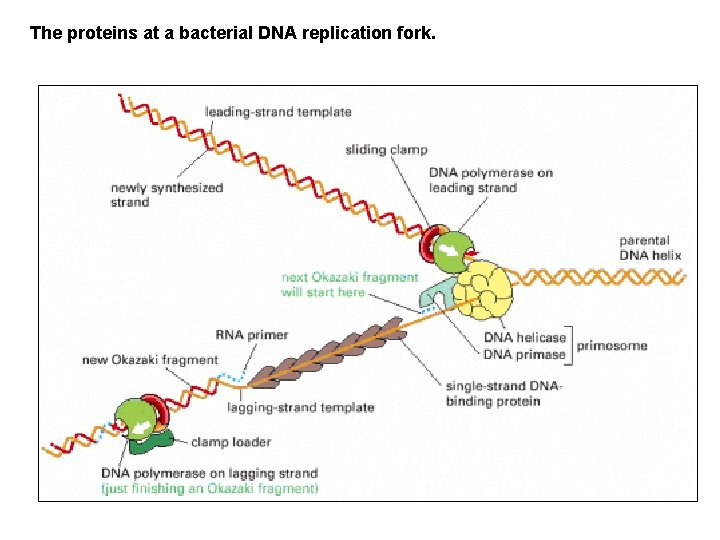
The proteins at a bacterial DNA replication fork.

The mammalian fork differs in at least two important respects. First, it uses two different DNA polymerases on the lagging strand. Second, the mammalian DNA primase is a subunit of one of the lagging-strand DNA polymerases, DNA polymerase α, while that of bacteria is associated with a DNA helicase in the primosome. The polymerase α (with its associated primase) begins chains with RNA, extends them with DNA, and then hands the chains over to the second polymerase (δ), which elongates them. The major mammalian DNA helicase seems to be based on a ring formed from six different Mcm proteins; this ring may move along the leading strand, rather than along the lagging-strand template shown here.

Összegzés Az öröklõdésért felelõs anyag molekuláris természetének feltárására irányuló kísérletes munka egyértelmûen igazolta, hogy a DNS (nem a fehérje, az RNS, vagy más természetû anyag) a genetikai anyag. Mésok adatainak felhasználásával Watson és Crick kettõs spirál modellt készített két, egymás köré tekeredõ, antiparallel irányú DNS szálból. A két szál összekapcsolódásának specifitását az adanin (A) timinhez (T) és a guanin (G) citozinhoz (C) kapcsolódása biztosítja. Ez egyik párt két, az utóbbit három hidrogén híd biztosítja. A Watson-Crick modell sejteti a DNS replikáció módját is, ami a genetikai anyag alaptulajdonsága kell legyen. A replikáció mind a prokariótákban, mind az eukariótákban szemikonzervatív módon történik. Egy kettõs spirál két azonos helixé replikálódik, amelyek mindegyike a nukleotidok lineáris sorrendjében azonos; a keletkezett két kettõs spirál egy eredeti és egy újonnan polimerizálódott DNS láncból áll. A replikáció folyamatában több enzim vesz részt, ezek a DNS polimeráz, a giráz és a helikáz. A megkettõzõdés egy replikációs origónak nevezett speciális DNS szakaszon kezdõdik, és mindkét irányba halad. Mivel a DNS polimeráz csak 5'->3' irányba halad, minden replikációs villában az egyik új szál rövid szakaszokban készül el, amiket azután a ligáz enzim kapcsol össze. A DNS polimeráz nem képes elkezdeni a polimerizációt egy rövid kezdõ, vagy primer szakasz nélkül. A primert egy specifikus enzim készíti el.

DNS repair

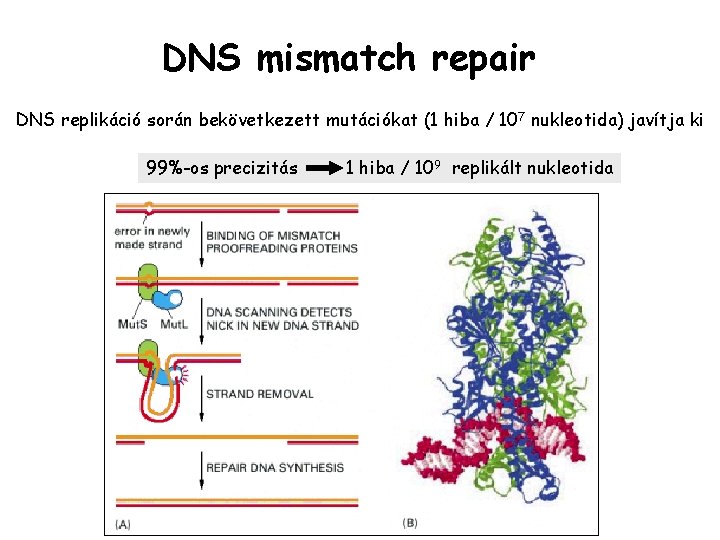
DNS mismatch repair DNS replikáció során bekövetkezett mutációkat (1 hiba / 107 nukleotida) javítja ki 99%-os precizitás 1 hiba / 109 replikált nukleotida

A model for strand-directed mismatch repair in eucaryotes. (A) The two proteins shown are present in both bacteria and eucaryotic cells: Mut. S binds specifically to a mismatched base pair, while Mut. L scans the nearby DNA for a nick. Once a nick is found, Mut. L triggers the degradation of the nicked strand all the way back through the mismatch. Because nicks are largely confined to newly replicated strands in eucaryotes, replication errors are selectively removed. In bacteria, the mechanism is the same, except that an additional protein in the complex (Mut. H) nicks unmethylated (and therefore newly replicated) GATC sequences, thereby beginning the process illustrated here. (B) The structure of the Mut. S protein bound to a DNA mismatch. This protein is a dimer, which grips the DNA double helix as shown, kinking the DNA at the mismatched base pair. It seems that the Mut. S protein scans the DNA for mismatches by testing for sites that can be readily kinked, which are those without a normal complementary base pair. (B, from G. Obmolova et al. , Nature 407: 703– 710, 2000. © Macmillan Magazines Ltd. )

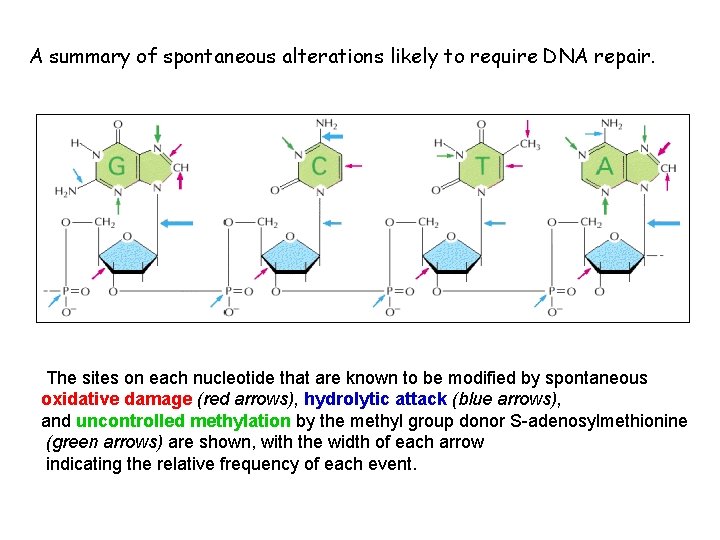
A summary of spontaneous alterations likely to require DNA repair. The sites on each nucleotide that are known to be modified by spontaneous oxidative damage (red arrows), hydrolytic attack (blue arrows), and uncontrolled methylation by the methyl group donor S-adenosylmethionine (green arrows) are shown, with the width of each arrow indicating the relative frequency of each event.

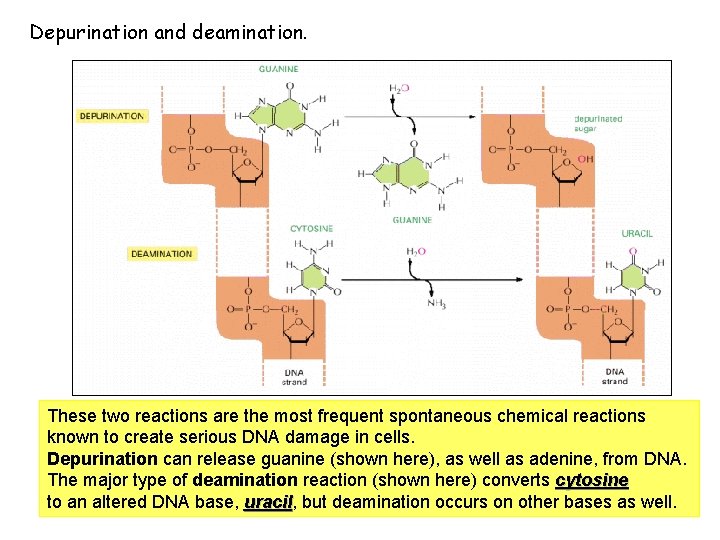
Depurination and deamination. These two reactions are the most frequent spontaneous chemical reactions known to create serious DNA damage in cells. Depurination can release guanine (shown here), as well as adenine, from DNA. The major type of deamination reaction (shown here) converts cytosine to an altered DNA base, uracil, but deamination occurs on other bases as well. uracil

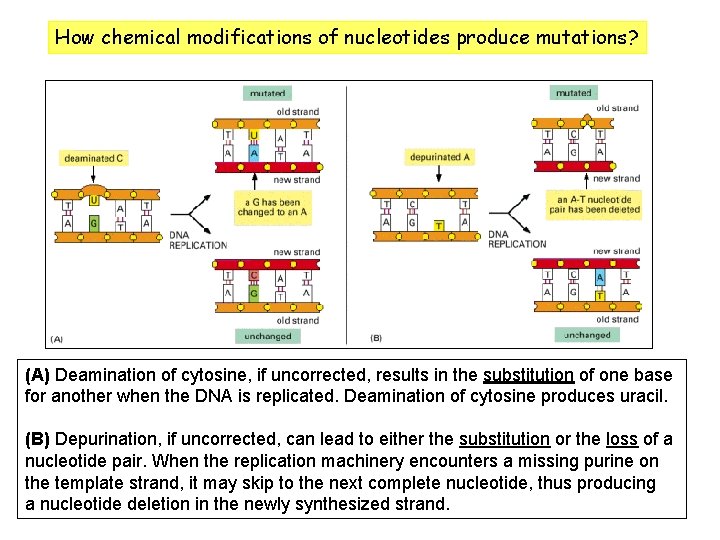
How chemical modifications of nucleotides produce mutations? (A) Deamination of cytosine, if uncorrected, results in the substitution of one base for another when the DNA is replicated. Deamination of cytosine produces uracil. (B) Depurination, if uncorrected, can lead to either the substitution or the loss of a nucleotide pair. When the replication machinery encounters a missing purine on the template strand, it may skip to the next complete nucleotide, thus producing a nucleotide deletion in the newly synthesized strand.

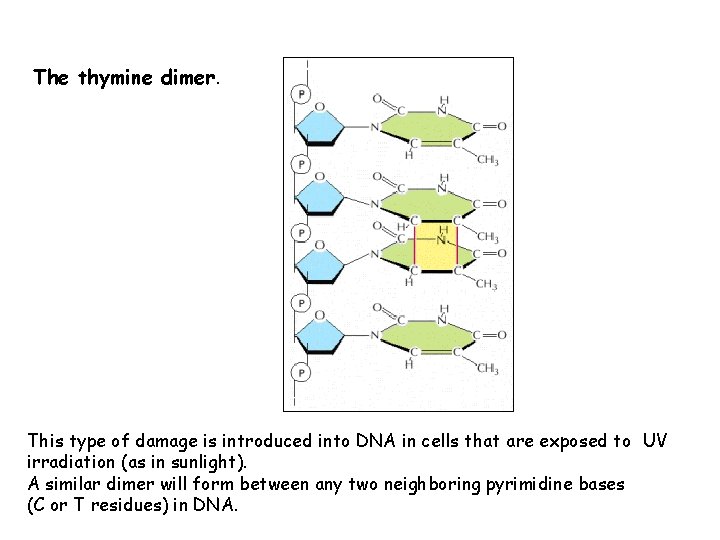
The thymine dimer. This type of damage is introduced into DNA in cells that are exposed to UV irradiation (as in sunlight). A similar dimer will form between any two neighboring pyrimidine bases (C or T residues) in DNA.



General recombination. The breaking and rejoining of two homologous DNA double helices creates two DNA molecules that have “crossed over. ” In meiosis, this process causes each chromosome in a germ cell to contain a mixture of maternally and paternally inherited genes.

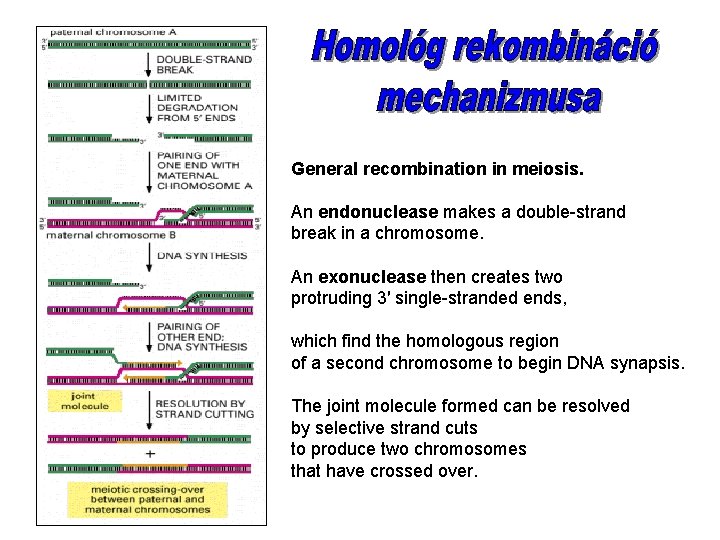
General recombination in meiosis. An endonuclease makes a double-strand break in a chromosome. An exonuclease then creates two protruding 3′ single-stranded ends, which find the homologous region of a second chromosome to begin DNA synapsis. The joint molecule formed can be resolved by selective strand cuts to produce two chromosomes that have crossed over.

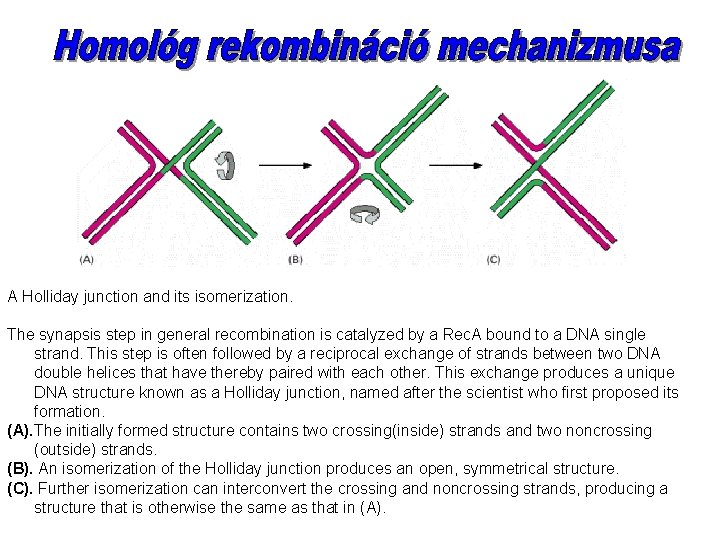
A Holliday junction and its isomerization. The synapsis step in general recombination is catalyzed by a Rec. A bound to a DNA single strand. This step is often followed by a reciprocal exchange of strands between two DNA double helices that have thereby paired with each other. This exchange produces a unique DNA structure known as a Holliday junction, named after the scientist who first proposed its formation. (A). The initially formed structure contains two crossing(inside) strands and two noncrossing (outside) strands. (B). An isomerization of the Holliday junction produces an open, symmetrical structure. (C). Further isomerization can interconvert the crossing and noncrossing strands, producing a structure that is otherwise the same as that in (A).


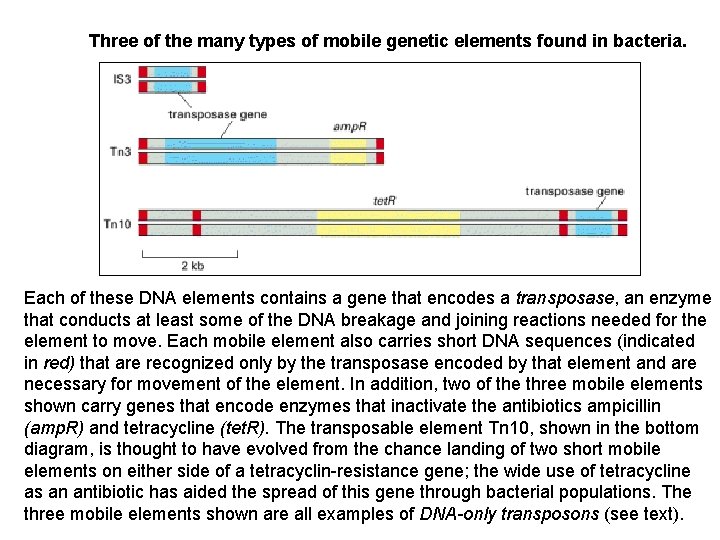
Three of the many types of mobile genetic elements found in bacteria. Each of these DNA elements contains a gene that encodes a transposase, an enzyme that conducts at least some of the DNA breakage and joining reactions needed for the element to move. Each mobile element also carries short DNA sequences (indicated in red) that are recognized only by the transposase encoded by that element and are necessary for movement of the element. In addition, two of the three mobile elements shown carry genes that encode enzymes that inactivate the antibiotics ampicillin (amp. R) and tetracycline (tet. R). The transposable element Tn 10, shown in the bottom diagram, is thought to have evolved from the chance landing of two short mobile elements on either side of a tetracyclin-resistance gene; the wide use of tetracycline as an antibiotic has aided the spread of this gene through bacterial populations. The three mobile elements shown are all examples of DNA-only transposons (see text).

Cut-and-paste transposition DNA-only transposons can be recognized in chromosomes by the “inverted repeat DNA sequences” (red) at their ends. These sequences, which can be as short as 20 nucleotides, are all that is necessary for the DNA between them to be transposed by the particular transposase enzyme associated with the element. The cut-and-paste movement of a DNA-only transposable element from one chromosomal site to another begins when the transposase brings the two inverted DNA sequences together, forming a DNA loop. Insertion into the target chromosome, catalyzed by the transposase, occurs at a random site through the creation of staggered breaks in the target chromosome (red arrowheads). As a result, the insertion site is marked by a short direct repeat of the target DNA sequence, as shown. Although the break in the donor chromosome (green) is resealed, the breakage-and-repair process often alters the DNA sequence, causing a mutation at the original site of the excised transposable element (not shown).

Replicative transposition. In the course of replicative transposition, the DNA sequence of the transposon is copied by DNA replication. The end products are a DNA molecule that is identical to the original donor and a target DNA molecule that has a transposon inserted into it. In general, a particular DNA-only transposon moves either by the cut-and-paste pathway shown in or by the replicative pathway outlined here. However, the two mechanisms have many enzymatic similarities, and a few transposons can move by either pathway.

Retrotranszpozonok RNS – intermedierek keresztül történik a transzpozíció The human genome contains five functional β-globin-like genes (orange); the comparable region from the mouse genome has only four. The positions of the human Alu sequence are indicated by green circles, and the human L 1 sequences by red circles. The mouse genome contains different but related transposable elements: the positions of B 1 elements (which are related to the human Alu sequences) are indicated by blue triangles, and the positions of the mouse L 1 elements (which are related to the human L 1 sequences) are indicated by yellow triangles.

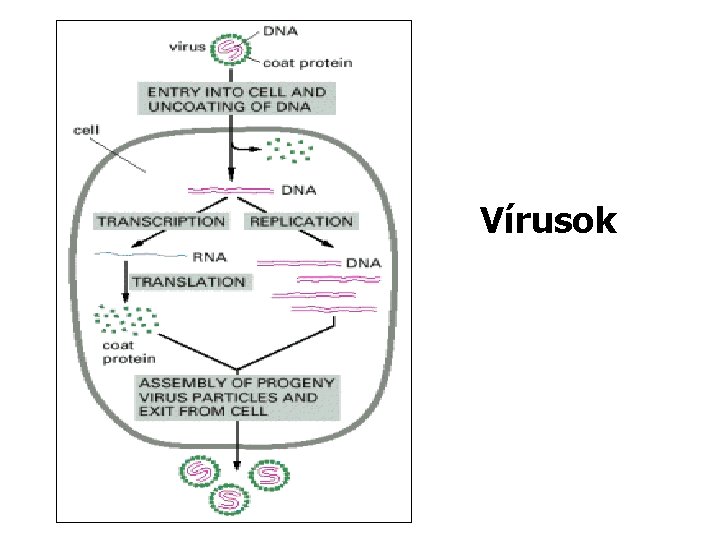
Vírusok

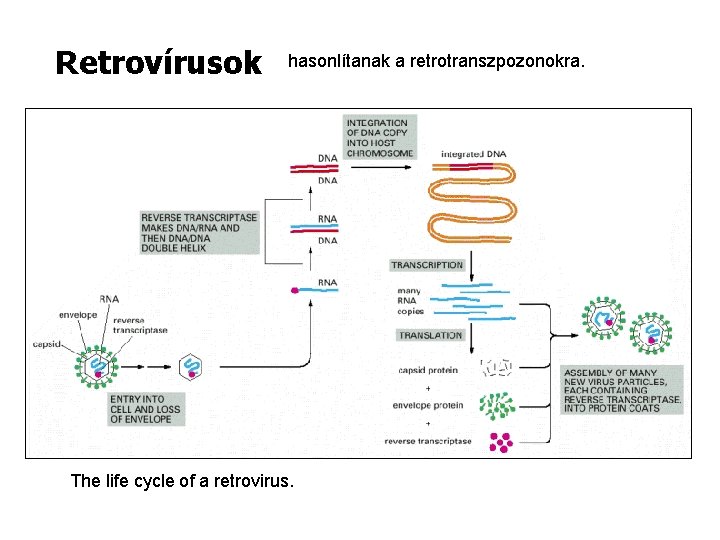
Retrovírusok The retrovirus genome consists of an RNA molecule of about 8500 nucleotides; two such molecules are packaged into each viral particle. The enzyme reverse transcriptase first makes a DNA copy of the viral RNA molecule and then a second DNA strand, generating a double-stranded DNA copy of the RNA genome. The integration of this DNA double helix into the host chromosome is then catalyzed by a virus-encoded integrase enzyme. This integration is required for the synthesis of new viral RNA molecules by the host cell RNA polymerase, the enzyme that transcribes DNA into RNA.

Retrovírusok hasonlítanak a retrotranszpozonokra. The life cycle of a retrovirus.


An explanation for the 5′-to-3′ direction of DNA chain growth. Growth in the 5′-to-3′ direction, allows the chain to continue to be elongated when a mistake in polymerization has been removed by exonucleolytic proofreading. In contrast, exonucleolytic proofreading in the hypothetical 3′-to-5′ polymerization scheme, would block further chain elongation. For convenience, only the primer strand of the DNA double helix is shown
 Revers transkriptaz enzimi nedir
Revers transkriptaz enzimi nedir Dns szerkezete
Dns szerkezete Arna kmiai
Arna kmiai Magnéziumszalag égése
Magnéziumszalag égése Kmiai
Kmiai Kölcsey ferenc himnusz műfaja
Kölcsey ferenc himnusz műfaja Sternum punkciós tű
Sternum punkciós tű Novorapid hatásgörbéje
Novorapid hatásgörbéje Szent andrás törésvonal
Szent andrás törésvonal A föld belső szerkezete
A föld belső szerkezete Novella szerkezete
Novella szerkezete Haimon kreon konfliktus
Haimon kreon konfliktus Mese szerkezete
Mese szerkezete Drma
Drma Kőszívű ember fiai konfliktusok
Kőszívű ember fiai konfliktusok Csontok szerkezete
Csontok szerkezete A világegyetem szerkezete
A világegyetem szerkezete Vin bravais
Vin bravais Retorika tárgya
Retorika tárgya Tökéletes síkság fogalma
Tökéletes síkság fogalma Mi az epika
Mi az epika Biblia szerkezete
Biblia szerkezete Tartuffe szerkezete
Tartuffe szerkezete Egy estém otthon szerkezete
Egy estém otthon szerkezete Thomas kuhn a tudományos forradalmak szerkezete pdf
Thomas kuhn a tudományos forradalmak szerkezete pdf Lobulus hepatis
Lobulus hepatis Ivan iljics halála szerkezete
Ivan iljics halála szerkezete Halmozás jános vitéz
Halmozás jános vitéz Gogol köpönyeg szerkezete
Gogol köpönyeg szerkezete Az elektronburok szerkezete
Az elektronburok szerkezete Iliász szerkezete
Iliász szerkezete Kloroplasztisz szerkezete
Kloroplasztisz szerkezete Goethe hamlet
Goethe hamlet Dns
Dns Response policy zone
Response policy zone Ripe reverse dns
Ripe reverse dns Dns computer center
Dns computer center Kodols
Kodols Dhcppack
Dhcppack How to disable dns lookup in packet tracer
How to disable dns lookup in packet tracer Check glue records
Check glue records Dns
Dns Importance of dns
Importance of dns Le dns
Le dns Nsec dns
Nsec dns Dns 200
Dns 200 Mobility printer server
Mobility printer server Ha dns
Ha dns Curso dns básico
Curso dns básico Dns server korea
Dns server korea Egyptian universities network
Egyptian universities network Salesforce outage dns
Salesforce outage dns Gēnu inženierija
Gēnu inženierija Hinet dns
Hinet dns Umt dns road map
Umt dns road map Local primary name
Local primary name Dns @ vs *
Dns @ vs * Windows dns log parser
Windows dns log parser Dns
Dns Dns xampp
Dns xampp Elsődleges dns kiszolgáló lekérdezése
Elsődleges dns kiszolgáló lekérdezése Dns server with gui
Dns server with gui Dns ho
Dns ho Cirkuláris dns
Cirkuláris dns Domaintools iris passive dns
Domaintools iris passive dns Name
Name Root dns servers locations
Root dns servers locations Dns spoofing
Dns spoofing