Des Protines aux Gnes Premire partie Jrme GARIN








- Slides: 15

Des Protéines aux Gènes … (Première partie) Jérôme GARIN Laboratoire de Chimie des Protéines CEA/INSERM/UJF (Génopole Rhône-Alpes) IN’Tech 2003

L’avènement de la Post-Génomique GENES Génomique (33. 000 gènes) ARNm Transcriptomique PROTEINES (106 Protéomique formes protéiques ? ) • Maturation • Modif. Post-Trad. • Demi-vie • Localisation • Partenaires IN’Tech 2003

Les outils de la Protéomique IN’Tech 2003

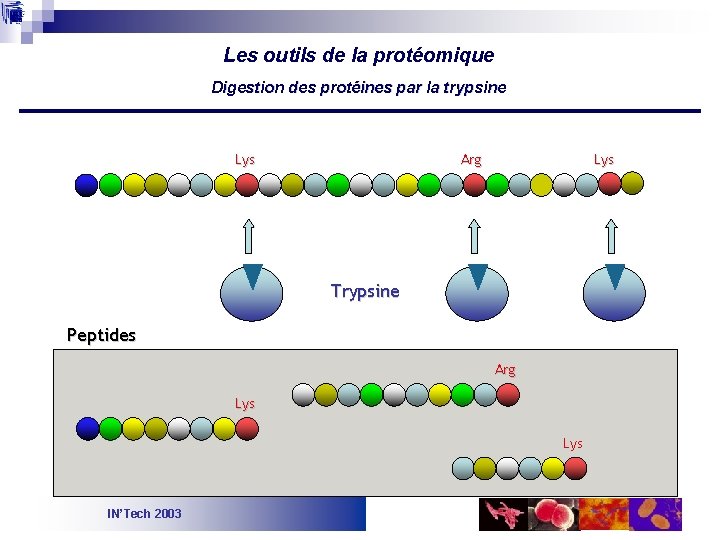
Les outils de la protéomique Digestion des protéines par la trypsine Lys Arg Lys Trypsine Peptides Arg Lys IN’Tech 2003

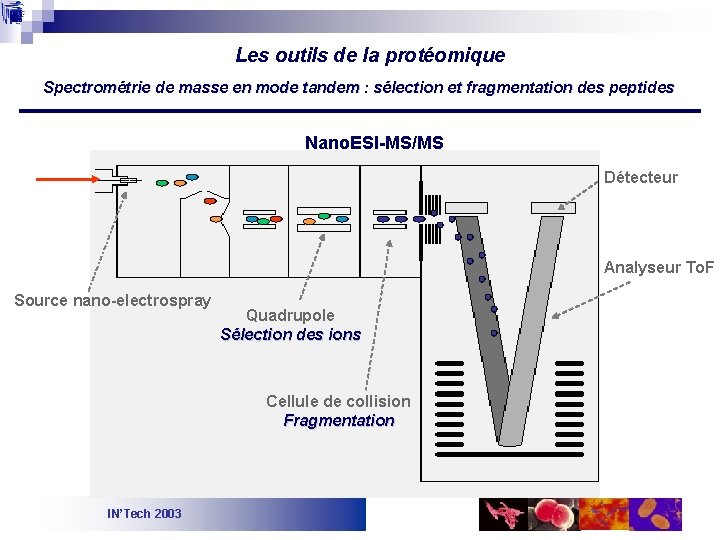
Les outils de la protéomique Spectrométrie de masse en mode tandem : sélection et fragmentation des peptides Nano. ESI-MS/MS Détecteur Analyseur To. F Source nano-electrospray Quadrupole Sélection des ions Cellule de collision Fragmentation IN’Tech 2003

Les outils de la protéomique Séquençage des peptides par nano. ESI-MS/MS R 100 P Q G P M W E 554. 33 159. 09 % 288. 14 272. 17 685. 37 871. 46 1000. 49 0 100 200 300 400 500 600 700 800 900 1000 Banques Protéiques ou Génomiques BLASTP t. BLASTN EWMPGQPR. . VGRRHGSRVSKSAEWMPGQPRPPHLDGSAPGD. . IN’Tech 2003

Les outils de la protéomique Analyses nano. LC-nano. ESI-MS/MS ( « Shot gun Proteomics » ) nano. LC MS/MS Colonne capillaire RP (75 µm x 15 cm) Séparation Peptides + concentration (profil UV) IN’Tech 2003 Spectres MS/MS

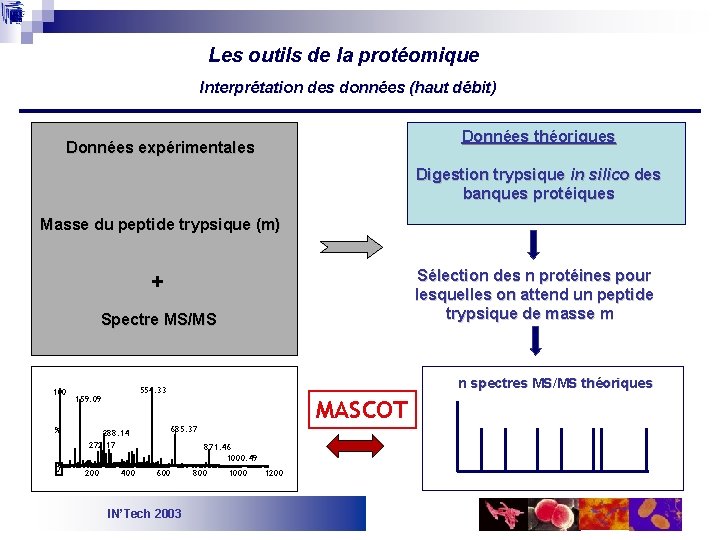
Les outils de la protéomique Interprétation des données (haut débit) Données théoriques Données expérimentales Digestion trypsique in silico des banques protéiques Masse du peptide trypsique (m) Sélection des n protéines pour lesquelles on attend un peptide trypsique de masse m + Spectre MS/MS 100 % 0 159. 09 288. 14 272. 17 200 n spectres MS/MS théoriques 554. 33 400 MASCOT 685. 37 871. 46 1000. 49 600 IN’Tech 2003 800 1000 1200

Les outils de la protéomique Interprétation des données (haut débit) Banques de données protéiques Protéome (spectres MS/MS) IN’Tech 2003

Quelques chiffres … 2003 : nano. LC-MS/MS « Haut débit » : 100 - 1000 protéines/jour « Haute sensibilité » : 20 femtomoles/protéine 1996 : Séquençage d’Edman « Haut débit » : 1 protéine/jour … « Haute sensibilité » : 20 picomoles/protéine IN’Tech 2003

La Protéomique : un outil d’annotation fonctionnelle des Génomes IN’Tech 2003

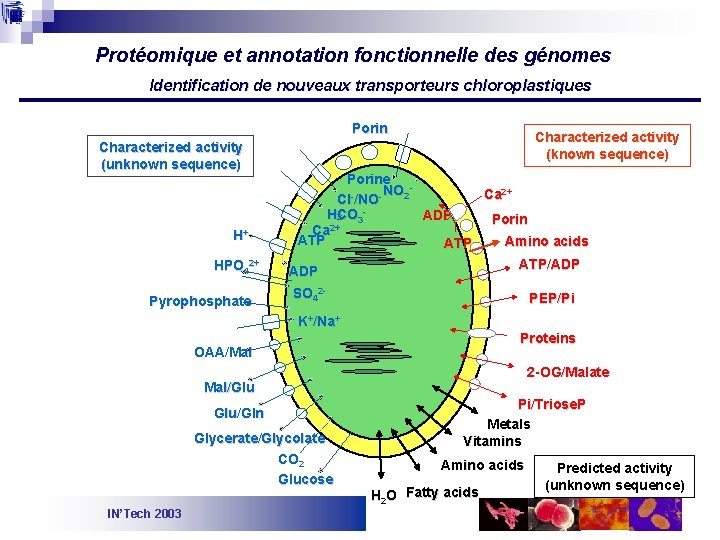
Protéomique et annotation fonctionnelle des génomes Identification de nouveaux transporteurs chloroplastiques Porin Characterized activity (unknown sequence) H+ HPO 42+ Pyrophosphate Characterized activity (known sequence) Porine NO 2 Ca 2+ Cl /NO HCO ADP 2 3 Porin 2+ Ca ATP Amino acids ATP/ADP SO 42 - PEP/Pi K+/Na+ Proteins OAA/Mal 2 -OG/Malate Mal/Glu Glu/Gln Glycerate/Glycolate CO 2 Glucose IN’Tech 2003 Pi/Triose. P Metals Vitamins Amino acids H 2 O Fatty acids Predicted activity (unknown sequence)

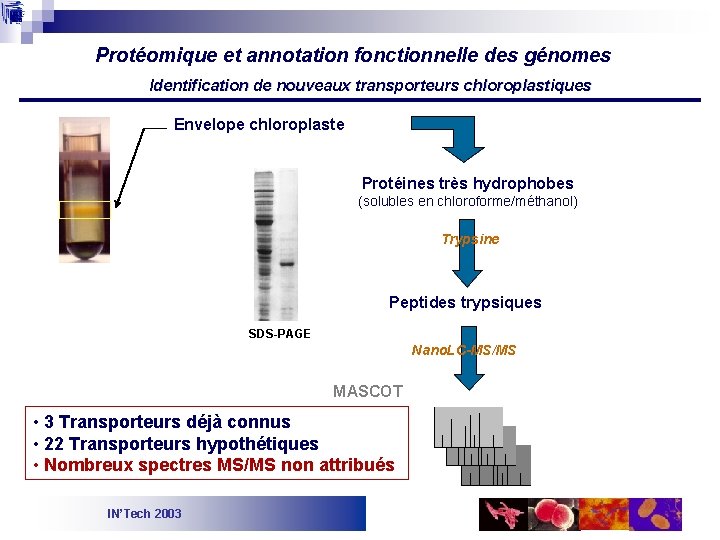
Protéomique et annotation fonctionnelle des génomes Identification de nouveaux transporteurs chloroplastiques Envelope chloroplaste Protéines très hydrophobes (solubles en chloroforme/méthanol) Trypsine Peptides trypsiques SDS-PAGE Nano. LC-MS/MS MASCOT • 3 Transporteurs déjà connus • 22 Transporteurs hypothétiques • Nombreux spectres MS/MS non attribués IN’Tech 2003

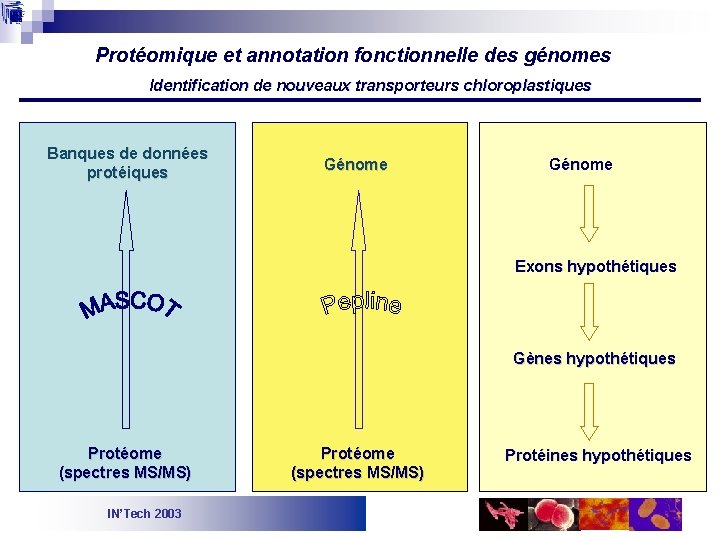
Protéomique et annotation fonctionnelle des génomes Identification de nouveaux transporteurs chloroplastiques Banques de données protéiques Génome Exons hypothétiques Gènes hypothétiques Protéome (spectres MS/MS) IN’Tech 2003 Protéome (spectres MS/MS) Protéines hypothétiques

Myriam FERRO Romain CAHUZAC Gilles ANDRE Jérôme GARIN Jacques JOYARD Norbert ROLLAND Yves VANDENBROUCK IN’Tech 2003 Erwan RÉGUER Estelle NUGUES Alain VIARI