Centro de Cincias Exatas e da Natureza Departamento














- Slides: 33

Centro de Ciências Exatas e da Natureza Departamento de Biologia Molecular Curso: Ciências Biológicas Disciplina: Genética Molecular Replicação do DNA Eleonidas Moura Lima

Transferência da informação genética transcrição DNA duplicação tradução RNA PROTEÍNA

REPLICAÇÃO DO DNA

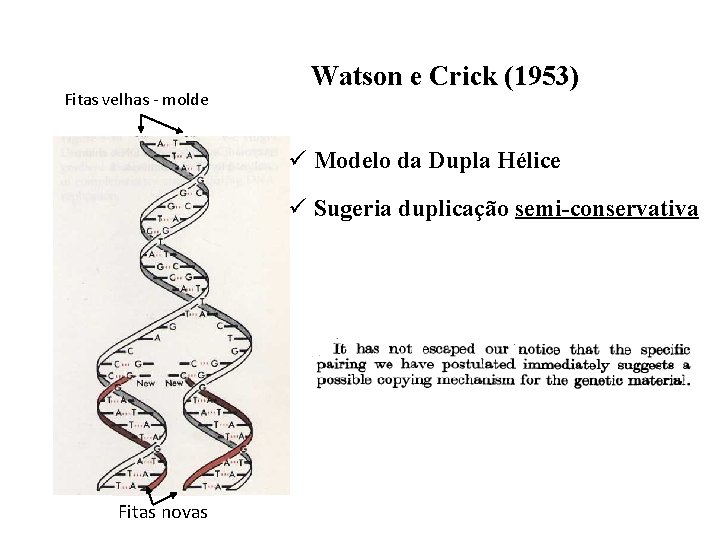
Fitas velhas - molde Watson e Crick (1953) ü Modelo da Dupla Hélice ü Sugeria duplicação semi-conservativa Fitas novas

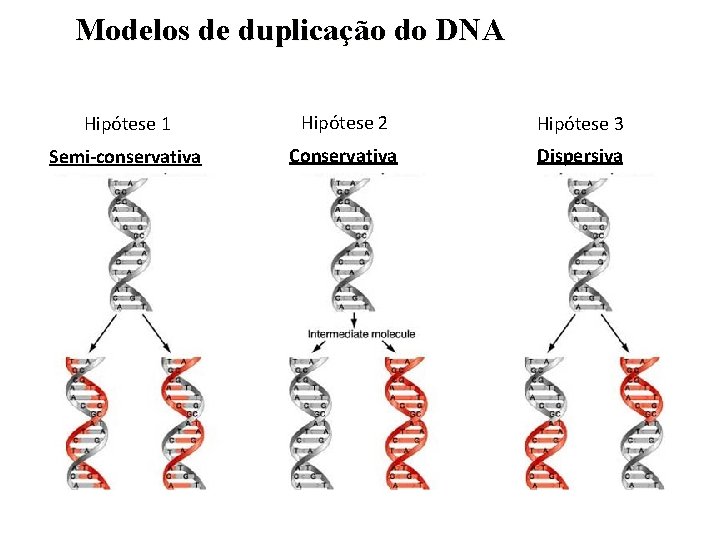
Modelos de duplicação do DNA Hipótese 1 Hipótese 2 Hipótese 3 Semi-conservativa Conservativa Dispersiva

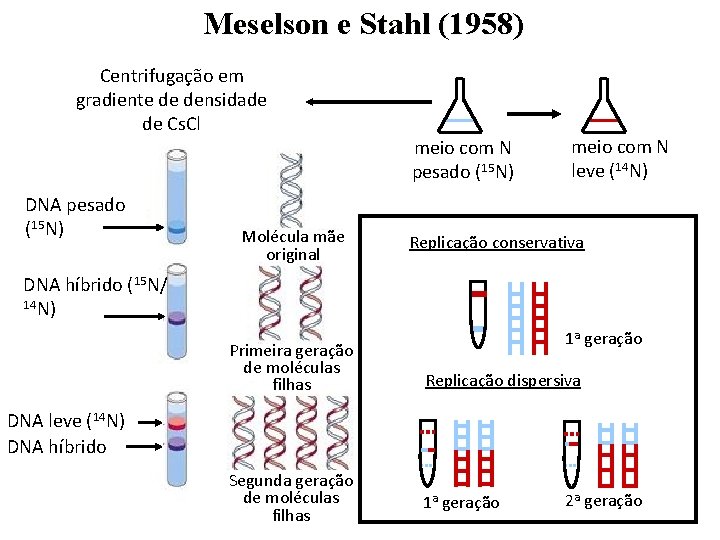
Meselson e Stahl (1958) Centrifugação em gradiente de densidade de Cs. Cl DNA pesado (15 N) Molécula mãe original meio com N pesado (15 N) meio com N leve (14 N) Replicação conservativa DNA híbrido (15 N/ 14 N) Primeira geração de moléculas filhas 1 a geração Replicação dispersiva DNA leve (14 N) DNA híbrido Segunda geração de moléculas filhas 1 a geração 2 a geração

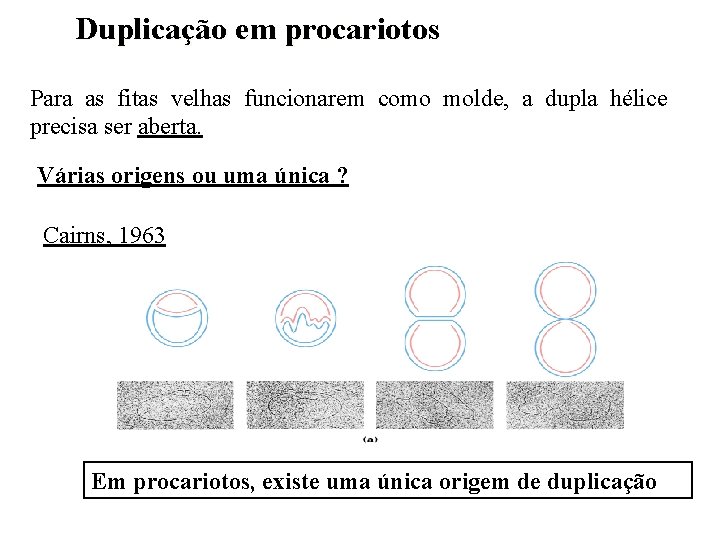
Duplicação em procariotos Para as fitas velhas funcionarem como molde, a dupla hélice precisa ser aberta. Várias origens ou uma única ? Cairns, 1963 Em procariotos, existe uma única origem de duplicação

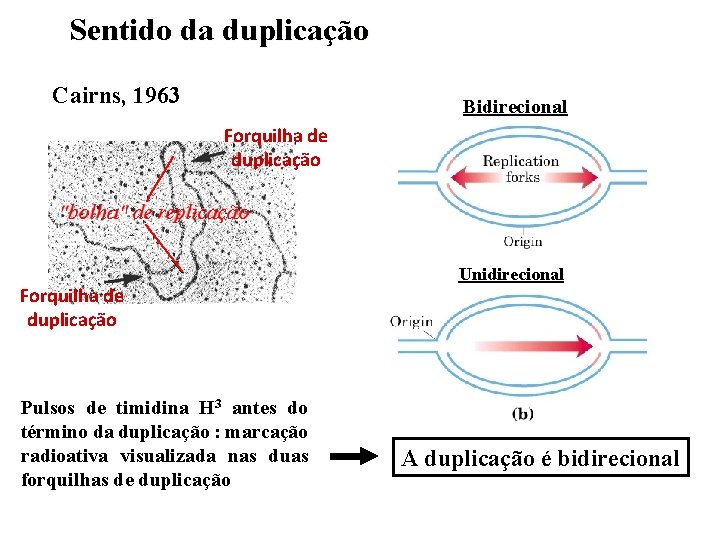
Sentido da duplicação Cairns, 1963 Bidirecional Forquilha de duplicação Pulsos de timidina H 3 antes do término da duplicação : marcação radioativa visualizada nas duas forquilhas de duplicação Unidirecional A duplicação é bidirecional

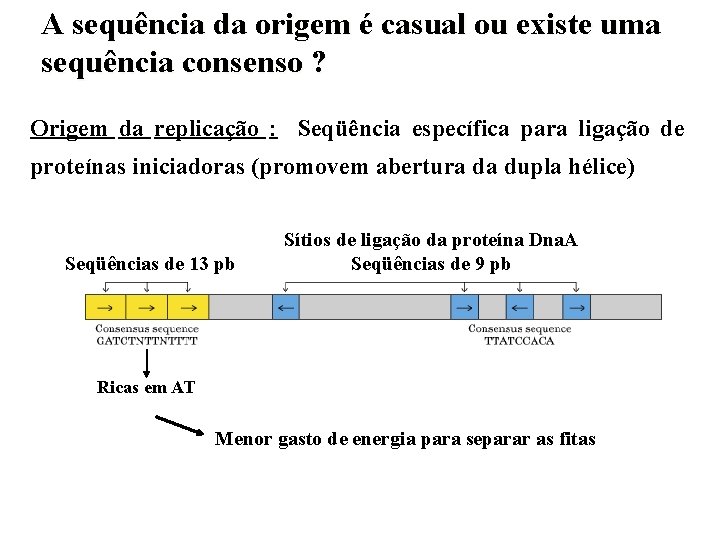
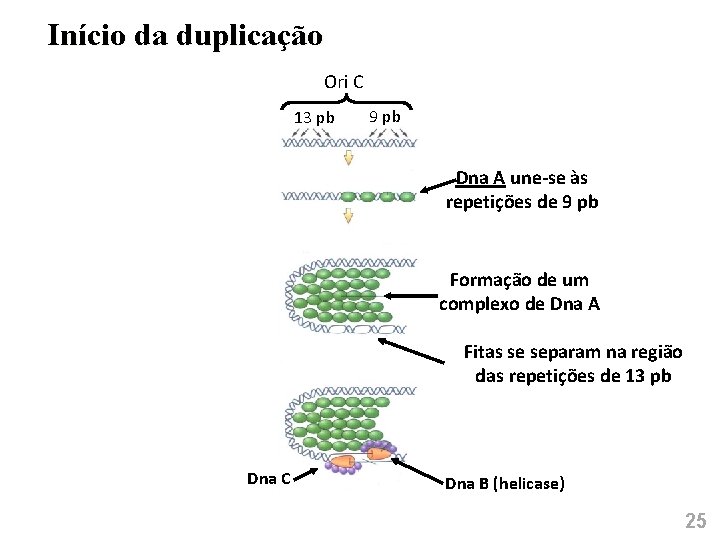
A sequência da origem é casual ou existe uma sequência consenso ? Origem da replicação : Seqüência específica para ligação de proteínas iniciadoras (promovem abertura da dupla hélice) Seqüências de 13 pb Sítios de ligação da proteína Dna. A Seqüências de 9 pb Ricas em AT Menor gasto de energia para separar as fitas

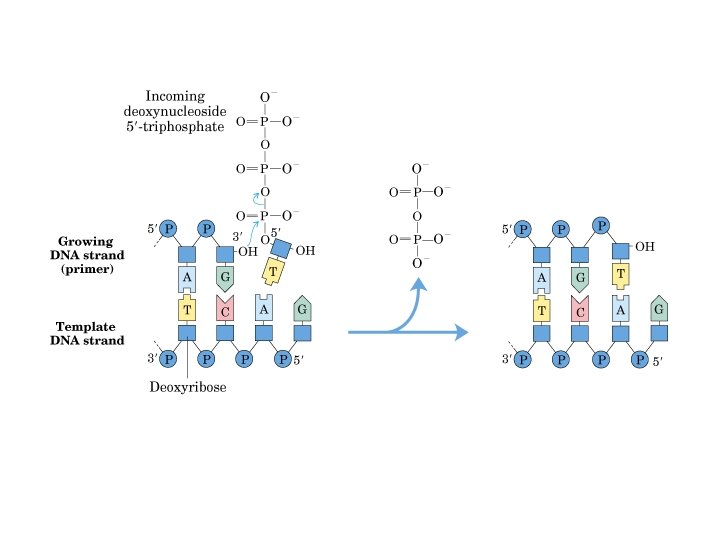
Uma vez que a dupla hélice está aberta, como é feita a síntese da nova fita ? DNA polimerases Kornberg (1957) : DNA polimerase I Requerimentos da DNA pol I para sintetizar nova fita de DNA : ü Desoxinucleotídeos trifosfatados ü Íons Mg 2+ ü DNA pré-existente, para servir como molde ü Uma extremidade OH-3’ de uma cadeia de DNA pré-existente para realizar a ligação fosfodiéster 10

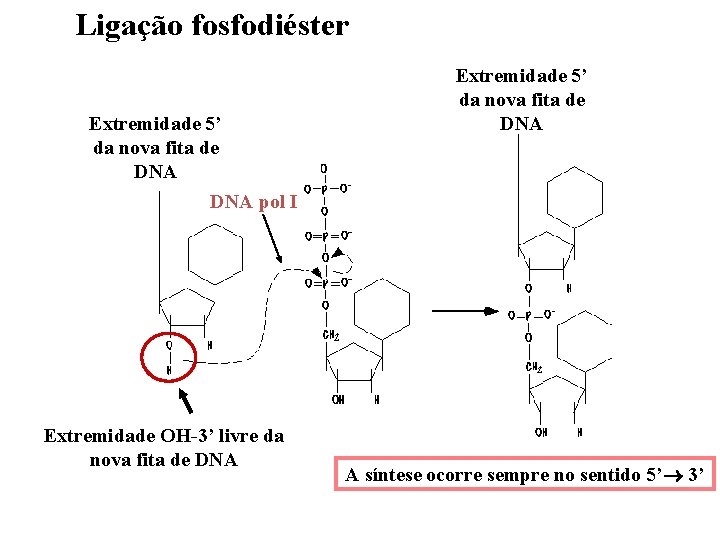
Ligação fosfodiéster Extremidade 5’ da nova fita de DNA pol I Extremidade OH-3’ livre da nova fita de DNA Extremidade 5’ da nova fita de DNA A síntese ocorre sempre no sentido 5’ 3’

O problema da iniciação DNA pol I não consegue iniciar a síntese da nova fita – ausência de extremidade OH-3’ livre. Primase : sintetiza oligonucleotídeo (primer) de RNA, fornecendo a extremidade OH-3’ livre para a DNA pol I Primer 5’ 3’ 3’ 5’


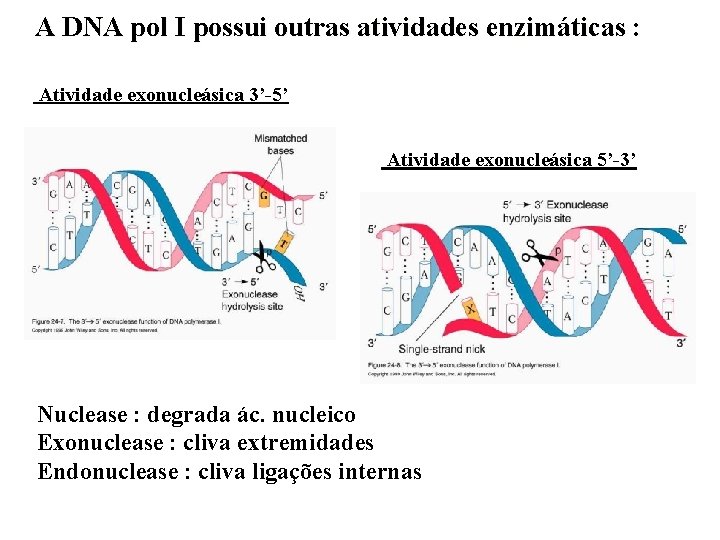
A DNA pol I possui outras atividades enzimáticas : Atividade exonucleásica 3’-5’ Atividade exonucleásica 5’-3’ Nuclease : degrada ác. nucleico Exonuclease : cliva extremidades Endonuclease : cliva ligações internas

Outras DNA polimerases foram descobertas : DNA pol II : • Reparo do DNA • Atividade polimerase 5’-3’ • Atividade exonuclease 3’ – 5’ DNA pol III : • Possui várias subunidades • Atividade polimerase 5’- 3’ • Atividade exonuclease 3’ – 5’ DNA polimerase IV e V – envolvidas em diferentes processos de reparo. 15

Processividade : habilidade da enzima de repetir a sua função catalítica sem dissociar-se do seu substrato Para a DNA pol : Definida pelo no médio de nt incorporados sem que a DNA pol se dissocie do DNA molde As DNA pol diferem em processividade

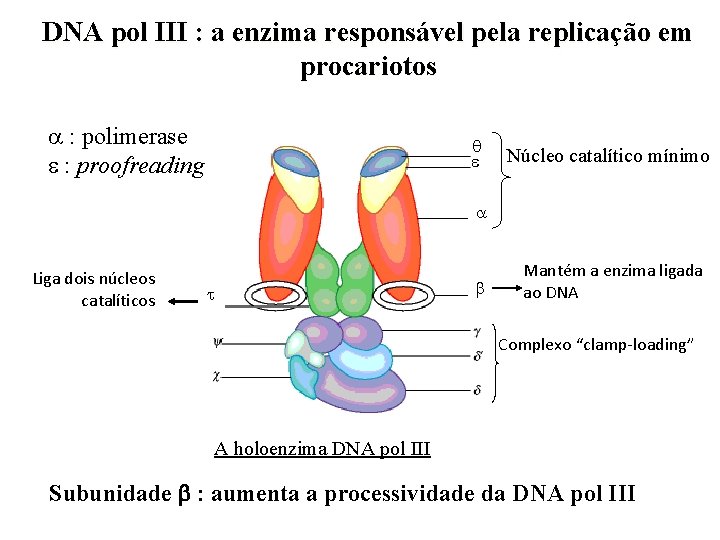
DNA pol III : a enzima responsável pela replicação em procariotos : polimerase : proofreading Núcleo catalítico mínimo Liga dois núcleos catalíticos Mantém a enzima ligada ao DNA Complexo “clamp-loading” A holoenzima DNA pol III Subunidade : aumenta a processividade da DNA pol III

Duplicação do DNA : um processo de alta fidelidade E. coli – 1 erro a ~ cada 109 bases adicionadas ou a após mil ou 10 mil eventos de replicação ü DNA polimerase tem um sítio ativo que só se fixa quando as bases estão pareadas corretamente ü DNA polimerase tem capacidade de verificar a fidelidade da replicação na cadeia nascente (proofreading) e corrigir os erros (atividades exonucleásicas) ü Depois da replicação, existe um sistema de reparo que corrige qualquer erro de pareamento de bases

Duplicação do DNA : um processo de alta fidelidade Pareamento incorreto : • Geometria diferente • Não acomoda-se no sítio ativo da DNA pol

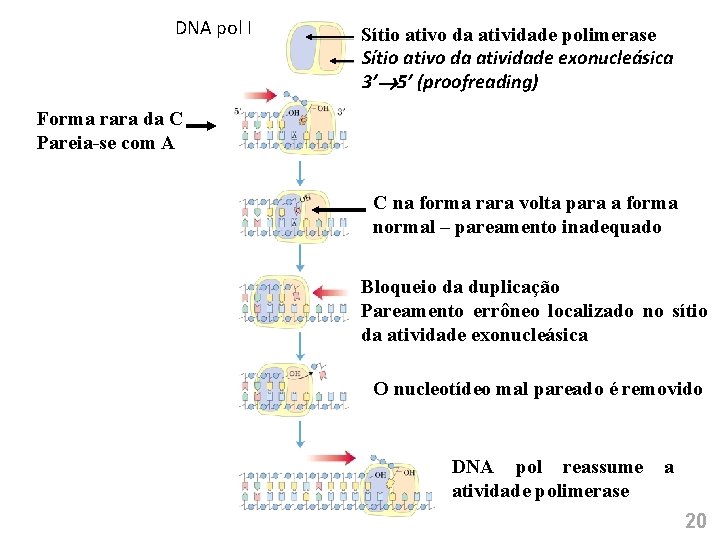
DNA pol I Sítio ativo da atividade polimerase Sítio ativo da atividade exonucleásica 3’ 5’ (proofreading) Forma rara da C Pareia-se com A C na forma rara volta para a forma normal – pareamento inadequado Bloqueio da duplicação Pareamento errôneo localizado no sítio da atividade exonucleásica O nucleotídeo mal pareado é removido DNA pol reassume atividade polimerase a 20

A síntese é contínua numa fita e descontínua na outra. . . 3’ 5’ 5’ 3’ 3’ 5’ Direção em que as fitas se estendem DNA pol só age no sentido 5’- 3’ !!! 3’ 5’ Direção em que as fitas se estendem 3’ 5’ Fita líder (contínua) 5’ 3’ 3’ 5’ Fita descontínua

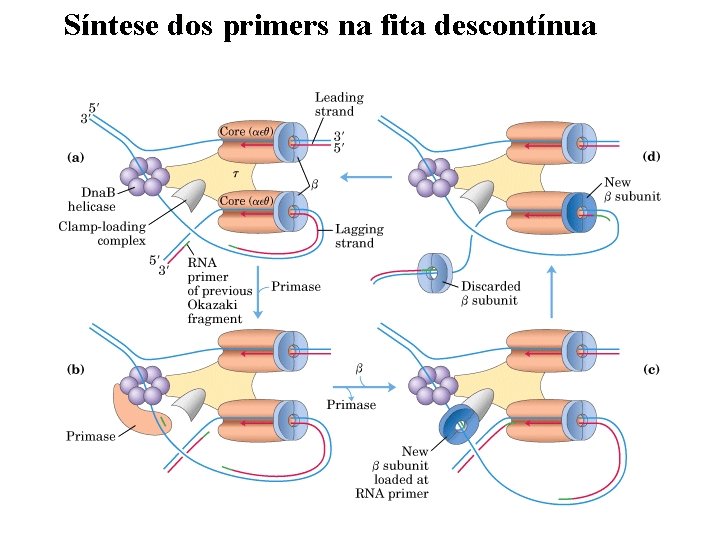
Síntese dos primers na fita descontínua

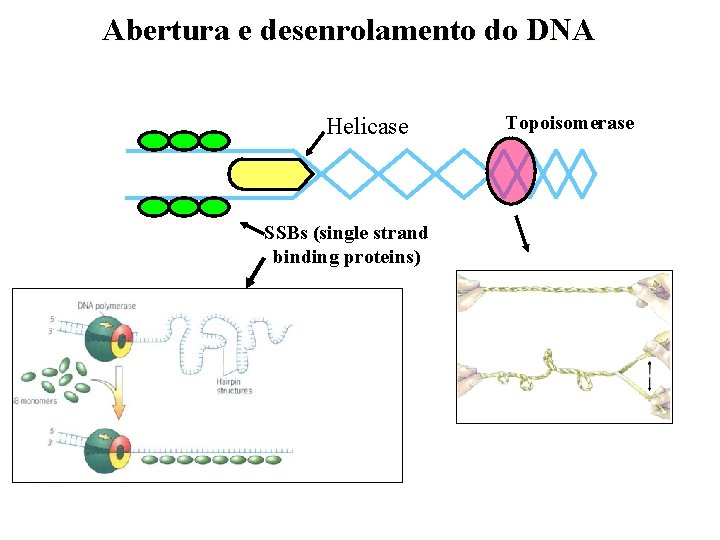
Abertura e desenrolamento do DNA Helicase SSBs (single strand binding proteins) Topoisomerase


Início da duplicação Ori C 13 pb 9 pb Dna A une-se às repetições de 9 pb Formação de um complexo de Dna A Fitas se separam na região das repetições de 13 pb Dna C Dna B (helicase) 25

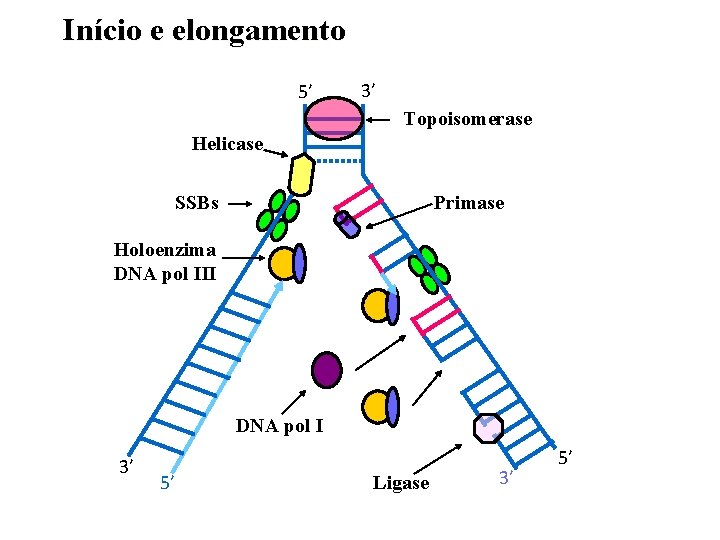
Início e elongamento 5’ 3’ Topoisomerase Helicase SSBs Primase Holoenzima DNA pol III DNA pol I 3’ 5’ Ligase 3’ 5’

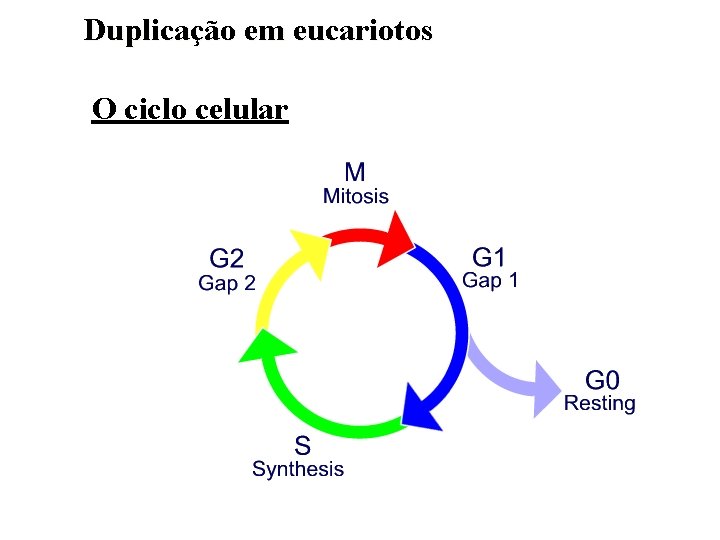
Duplicação em eucariotos O ciclo celular

Várias origens de duplicação Genoma humano – ~ 10 mil locais de origem (maior velocidade de replicação)

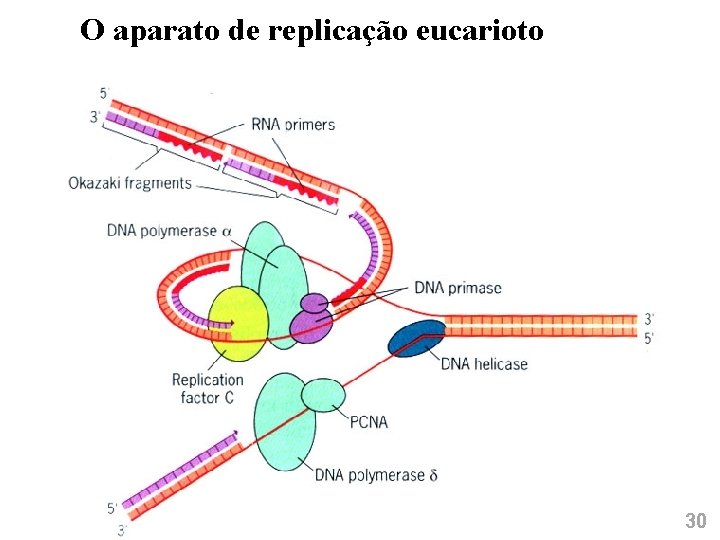
Tipos de DNA polimerases (Eucariotos) • POLIMERASE α - Núcleo - Atividade de polimerização e primase Fita descontínua - Sem atividade exonuclease 3’-5’ • POLIMERASE δ - Núcleo - Atividade de polimerização e exo 3’-5’ - Sem atividade primase • POLIMERASE ε - Núcleo - Atividade de exonuclease 3’-5’ (~ DNA Poli I) - Reparo de lesões provocadas por UV • RPA - Núcleo - Atividade semelhante à das SSB de procariotos • POLIMERASE γ - Mitocôndria - Replicação do genoma mitocondrial

O aparato de replicação eucarioto 30

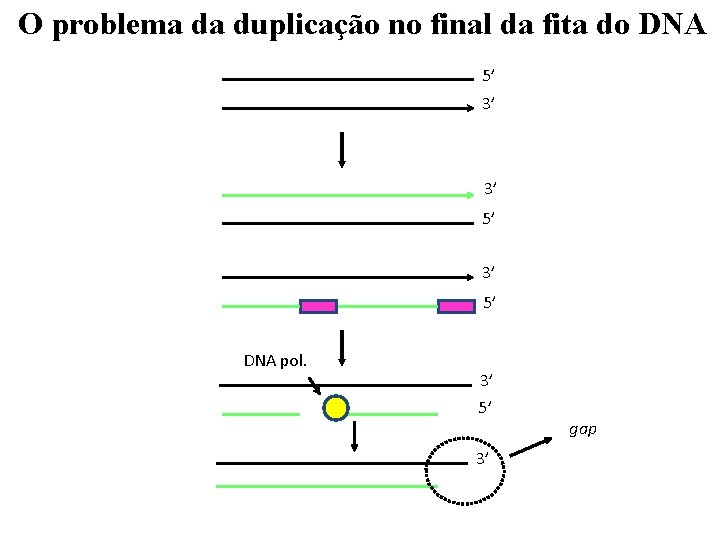
O problema da duplicação no final da fita do DNA 5’ 3’ 3’ 5’ DNA pol. 3’ 5’ 3’ gap

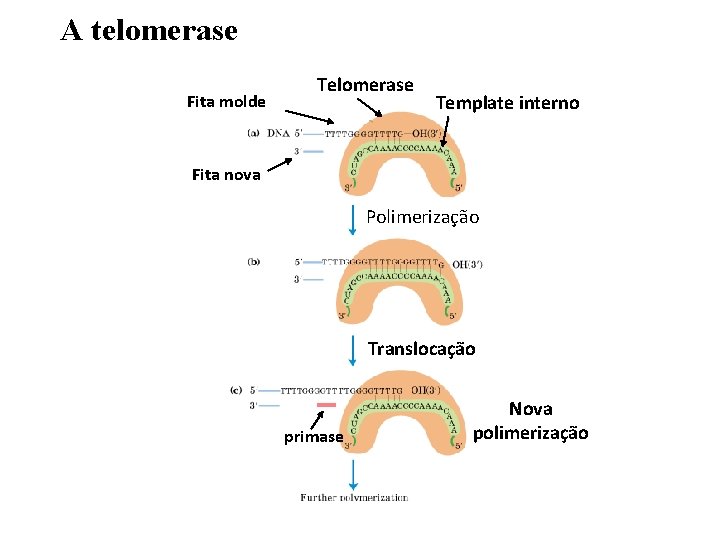
A telomerase Fita molde Telomerase Template interno Fita nova Polimerização Translocação primase Nova polimerização

Centro de Ciências Exatas e da Natureza Departamento de Biologia Molecular Curso: Ciências Biológicas Disciplina: Genética Molecular Replicação do DNA Bons Estudos Eleonidas Moura Lima
 Centro zonal norte centro historico
Centro zonal norte centro historico Departamento ceres
Departamento ceres Projeto doutorado
Projeto doutorado Departamento de economia
Departamento de economia Organigrama de room service
Organigrama de room service Prepa abierta puebla plan modular
Prepa abierta puebla plan modular La señora guajardo compra un departamento
La señora guajardo compra un departamento Organización del departamento de enfermería
Organización del departamento de enfermería El significado de departamento
El significado de departamento Objetivos del departamento de enfermería
Objetivos del departamento de enfermería Potencialidades productivas del departamento de cochabamba
Potencialidades productivas del departamento de cochabamba Organigrama de departamento de contabilidad
Organigrama de departamento de contabilidad Departamento ciencias naturales ies suel
Departamento ciencias naturales ies suel Finestra terapeutica
Finestra terapeutica Departamento solicitante
Departamento solicitante Departamento del valle del cauca
Departamento del valle del cauca Departamento forestal
Departamento forestal Zootecnia ufpr
Zootecnia ufpr Departamento ceres
Departamento ceres Departamentos en una empresa
Departamentos en una empresa Que es atributo multivaluado
Que es atributo multivaluado Organigrama departamento de marketing
Organigrama departamento de marketing Universidad ixil
Universidad ixil Departamento de higiene de los alimentos
Departamento de higiene de los alimentos Estructura de una agencia publicitaria
Estructura de una agencia publicitaria Natureza cordel
Natureza cordel Instrumentos de navegação
Instrumentos de navegação Um pedacinho de terra perdido no mar
Um pedacinho de terra perdido no mar A filosofia grega parece começar com uma ideia absurda
A filosofia grega parece começar com uma ideia absurda Descarto-me da tronga
Descarto-me da tronga Replicação do dna
Replicação do dna Atividade de paisagem natural e modificada
Atividade de paisagem natural e modificada Verbo de ação
Verbo de ação John locke estado natural
John locke estado natural

























































